catELMo
1.0.0
Catelmo是一种双向氨基酸嵌入模型,它学习上下文化的氨基酸表示,将氨基酸视为单词和序列作为句子。它通过预测每个氨基酸令牌的图案来学习氨基酸序列的模式,鉴于其先前的令牌。它已经接受了4,173,895 TCR的培训
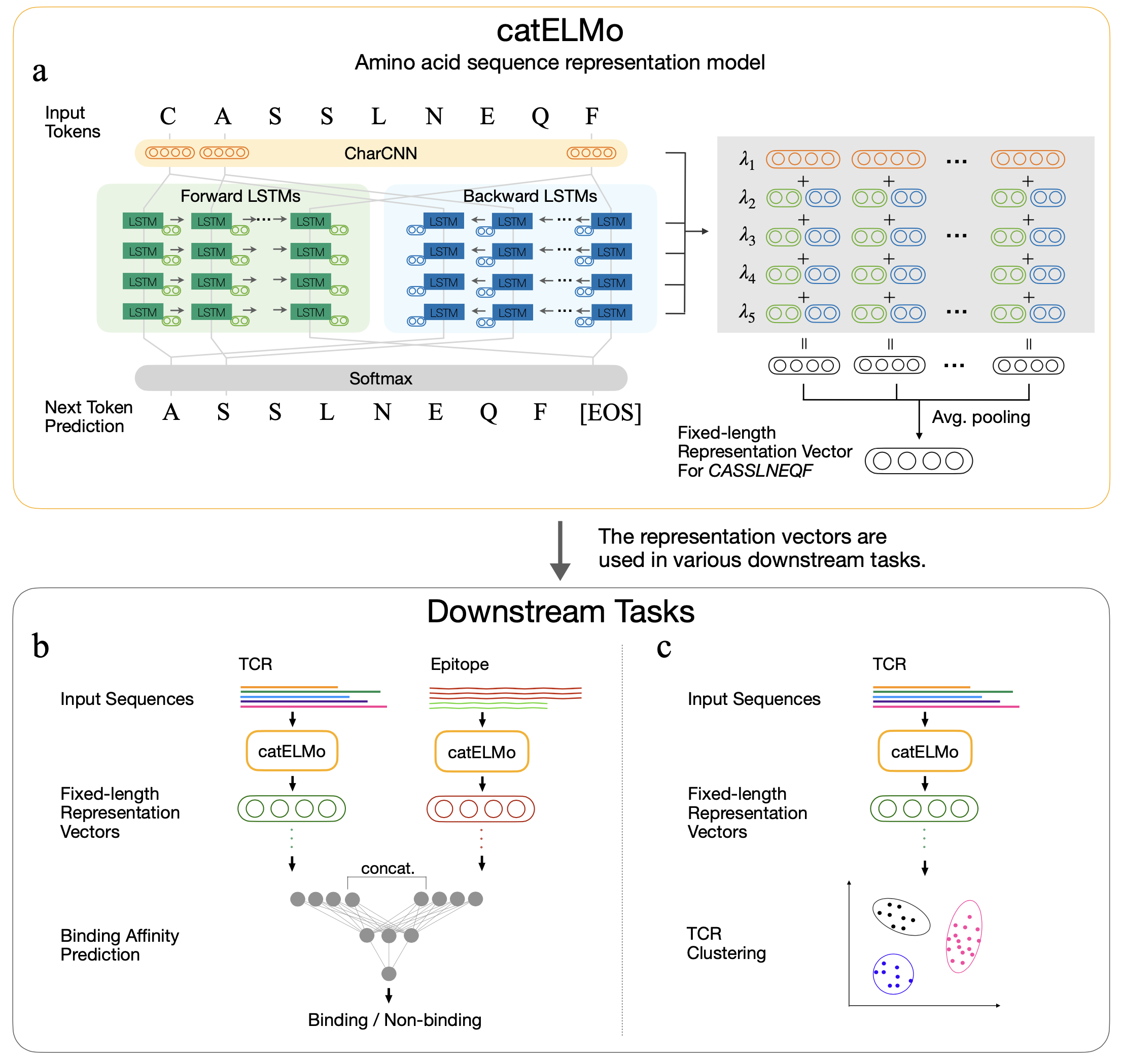
上下文感知的氨基酸嵌入进展分析TCR-质相互作用
Pengfei Zhang 1,2 ,Michael Cai 1,2 ,Seojin Bang 2 ,Heewook Lee 1,2
1亚利桑那州立大学的计算与增强情报学院,亚利桑那州立大学生物设计学院2
发表于: Elife,2023年。
纸|代码|海报|幻灯片|演示文稿(YouTube)
git clone https://github.com/Lee-CBG/catELMo
cd catELMo/
conda create --name bap python=3.6.13
pip install pandas==1.1.5 tensorflow==2.6.0 keras==2.6.0 scikit-learn==0.24.2 tqdm
source activate bapdatasets集文件夹下载培训和测试数据。embedders文件夹的说明,获取TCR和表位的嵌入。表位分裂的一个例子
python -W ignore bap.py
--embedding catELMo_4_layers_1024
--split epitope
--gpu 0
--fraction 1
--seed 42如果您使用此代码或使用我们的Catelmo进行研究,请引用我们的论文:
@article {catelmobiorxiv,
author = {Pengfei Zhang and Seojin Bang and Michael Cai and Heewook Lee},
title = {Context-Aware Amino Acid Embedding Advances Analysis of TCR-Epitope Interactions},
elocation-id = {2023.04.12.536635},
year = {2023},
doi = {10.1101/2023.04.12.536635},
publisher = {Cold Spring Harbor Laboratory},
journal = {bioRxiv}
}
这项工作是在创意共享归因非商业 - 诺迪毒素4.0国际许可下获得许可的。