Catelmo es un modelo de incrustación de aminoácidos bidireccionales que aprende representaciones de aminoácidos contextualizadas, tratando un aminoácido como una palabra y una secuencia como oración. Aprende patrones de secuencias de aminoácidos con su señal de auto-supervisión, al predecir cada uno el siguiente token de aminoácidos dados sus tokens anteriores. Ha sido entrenado en 4,173.895 TCR
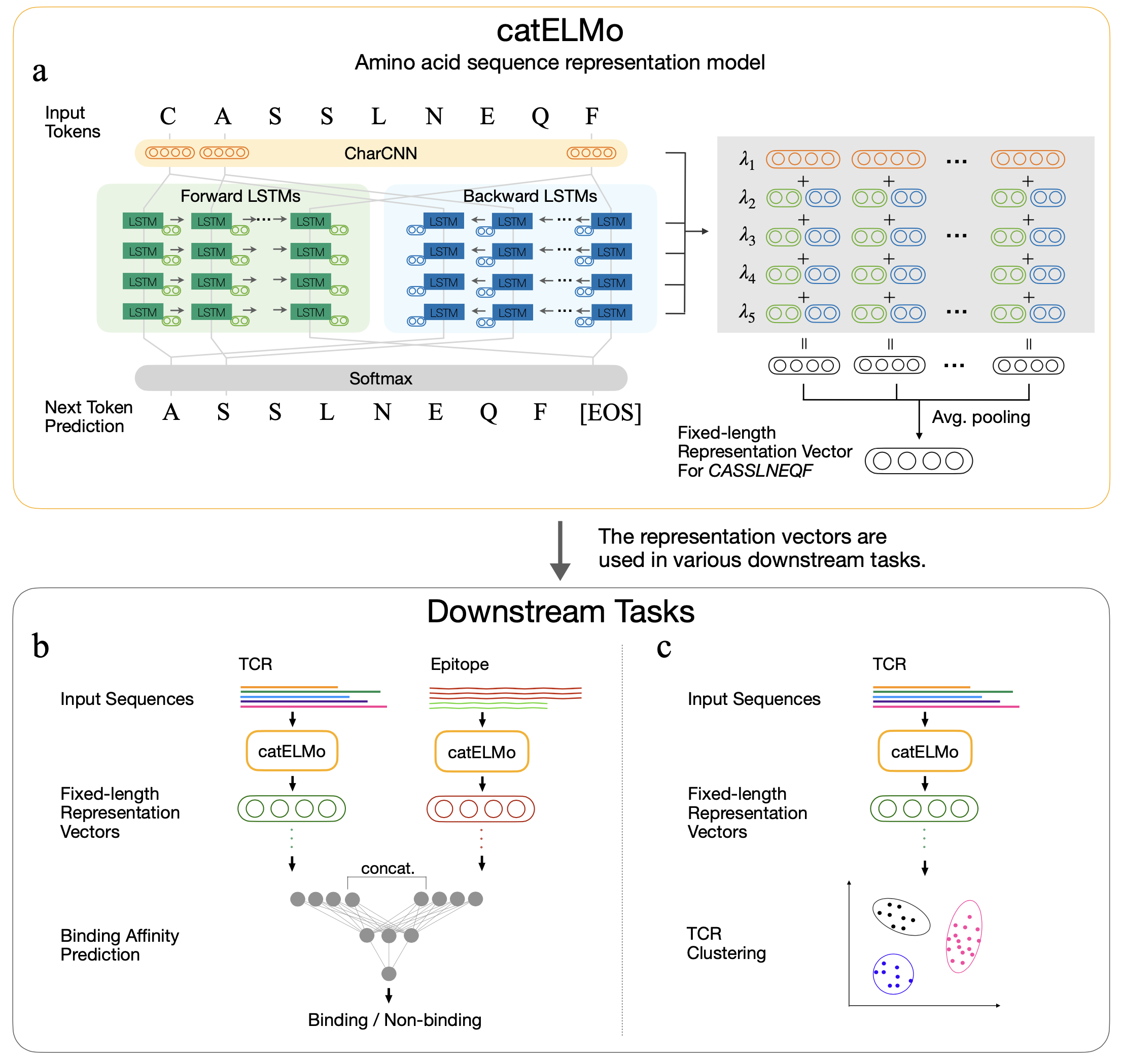
El análisis de los aminoácidos conscientes del contexto avanza el análisis de las interacciones TCR-Epitope
Pengfei Zhang 1,2 , Michael Cai 1,2 , Seojin Bang 2 , Heewook Lee 1,2
1 Escuela de Computación e Inteligencia Aumentada, Universidad Estatal de Arizona, 2 BioDesign Institute, Arizona State University
Publicado en: Elife, 2023.
Papel | Código | Cartel | Diapositivas | Presentación (YouTube)
git clone https://github.com/Lee-CBG/catELMo
cd catELMo/
conda create --name bap python=3.6.13
pip install pandas==1.1.5 tensorflow==2.6.0 keras==2.6.0 scikit-learn==0.24.2 tqdm
source activate bapdatasets .embedders .Un ejemplo de división de epítopos
python -W ignore bap.py
--embedding catELMo_4_layers_1024
--split epitope
--gpu 0
--fraction 1
--seed 42Si usa este código o usa nuestro Catelmo para su investigación, cite nuestro documento:
@article {catelmobiorxiv,
author = {Pengfei Zhang and Seojin Bang and Michael Cai and Heewook Lee},
title = {Context-Aware Amino Acid Embedding Advances Analysis of TCR-Epitope Interactions},
elocation-id = {2023.04.12.536635},
year = {2023},
doi = {10.1101/2023.04.12.536635},
publisher = {Cold Spring Harbor Laboratory},
journal = {bioRxiv}
}
Este trabajo tiene licencia bajo una licencia internacional de Atribución de Commons-Commons-Noderivatives 4.0.