Catelmo هو نموذج تضمين الأحماض الأمينية ثنائية الاتجاه يتعلم تمثيلات الأحماض الأمينية السياقية ، ويتعامل مع الأحماض الأمينية ككلمة وتسلسل كجملة. إنه يتعلم أنماط تسلسل الأحماض الأمينية مع إشارة الإشراف الذاتي ، من خلال التنبؤ بكل رمز الأحماض الأمينية التالية بالنظر إلى الرموز السابقة. تم تدريبه على 4،173،895 TCR
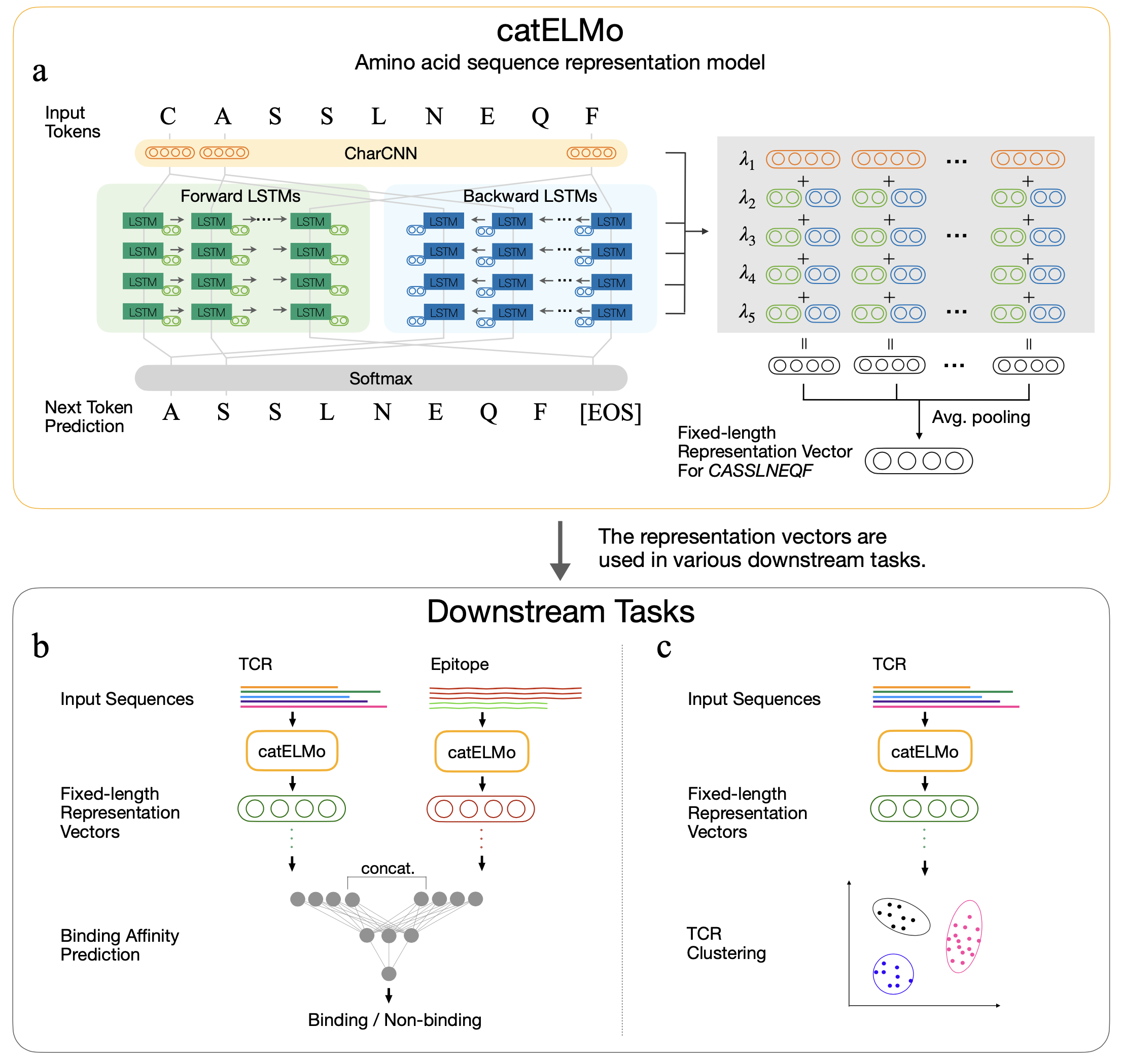
السياق-يدرك الأحماض الأمينية تحليل تحليل تفاعلات TCR-Epitope
Pengfei Zhang 1،2 ، Michael Cai 1،2 ، Seojin Bang 2 ، Heewook Lee 1،2
مدرسة واحدة للحوسبة والاستخبارات المعززة ، جامعة ولاية أريزونا ، 2 معهد التصميم الحيوي ، جامعة ولاية أريزونا
نشرت في: Elife ، 2023.
ورقة | رمز | ملصق | الشرائح | عرض تقديمي (يوتيوب)
git clone https://github.com/Lee-CBG/catELMo
cd catELMo/
conda create --name bap python=3.6.13
pip install pandas==1.1.5 tensorflow==2.6.0 keras==2.6.0 scikit-learn==0.24.2 tqdm
source activate bapdatasets .embedders .مثال لتقسيم epitope
python -W ignore bap.py
--embedding catELMo_4_layers_1024
--split epitope
--gpu 0
--fraction 1
--seed 42إذا كنت تستخدم هذا الرمز أو استخدمت catelmo لدينا لبحثك ، فيرجى الاستشهاد بورقة:
@article {catelmobiorxiv,
author = {Pengfei Zhang and Seojin Bang and Michael Cai and Heewook Lee},
title = {Context-Aware Amino Acid Embedding Advances Analysis of TCR-Epitope Interactions},
elocation-id = {2023.04.12.536635},
year = {2023},
doi = {10.1101/2023.04.12.536635},
publisher = {Cold Spring Harbor Laboratory},
journal = {bioRxiv}
}
تم ترخيص هذا العمل بموجب ترخيص Creative Commons Noncommercial-Noderivivative 4.0 الدولي.