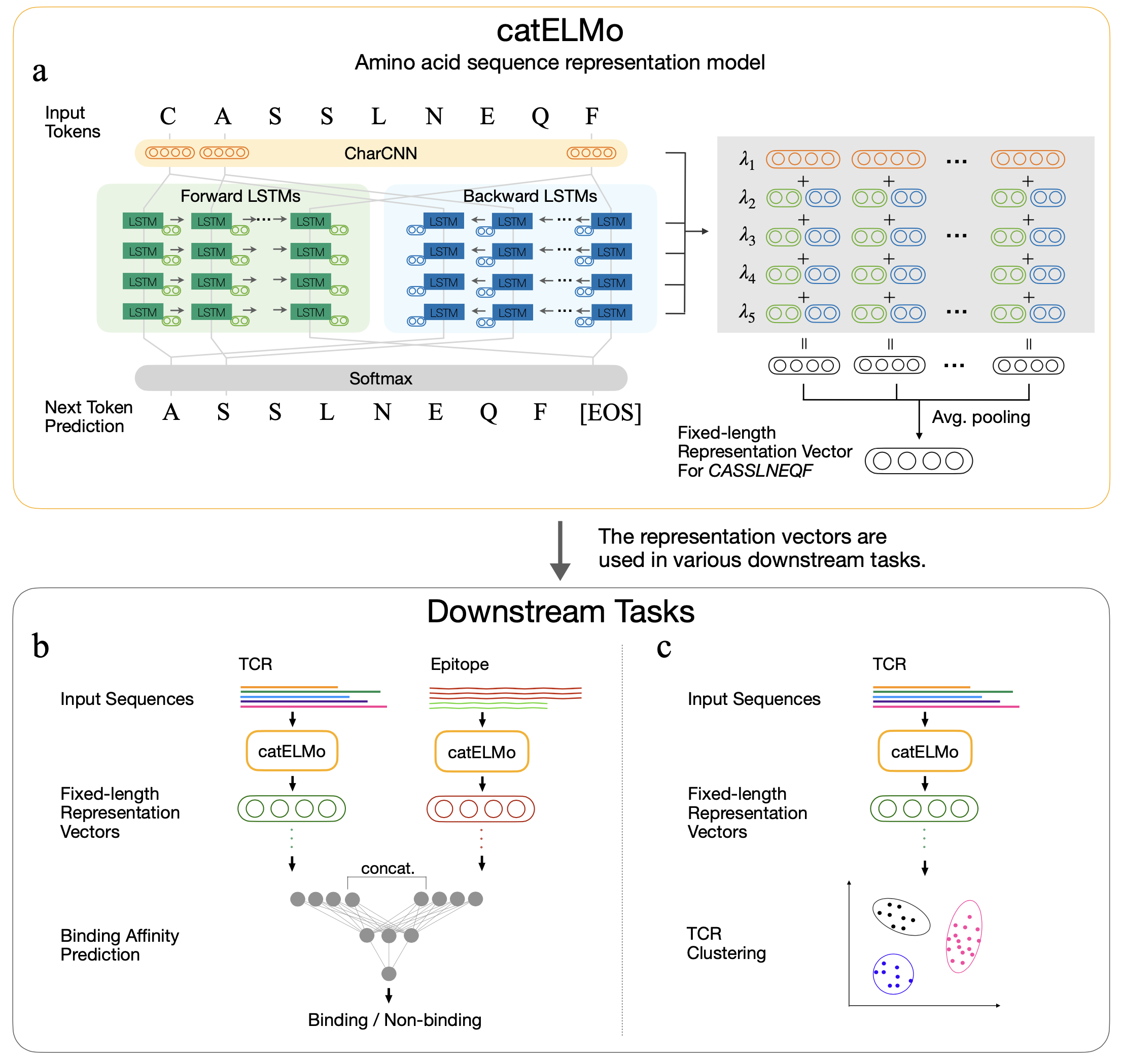
Catelmo adalah model embedding asam amino dua arah yang mempelajari representasi asam amino kontekstual, memperlakukan asam amino sebagai kata dan urutan sebagai kalimat. Ia mempelajari pola urutan asam amino dengan sinyal persiapan sendiri, dengan memprediksi masing-masing token asam amino berikutnya yang diberikan token sebelumnya. Telah dilatih pada 4.173.895 TCR
Konteks-Aware Amino Acid Embedding Advances Analisis Interaksi TCR-Epitope
Pengfei Zhang 1,2 , Michael Cai 1,2 , Seojin Bang 2 , Heewook Lee 1,2
1 Sekolah Komputasi dan Augmented Intelligence, Arizona State University, 2 Biodesign Institute, Arizona State University
Diterbitkan di: Elife, 2023.
Kertas | Kode | Poster | Slide | Presentasi (YouTube)
git clone https://github.com/Lee-CBG/catELMo
cd catELMo/
conda create --name bap python=3.6.13
pip install pandas==1.1.5 tensorflow==2.6.0 keras==2.6.0 scikit-learn==0.24.2 tqdm
source activate bapdatasets .embedders .Contoh untuk split epitope
python -W ignore bap.py
--embedding catELMo_4_layers_1024
--split epitope
--gpu 0
--fraction 1
--seed 42Jika Anda menggunakan kode ini atau menggunakan catelmo kami untuk penelitian Anda, silakan kutip makalah kami:
@article {catelmobiorxiv,
author = {Pengfei Zhang and Seojin Bang and Michael Cai and Heewook Lee},
title = {Context-Aware Amino Acid Embedding Advances Analysis of TCR-Epitope Interactions},
elocation-id = {2023.04.12.536635},
year = {2023},
doi = {10.1101/2023.04.12.536635},
publisher = {Cold Spring Harbor Laboratory},
journal = {bioRxiv}
}
Karya ini dilisensikan di bawah lisensi internasional Atribusi-Nonkomersial-Noderivatif 4.0 Creative Commons.