O Catelmo é um modelo de incorporação de aminoácidos bidirecionais que aprende representações contextualizadas de aminoácidos, tratando um aminoácido como uma palavra e uma sequência como frase. Aprende padrões de seqüências de aminoácidos com seu sinal de auto-supervisão, prevendo cada um do próximo token de aminoácidos, dados seus tokens anteriores. Foi treinado em 4.173.895 TCR
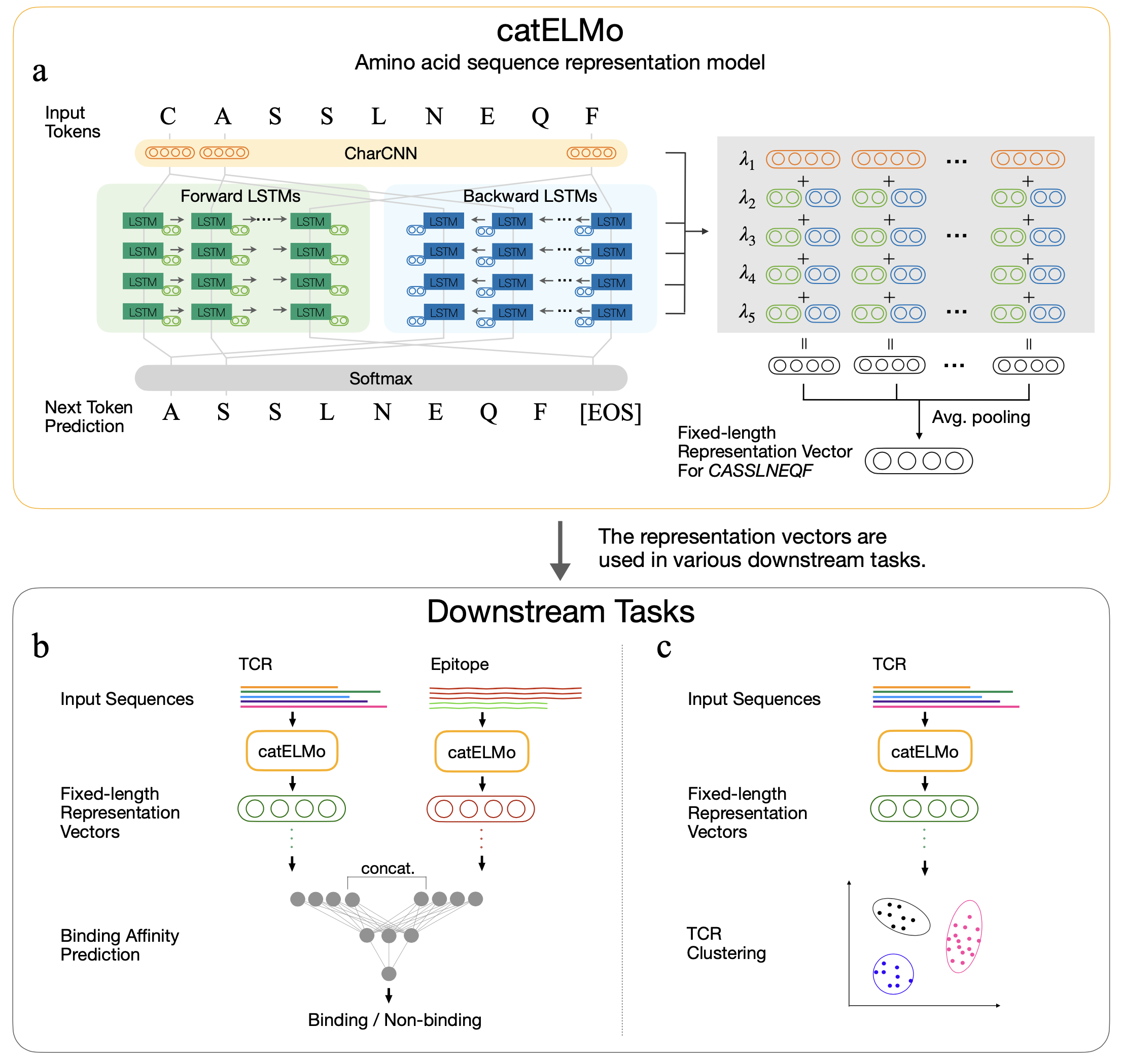
Aminoácidos com reconhecimento de contexto Analisar a análise das interações do epítopo TCR-Epitopo
Pengfei Zhang 1,2 , Michael Cai 1,2 , Seojin Bang 2 , Heewook Lee 1,2
1 Escola de Computação e Inteligência Aumentada, Arizona State University, 2 Biodesign Institute, Arizona State University
Publicado em: ELIFE, 2023.
Papel | Código | Pôster | Slides | Apresentação (YouTube)
git clone https://github.com/Lee-CBG/catELMo
cd catELMo/
conda create --name bap python=3.6.13
pip install pandas==1.1.5 tensorflow==2.6.0 keras==2.6.0 scikit-learn==0.24.2 tqdm
source activate bapdatasets .embedders .Um exemplo para divisão de epítopo
python -W ignore bap.py
--embedding catELMo_4_layers_1024
--split epitope
--gpu 0
--fraction 1
--seed 42Se você usar este código ou usar nosso Catelmo para sua pesquisa, cite nosso artigo:
@article {catelmobiorxiv,
author = {Pengfei Zhang and Seojin Bang and Michael Cai and Heewook Lee},
title = {Context-Aware Amino Acid Embedding Advances Analysis of TCR-Epitope Interactions},
elocation-id = {2023.04.12.536635},
year = {2023},
doi = {10.1101/2023.04.12.536635},
publisher = {Cold Spring Harbor Laboratory},
journal = {bioRxiv}
}
Este trabalho é licenciado sob uma Licença Internacional Creative Commons Attribution-NonCommercial-Noderivatives 4.0 International.