Catelmo เป็นรูปแบบการฝังกรดอะมิโนแบบสองทิศทางที่เรียนรู้การเป็นตัวแทนกรดอะมิโนตามบริบทการรักษากรดอะมิโนเป็นคำและลำดับเป็นประโยค มันเรียนรู้รูปแบบของลำดับกรดอะมิโนที่มีสัญญาณการก่อการร้ายตนเองโดยทำนายโทเค็นกรดอะมิโนต่อไปแต่ละตัวตามโทเค็นก่อนหน้า ได้รับการฝึกอบรมใน 4,173,895 TCR
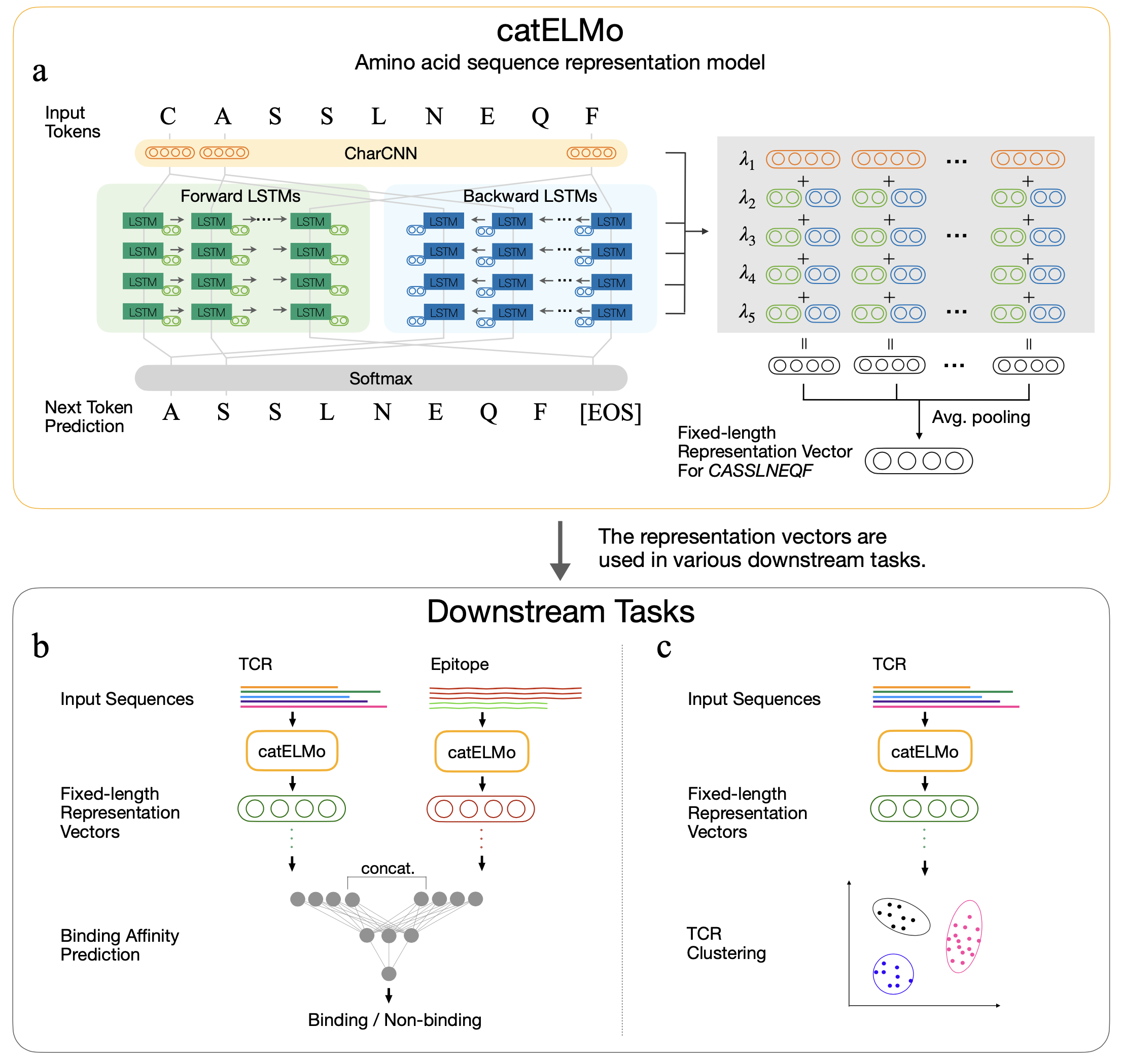
การฝังกรดอะมิโนที่รับรู้บริบทการวิเคราะห์ความก้าวหน้าของการปฏิสัมพันธ์ระหว่าง TCR-epitope
Pengfei Zhang 1,2 , Michael Cai 1,2 , Seojin Bang 2 , Heewook Lee 1,2
1 School of Computing and Augmented Intelligence, Arizona State University, 2 Biodesign Institute, Arizona State University
เผยแพร่ใน: Elife, 2023
กระดาษ | รหัส | โปสเตอร์ สไลด์ การนำเสนอ (YouTube)
git clone https://github.com/Lee-CBG/catELMo
cd catELMo/
conda create --name bap python=3.6.13
pip install pandas==1.1.5 tensorflow==2.6.0 keras==2.6.0 scikit-learn==0.24.2 tqdm
source activate bapdatasetsembeddersตัวอย่างสำหรับการแยก epitope
python -W ignore bap.py
--embedding catELMo_4_layers_1024
--split epitope
--gpu 0
--fraction 1
--seed 42หากคุณใช้รหัสนี้หรือใช้ catelmo ของเราสำหรับการวิจัยของคุณโปรดอ้างอิงบทความของเรา:
@article {catelmobiorxiv,
author = {Pengfei Zhang and Seojin Bang and Michael Cai and Heewook Lee},
title = {Context-Aware Amino Acid Embedding Advances Analysis of TCR-Epitope Interactions},
elocation-id = {2023.04.12.536635},
year = {2023},
doi = {10.1101/2023.04.12.536635},
publisher = {Cold Spring Harbor Laboratory},
journal = {bioRxiv}
}
งานนี้ได้รับใบอนุญาตภายใต้ Creative Commons Attribution-Noncommercial-Noderivatives 4.0 ใบอนุญาตระหว่างประเทศ