catELMo
1.0.0
Catelmoは、文脈化されたアミノ酸表現を学習し、アミノ酸を単語として扱い、文としてシーケンスとして扱う双方向アミノ酸埋め込みモデルです。前のトークンを考慮して、次のアミノ酸トークンをそれぞれ予測することにより、自己補給信号を使用してアミノ酸配列のパターンを学習します。 4,173,895 TCRで訓練されています
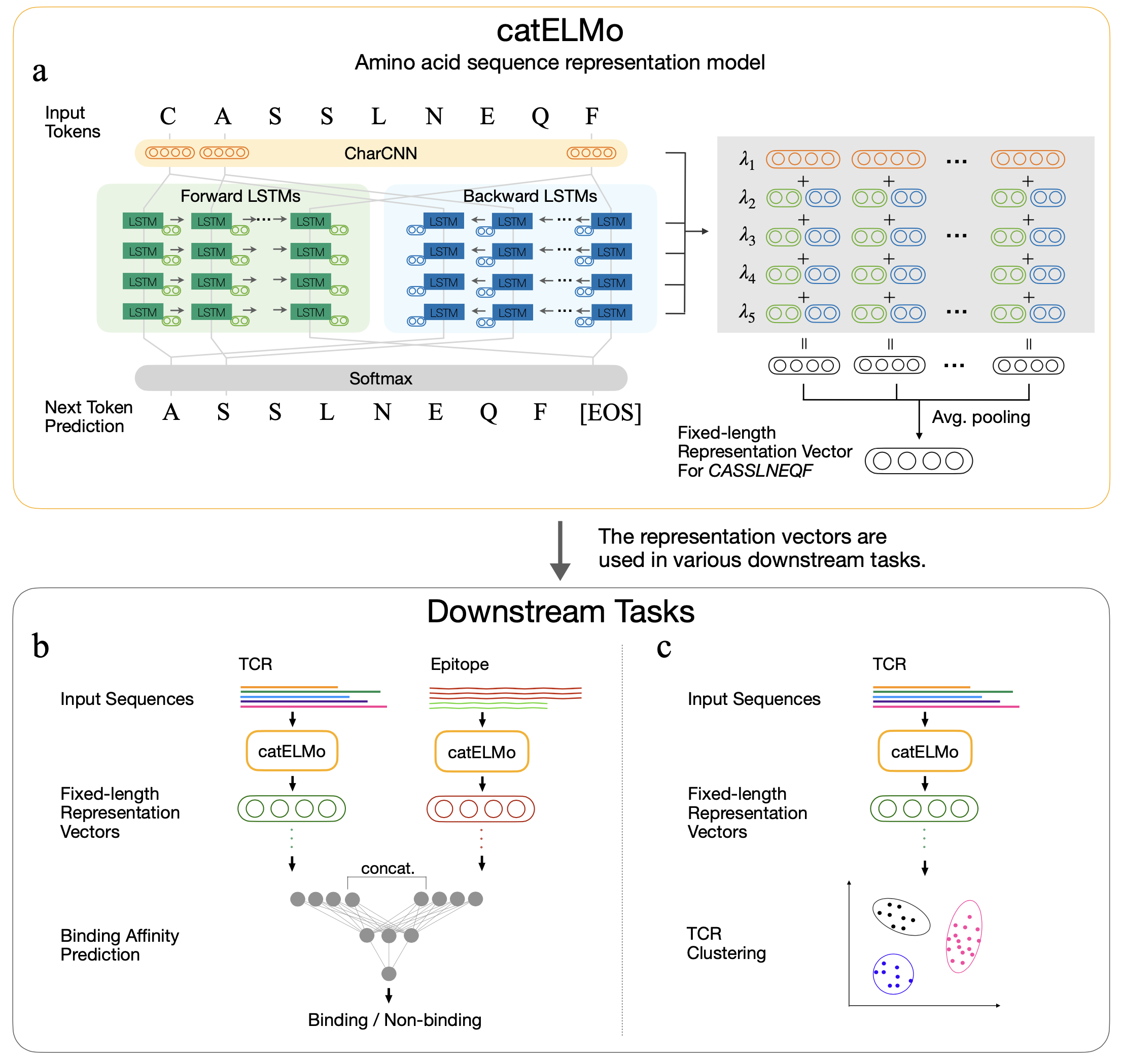
コンテキスト認識アミノ酸埋め込みTCRエピトープ相互作用の進歩分析
Pengfei Zhang 1,2 、Michael Cai 1,2 、Seojin Bang 2 、Heewook Lee 1,2
1アリゾナ州立大学のアリゾナ州立大学のコンピューティングと拡張インテリジェンスの学校
公開: Elife、2023。
論文|コード|ポスター|スライド|プレゼンテーション(YouTube)
git clone https://github.com/Lee-CBG/catELMo
cd catELMo/
conda create --name bap python=3.6.13
pip install pandas==1.1.5 tensorflow==2.6.0 keras==2.6.0 scikit-learn==0.24.2 tqdm
source activate bapdatasetsフォルダーからトレーニングとテストデータをダウンロードします。embeddersフォルダーから指示に従って、TCRおよびエピトープの埋め込みを取得します。エピトープ分割の例
python -W ignore bap.py
--embedding catELMo_4_layers_1024
--split epitope
--gpu 0
--fraction 1
--seed 42このコードを使用するか、Catelmoを使用して調査する場合は、私たちの論文を引用してください。
@article {catelmobiorxiv,
author = {Pengfei Zhang and Seojin Bang and Michael Cai and Heewook Lee},
title = {Context-Aware Amino Acid Embedding Advances Analysis of TCR-Epitope Interactions},
elocation-id = {2023.04.12.536635},
year = {2023},
doi = {10.1101/2023.04.12.536635},
publisher = {Cold Spring Harbor Laboratory},
journal = {bioRxiv}
}
この作業は、Creative Commons Attribution-NonCommercial-Noderivatives 4.0国際ライセンスの下でライセンスされています。