Catelmo est un modèle d'intégration d'acides aminés bidirectionnelle qui apprend des représentations contextualisées d'acides aminés, traitant un acide aminé comme un mot et une séquence comme une phrase. Il apprend des schémas de séquences d'acides aminés avec son signal d'auto-opération, en prédisant chacun du jeton d'acides aminés suivant compte tenu de ses jetons précédents. Il a été formé sur 4 173 895 TCR
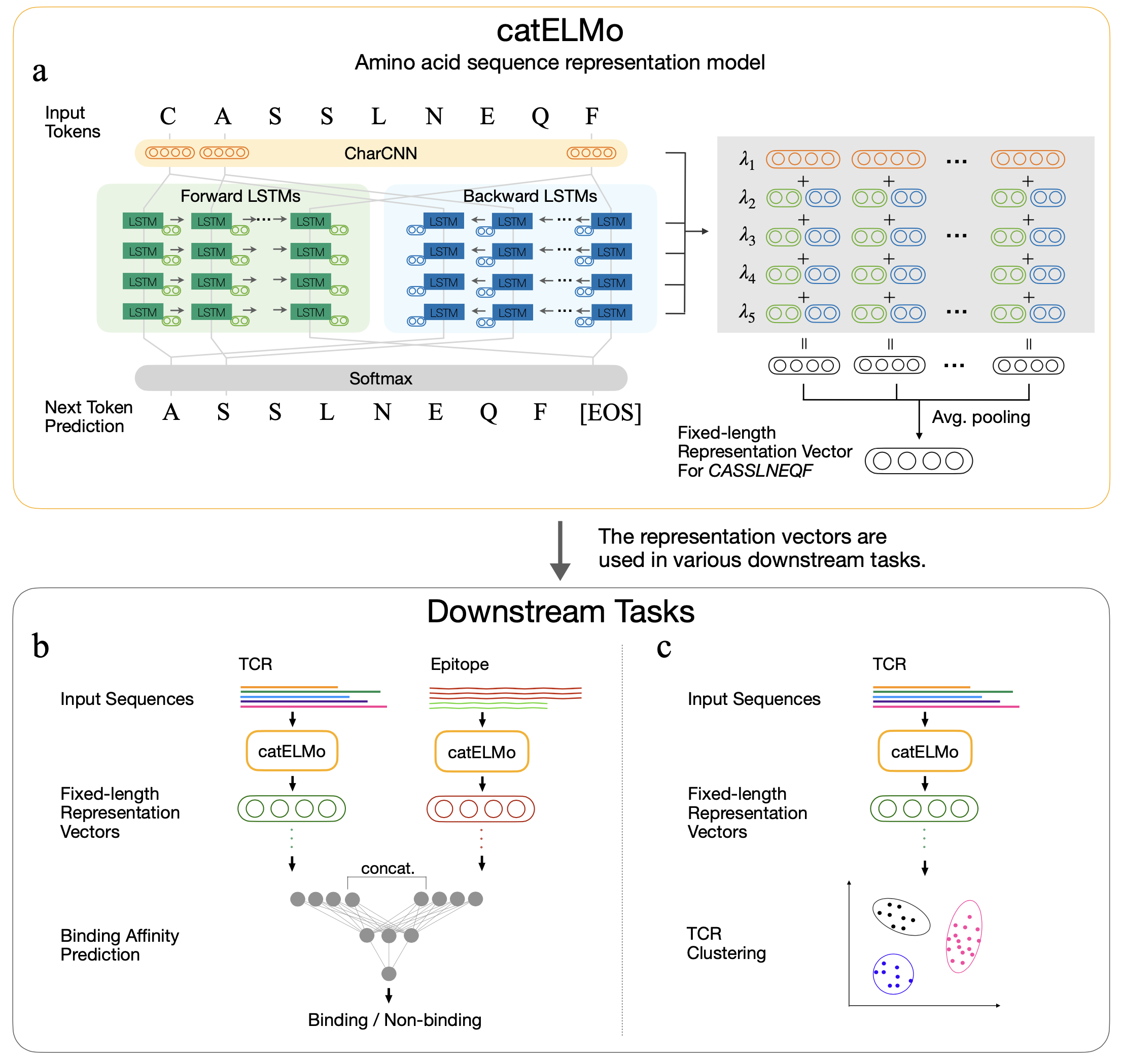
Le contexte de l'intégration des acides aminés avance l'analyse des interactions TCR-épitope
Pengfei Zhang 1,2 , Michael Cai 1,2 , Seojin Bang 2 , Heewook Lee 1,2
1 École d'informatique et de renseignement augmenté, Arizona State University, 2 BioDesign Institute, Arizona State University
Publié dans: Elife, 2023.
Papier | Code | Affiche | Diapositives | Présentation (YouTube)
git clone https://github.com/Lee-CBG/catELMo
cd catELMo/
conda create --name bap python=3.6.13
pip install pandas==1.1.5 tensorflow==2.6.0 keras==2.6.0 scikit-learn==0.24.2 tqdm
source activate bapdatasets .embedders .Un exemple de division d'épitopes
python -W ignore bap.py
--embedding catELMo_4_layers_1024
--split epitope
--gpu 0
--fraction 1
--seed 42Si vous utilisez ce code ou utilisez notre catelmo pour vos recherches, veuillez citer notre article:
@article {catelmobiorxiv,
author = {Pengfei Zhang and Seojin Bang and Michael Cai and Heewook Lee},
title = {Context-Aware Amino Acid Embedding Advances Analysis of TCR-Epitope Interactions},
elocation-id = {2023.04.12.536635},
year = {2023},
doi = {10.1101/2023.04.12.536635},
publisher = {Cold Spring Harbor Laboratory},
journal = {bioRxiv}
}
Ce travail est concédé sous licence Creative Commons Attribution-Noncommercial-Noderivatives 4.0 Licence internationale.