catELMo
1.0.0
Catelmo는 문맥 화 된 아미노산 표현을 배우는 양방향 아미노산 임베딩 모델로, 아미노산을 단어와 서열로 취급하여 문장으로 취급합니다. 이전 토큰이 주어진 다음 아미노산 토큰을 각각 예측하여 자체 감독 신호로 아미노산 서열의 패턴을 배웁니다. 4,173,895 TCR에 대한 교육을 받았습니다
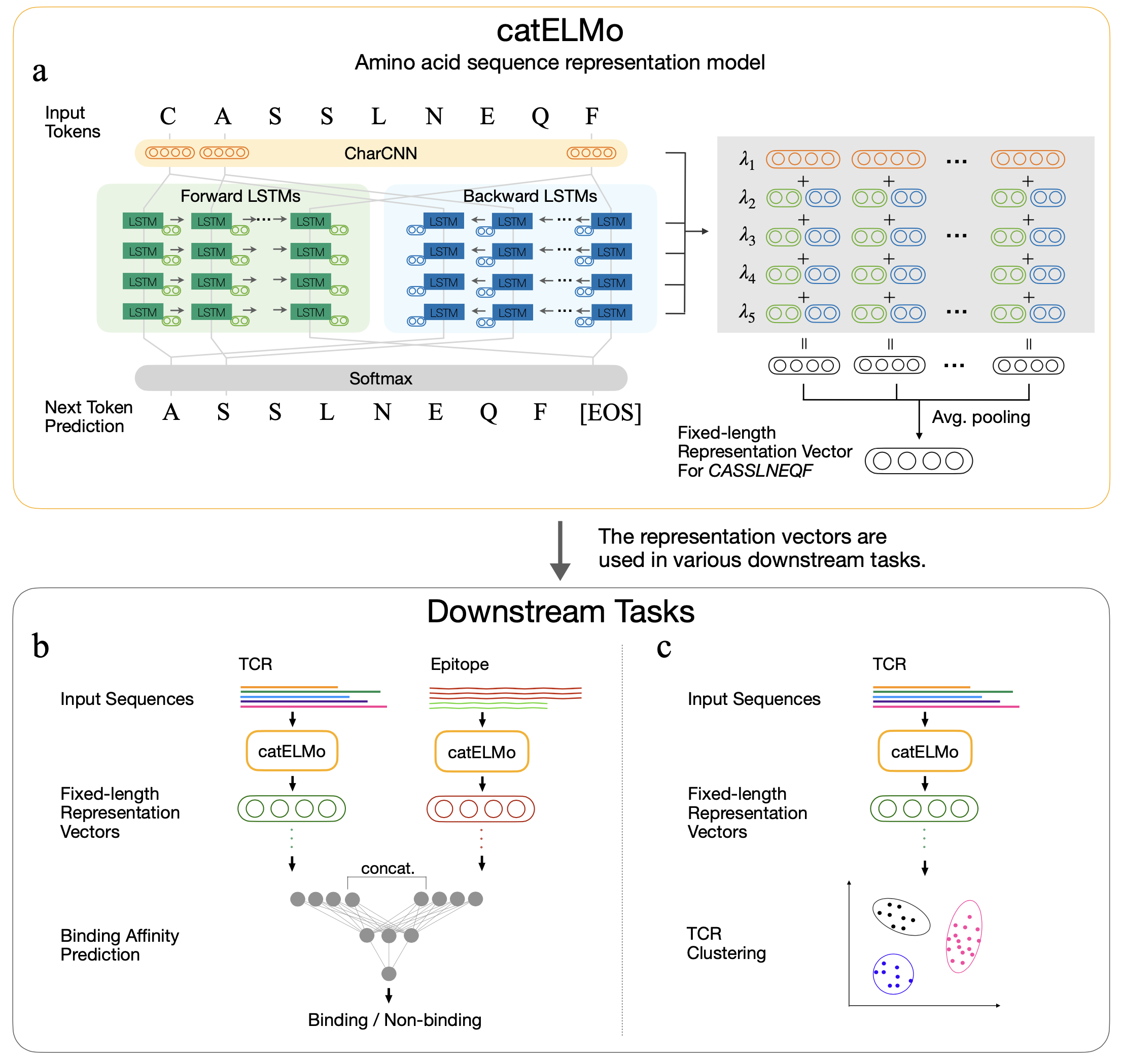
컨텍스트 인식 아미노산 임베딩 TCR- 에피토프 상호 작용의 분석
Pengfei Zhang 1,2 , Michael Cai 1,2 , Seojin Bang 2 , Hewook Lee 1,2
1 애리조나 주립 대학교, 애리조나 주립 대학교 (Arizona State University)의 컴퓨팅 및 증강 정보 학교
출판 : Elife, 2023.
종이 | 코드 | 포스터 | 슬라이드 | 프레젠테이션 (YouTube)
git clone https://github.com/Lee-CBG/catELMo
cd catELMo/
conda create --name bap python=3.6.13
pip install pandas==1.1.5 tensorflow==2.6.0 keras==2.6.0 scikit-learn==0.24.2 tqdm
source activate bapdatasets 폴더에서 교육 및 테스트 데이터를 다운로드하십시오.embedders 폴더의 지시에 따라 TCR 및 에피토프에 대한 임베딩을 얻습니다.에피토프 분할의 예
python -W ignore bap.py
--embedding catELMo_4_layers_1024
--split epitope
--gpu 0
--fraction 1
--seed 42이 코드를 사용하거나 연구에 카텔 모를 사용하는 경우 논문을 인용하십시오.
@article {catelmobiorxiv,
author = {Pengfei Zhang and Seojin Bang and Michael Cai and Heewook Lee},
title = {Context-Aware Amino Acid Embedding Advances Analysis of TCR-Epitope Interactions},
elocation-id = {2023.04.12.536635},
year = {2023},
doi = {10.1101/2023.04.12.536635},
publisher = {Cold Spring Harbor Laboratory},
journal = {bioRxiv}
}
이 작품은 Creative Commons Attribution-Noncommercial Noderivatives 4.0 International License에 따라 라이센스가 부여됩니다.