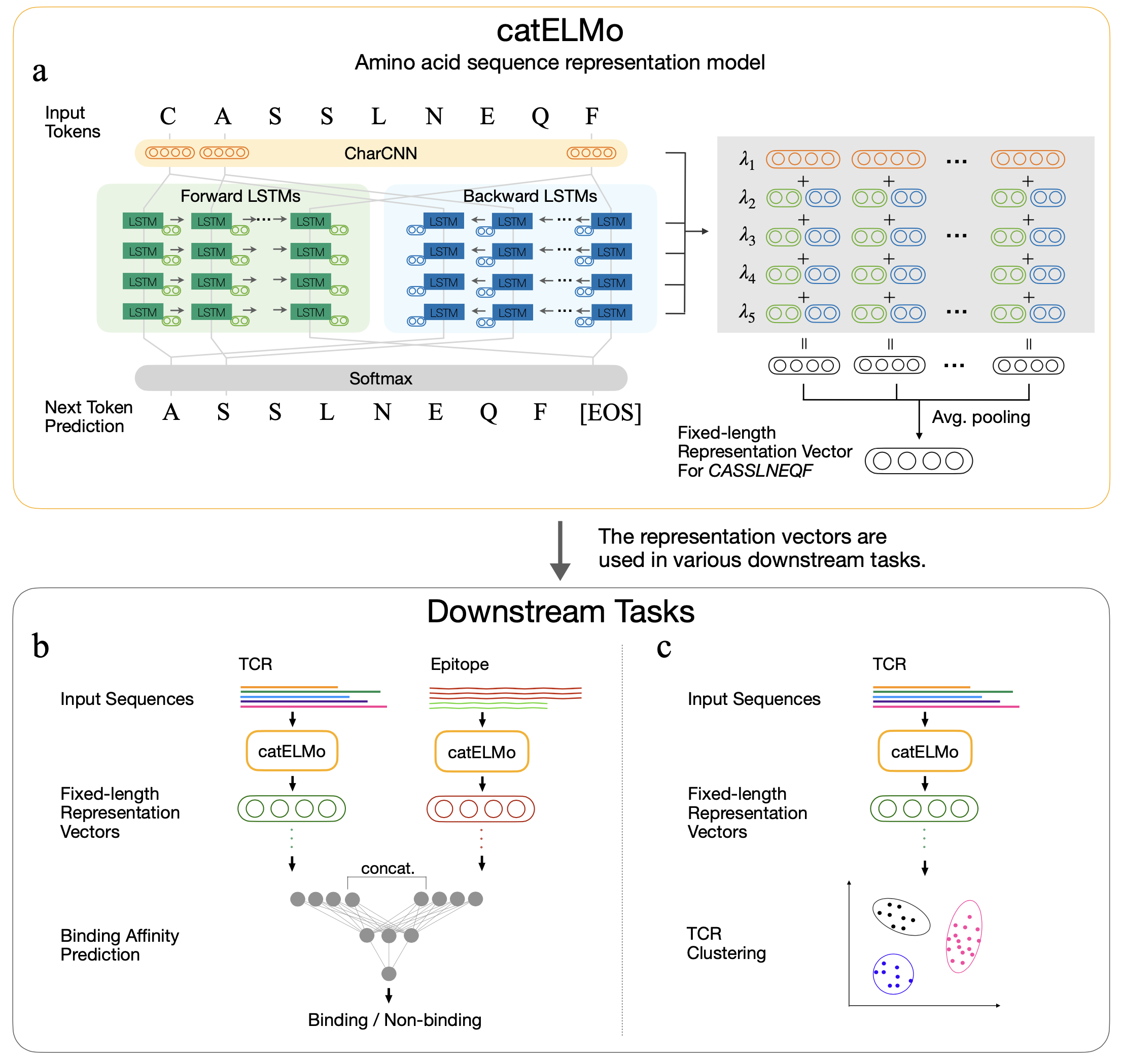
Catelmo-это двунаправленная модель аминокислот, которая изучает контекстуализированные аминокислотные представления, рассматривая аминокислоту как слово и последовательность как предложение. Он изучает паттерны аминокислотных последовательностей с его сигналом самоосудывания, предсказав каждый следующий токен аминокислот, учитывая его предыдущие токены. Он был обучен 4173 895 TCR
Анализ аминокислотных аминокислот-аминокислот аминокислота
Пенгфей Чжан 1,2 , Майкл Кай 1,2 , Сейдзин Банг 2 , Heewook Lee 1,2
1 Школа вычислительной и дополненной разведки, Университет штата Аризона, 2 Института биодизинга, Университет штата Аризона
Опубликовано в: Elife, 2023.
Бумага | Код | Плакат | Слайды | Презентация (YouTube)
git clone https://github.com/Lee-CBG/catELMo
cd catELMo/
conda create --name bap python=3.6.13
pip install pandas==1.1.5 tensorflow==2.6.0 keras==2.6.0 scikit-learn==0.24.2 tqdm
source activate bapdatasets .embedders .Пример для разделения эпитопа
python -W ignore bap.py
--embedding catELMo_4_layers_1024
--split epitope
--gpu 0
--fraction 1
--seed 42Если вы используете этот код или используете наш Catelmo для исследования, пожалуйста, укажите нашу статью:
@article {catelmobiorxiv,
author = {Pengfei Zhang and Seojin Bang and Michael Cai and Heewook Lee},
title = {Context-Aware Amino Acid Embedding Advances Analysis of TCR-Epitope Interactions},
elocation-id = {2023.04.12.536635},
year = {2023},
doi = {10.1101/2023.04.12.536635},
publisher = {Cold Spring Harbor Laboratory},
journal = {bioRxiv}
}
Эта работа лицензирована в соответствии с Creative Commons Attribution-Noncommercial-Noderivatives 4.0 International License.