Catelmo ist ein bidirektionales Aminosäure-Einbettungsmodell, das kontextualisierte Aminosäuredarstellungen lernt und eine Aminosäure als Wort und eine Sequenz als Satz behandelt. Es lernt Muster von Aminosäuresequenzen mit seinem Selbst-Supervision-Signal, indem sie jeweils die nächste Aminosäure-Token aufgrund seiner vorherigen Token vorhergesagt. Es wurde auf 4.173.895 TCR ausgebildet
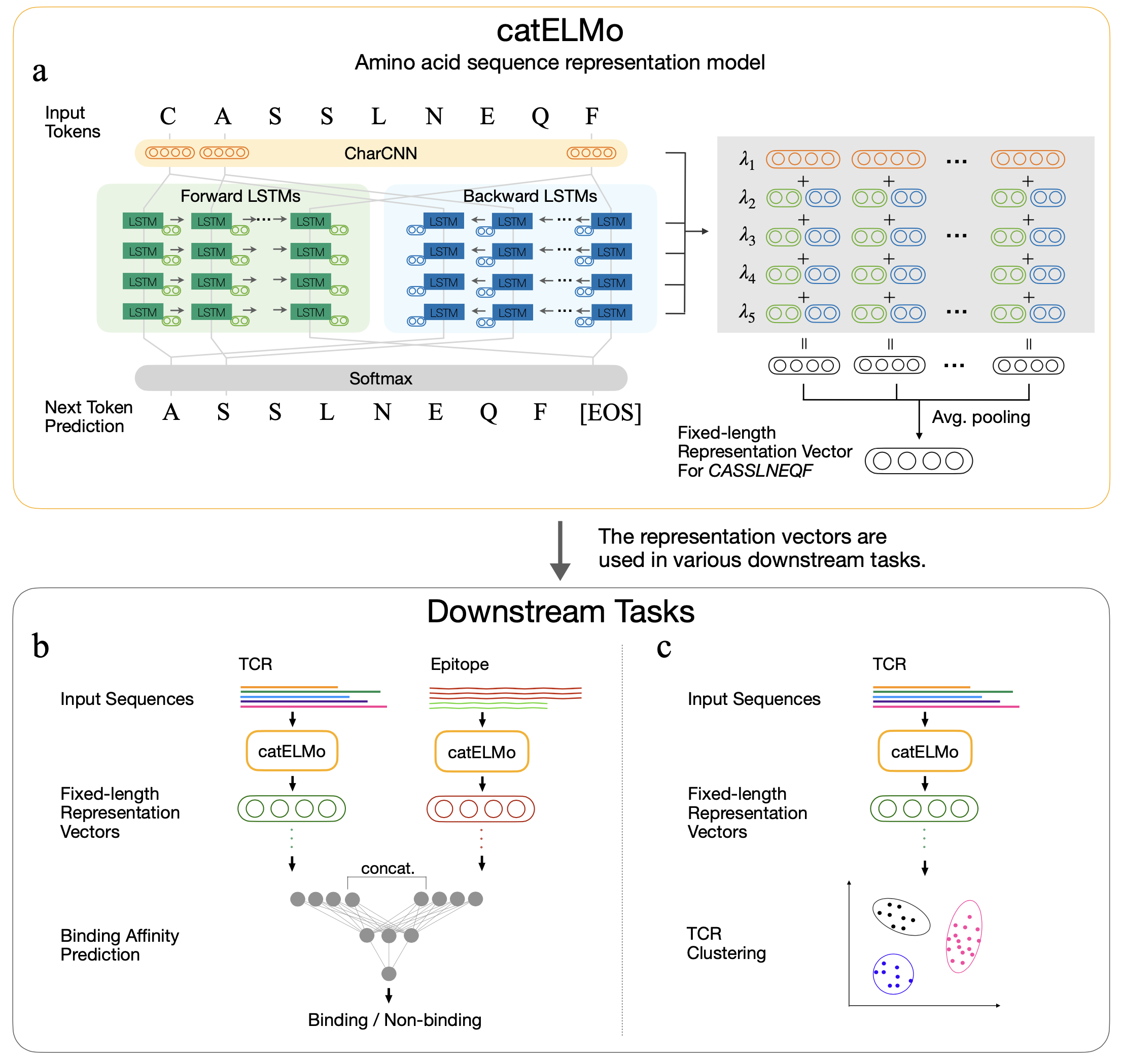
Kontextbewusste Aminosäure-Einbettungsanalyse der Analyse von TCR-Epitop-Wechselwirkungen
Pengfei Zhang 1,2 , Michael Cai 1,2 , Seojin Bang 2 , Heewook Lee 1,2
1 School of Computing and Augmented Intelligence, Arizona State University, 2 Biodesign Institute, Arizona State University
Veröffentlicht in: Elife, 2023.
Papier | Code | Poster | Folien | Präsentation (YouTube)
git clone https://github.com/Lee-CBG/catELMo
cd catELMo/
conda create --name bap python=3.6.13
pip install pandas==1.1.5 tensorflow==2.6.0 keras==2.6.0 scikit-learn==0.24.2 tqdm
source activate bapdatasets -Ordner herunter.embedders .Ein Beispiel für die Aufteilung von Epitopen
python -W ignore bap.py
--embedding catELMo_4_layers_1024
--split epitope
--gpu 0
--fraction 1
--seed 42Wenn Sie diesen Code verwenden oder unseren Catelmo für Ihre Recherche verwenden, zitieren Sie bitte unser Papier:
@article {catelmobiorxiv,
author = {Pengfei Zhang and Seojin Bang and Michael Cai and Heewook Lee},
title = {Context-Aware Amino Acid Embedding Advances Analysis of TCR-Epitope Interactions},
elocation-id = {2023.04.12.536635},
year = {2023},
doi = {10.1101/2023.04.12.536635},
publisher = {Cold Spring Harbor Laboratory},
journal = {bioRxiv}
}
Diese Arbeit ist im Rahmen einer kreativen Commons Attribution-Noncommercial-Noderivatives 4.0 International Lizenz lizenziert.