對廣闊的化學空間的自動計算分析對於諸如藥物發現和材料科學等眾多研究領域至關重要。最近採用了表示學習技術,其主要目的是生成複雜數據的緊湊而有益的數值表達。有效學習分子表示的一種方法是通過自然語言處理(NLP)算法處理基於弦的化學物質的符號。到目前為止,大多數提出的方法都將微笑符號用於此目的;但是,微笑與與有效性和魯棒性有關的許多問題有關,這可能會阻止該模型有效地發現隱藏數據中隱藏的知識。在這項研究中,我們提出了一種基於變壓器體系結構的化學語言模型,該模型利用100%有效,緊湊和表達的符號,自拍照作為輸入,以學習靈活和高質量的分子表示。自動型在200萬種藥物樣化合物中進行了預訓練,並針對各種分子財產預測任務進行了微調。我們的績效評估表明,在預測分子的水溶性和不良藥物反應的水溶性方面,自動士兵的表現優於所有競爭方法,包括基於圖學習的方法和基於笑容的化學語言模型。我們還可以觀察到通過降低維度學到的分子表示,這表明即使是預訓練的模型也可以區分具有不同結構特性的分子。我們共享自動型物作為程序化工具,以及其數據集和預訓練的模型。總體而言,我們的研究證明了在化學語言建模背景下使用自拍照符號的好處,並為設計和發現具有所需特徵的新型藥物候選者開闢了新的可能性。
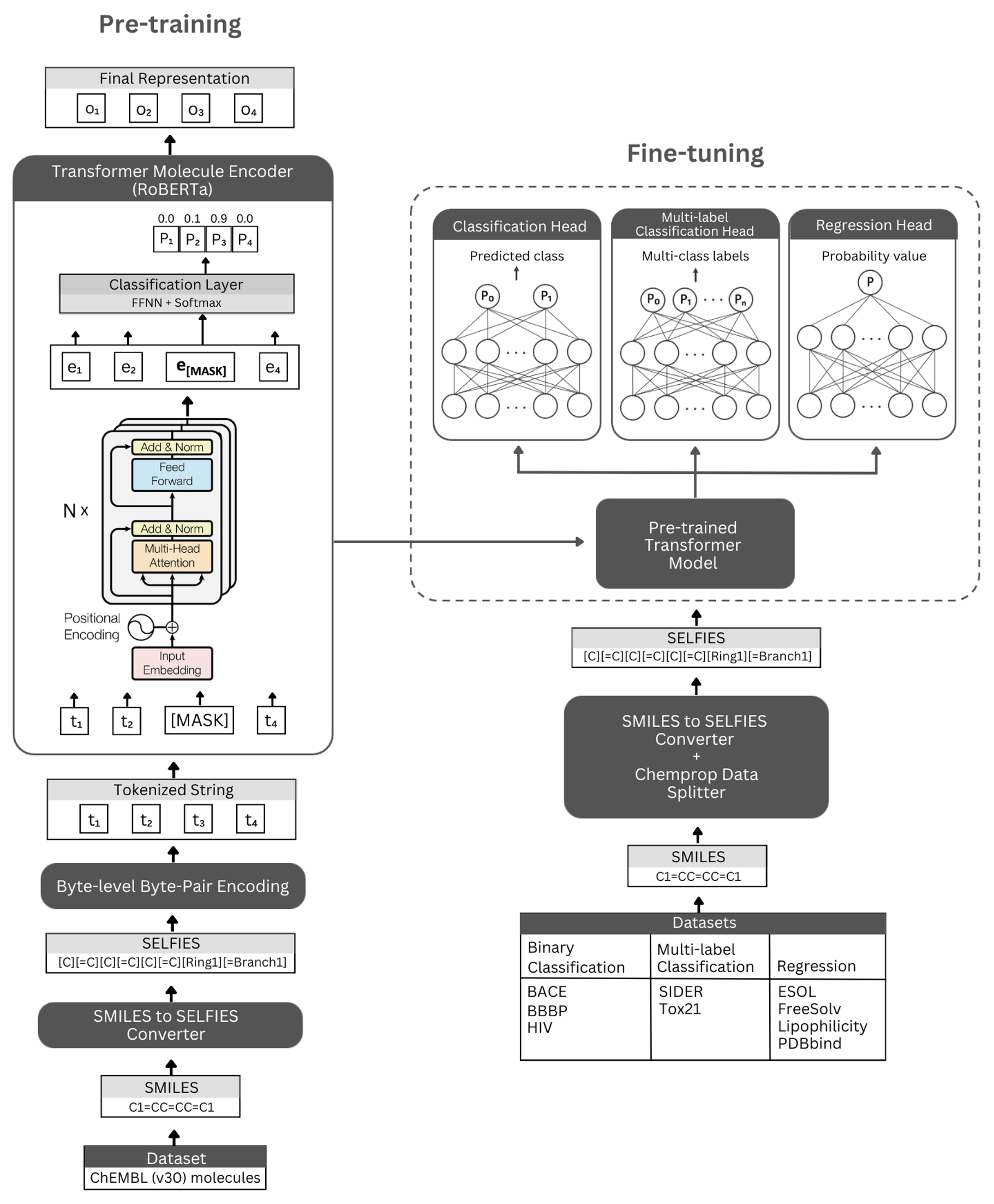
數字。自動架構的示意圖和實驗。左:自我監管的預訓練利用蒙版語言建模利用變壓器編碼器模塊來學習通過自拍照符號編碼的小分子的簡明和信息的表示。右:預先訓練的模型已在眾多基於分子屬性的分類和回歸任務上獨立進行了微調。
自動師基於Roberta Transformer架構,該體系結構利用與Bert相同的體系結構,但已發現某些修改可以改善模型性能或提供其他好處。一種修改是使用字節級字節對編碼(BPE)用於令牌化而不是字符級BPE。另一個是,羅伯塔(Roberta)在無視下一個句子預測(NSP)任務的同時,僅在蒙版語言建模(MLM)目標上進行預訓練。自我監管的預培訓模型(i)使用Transformer編碼器模塊來學習通過自拍照符號編碼的小分子的簡明和信息的代表性,以及(ii)使用預先修復模型的監督分類/回歸模型作為基礎作為基礎,並對許多分類和回歸基於基於回歸的基於基於回歸的分類物質預測性質。
我們的預訓練的編碼器模型被實現為“ Robertamaskedlm”,並以“ Robertafor-sequenceCeclassification”的形式實現。對於微調過程,自動架構包括預先培訓的Roberta模型作為基礎,將“ Robertacificationhead”類作為以下層(用於分類和回歸)。 “ Robertacificationhead”類由輟學層,密集層,Tanh激活函數,輟學層和最終線性層組成。在微調過程中,我們將預訓練的羅伯塔基本模型的序列輸出轉發到分類器。
我們強烈建議使用用於安裝依賴關係的Conda平台。安裝Conda後,請創建並激活具有依賴關係的環境,如下所示:
conda create -n SELFormer_env
conda activate SELFormer_env
conda env update --file data/requirements.yml
預先訓練的自動型模型可在此處下載。在此處可用。
您還可以使用預訓練的模型為自己的數據集生成嵌入式。為此,您需要分子的自拍照符號。您可以使用以下命令為您的微笑數據集生成自拍照符號。
如果要重現我們的代碼以生成Chembl30數據集的嵌入,則可以啟用Moalecule_dataset_smiles.zip和/或Morecule_dataset_selpies.zip文件在數據目錄中並將它們用作輸入Smiles和自拍照數據集。
python3 generate_selfies.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv
要使用預訓練的模型為自拍照分子數據集生成嵌入式,請運行以下命令:
python3 produce_embeddings.py --selfies_dataset=data/molecule_dataset_selfies.csv --model_file=data/pretrained_models/SELFormer --embed_file=data/embeddings.csv
可以在此處直接下載由我們最佳性能預訓練的分子數據模型生成的嵌入。
您還可以使用以下命令重新生成這些嵌入。
python3 get_moleculenet_embeddings.py --dataset_path=data/finetuning_datasets --model_file=data/pretrained_models/SELFormer
要預先訓練模型,請在下面運行命令。如果您有自拍照數據集,則可以通過將數據集的路徑直接使用到-selfies_dataset 。如果您有一個微笑數據集,則可以將數據集的路徑提供給-Smiles_dataset ,並且將在給出的路徑上創建自拍照表示。
python3 train_pretraining_model.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv --prepared_data_path=data/selfies_data.txt --bpe_path=data/BPETokenizer --roberta_fast_tokenizer_path=data/RobertaFastTokenizer --hyperparameters_path=data/pretraining_hyperparameters.yml --subset_size=100000
您可以使用下面的命令來微調各種分子屬性預測任務的預訓練模型。這些命令用於處理包含分子微笑表示的數據集。微笑表示形式應存儲在列中,並帶有名為“ Smiles”的標頭。您可以在數據/finetuning_datasets目錄中查看示例數據集。
二進制分類任務
要微調二進制分類數據集上的預訓練模型,請在下面運行命令。
python3 train_classification_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/classification/bbbp/bbbp.csv --save_to=data/finetuned_models/SELFormer_bbbp_classification --target_column_id=1 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
多標籤分類任務
要在多標籤分類數據集上微調預訓練的模型,請在下面運行命令。 RobertafastTokenizer文件應與預培訓模型相同的目錄存儲。
python3 train_classification_multilabel_model.py --model=data/saved_models/SELFormer --dataset=data/finetuning_datasets/classification/tox21/tox21.csv --save_to=data/finetuned_models/SELFormer_tox21_classification --use_scaffold=1 --batch_size=16 --num_epochs=25 --lr=5e-5 --wd=0
回歸任務
要在回歸數據集上微調預訓練的模型,請在下面運行命令。
python3 train_regression_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/regression/esol/esol.csv --save_to=data/finetuned_models/SELFormer_esol_regression --target_column_id=-1 --scaler=2 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
可以在此處下載微調的自動型模型。要使用這些模型進行預測,請按照下面的說明進行操作。
為了預測bace,BBBP和HIV數據集,請在下面運行命令。更改不同任務的指定參數。默認參數將在BBBP上加載微調模型。
python3 binary_class_pred.py --task=bbbp --model_name=data/finetuned_models/SELFormer_bbbp_scaffold_optimized --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/bbbp/bbbp_mock.csv --training_args=data/finetuned_models/SELFormer_bbbp_scaffold_optimized/training_args.bin
為了對TOX21和SIDE數據集進行預測,請在下面運行命令。更改不同任務的指定參數。默認參數將在SIDE上加載微調模型。
python3 multilabel_class_pred.py --task=sider --model_name=data/finetuned_models/SELFormer_sider_scaffold_optimized --pred_set=data/finetuning_datasets/classification/sider/sider_mock.csv --training_args=data/finetuned_models/SELFormer_sider_scaffold_optimized/training_args.bin --num_labels=27
為了對ESOL,FREESOLV,親脂性和PDBBIND數據集進行預測,請在下面運行命令。更改不同任務的指定參數。默認參數將在ESOL上加載微調模型。
python3 regression_pred.py --task=esol --model_name=data/finetuned_models/esol_regression --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/esol/esol_mock.csv --training_args=data/finetuned_models/esol_regression/training_args.bin
版權(C)2023 Hubiodatalab
該程序是免費的軟件:您可以根據自由軟件基金會發布的GNU通用公共許可證的條款對其進行重新分配和/或修改它,該版本是該許可證的版本3,或(按您的選項)任何以後的版本。
該程序的分佈是希望它將有用的,但沒有任何保修;即使沒有對特定目的的適銷性或適合性的隱含保證。有關更多詳細信息,請參見GNU通用公共許可證。
您應該已經收到了GNU通用公共許可證的副本以及此計劃。如果沒有,請參見http://www.gnu.org/licenses/。