膨大な化学空間の自動化された計算分析は、創薬や材料科学などの多くの研究分野にとって重要です。表現学習手法は、複雑なデータのコンパクトで有益な数値式を生成するという主要な目的で最近採用されています。分子表現を効率的に学習する1つのアプローチは、自然言語処理(NLP)アルゴリズムを介した化学物質の文字列ベースの表記を処理することです。これまで提案されている方法の大部分は、この目的のために笑顔の表記法を利用しています。ただし、笑顔は有効性と堅牢性に関連する多くの問題に関連しているため、モデルがデータに隠された知識を効果的に明らかにすることを妨げる可能性があります。この研究では、柔軟で高品質の分子表現を学ぶために、入力として100%有効でコンパクトで表現力豊かな表記法、セルフィーを利用するトランスアーキテクチャベースの化学言語モデルであるセルフマーを提案します。セルフマーは、200万人の薬物様化合物で事前に訓練されており、多様な分子特性予測タスクのために微調整されています。私たちのパフォーマンス評価により、セルフマーダーは、分子の水溶性と薬物反応の溶解度を予測する際に、グラフ学習ベースのアプローチや笑顔に基づく化学言語モデルを含むすべての競合する方法よりも優れていることが明らかになりました。また、次元の減少を介して自己監督によって学習した分子表現を視覚化しました。これは、事前に訓練されたモデルでさえ、構造特性が異なる分子を識別できることを示しました。セルフマーは、データセットと事前に訓練されたモデルとともに、プログラムツールとして共有しました。全体として、私たちの研究は、化学言語モデリングのコンテキストでセルフィー表記を使用することの利点を実証し、目的の特徴を持つ新しい薬物候補の設計と発見のための新しい可能性を開きます。
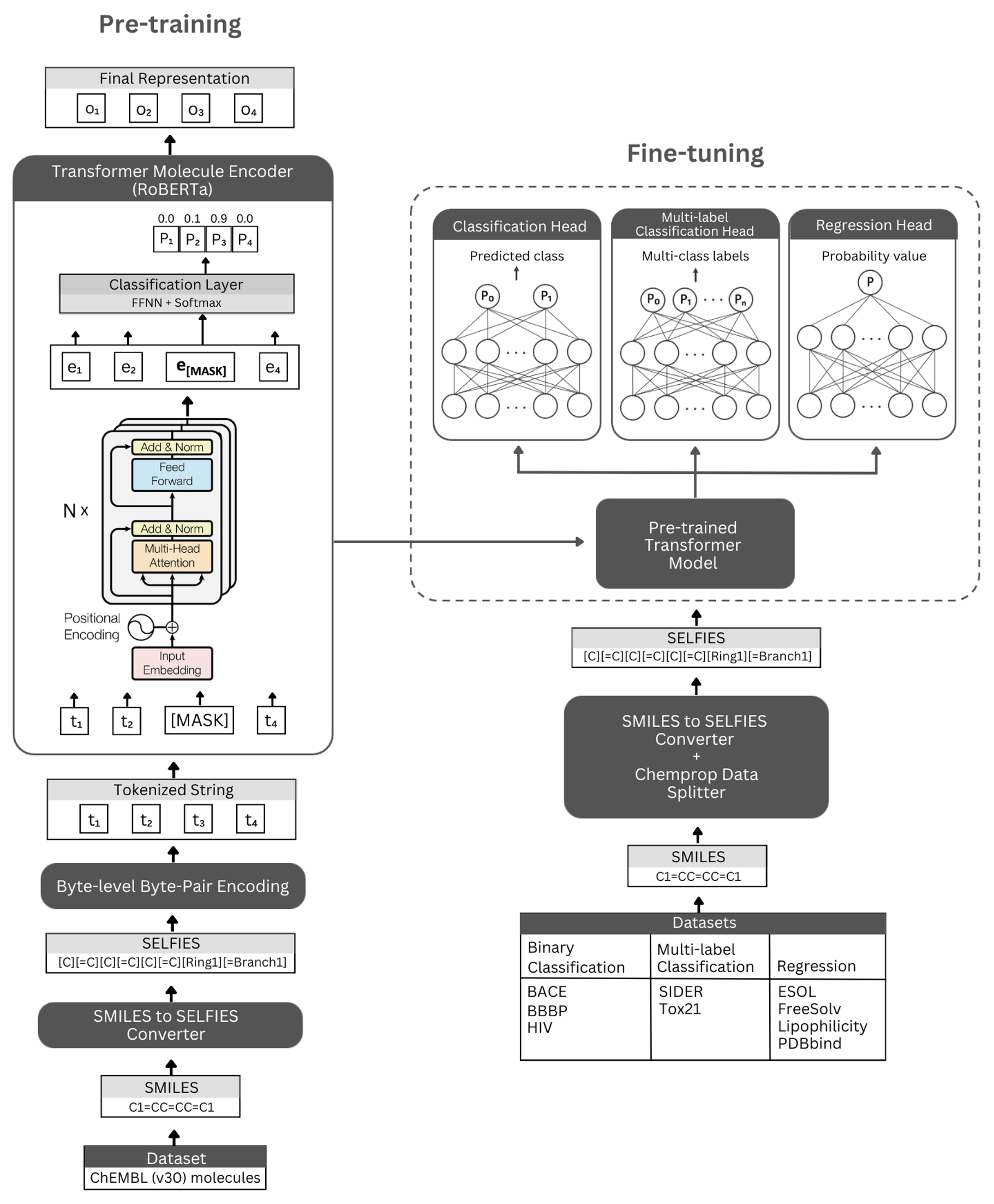
形。セルフマーアーキテクチャの概略図と実験された実験。左:自己監督の事前トレーニングは、自撮り表記によってコードされる小分子の簡潔で有益な表現を学習するためのマスクされた言語モデリングを介して、変圧器エンコーダーモジュールを利用しています。右:事前に訓練されたモデルは、多数の分子特性ベースの分類および回帰タスクで独立して微調整されています。
Self -Mormerは、Bertと同じアーキテクチャを利用するRoberta Transformer Architectureに基づいて構築されていますが、モデルのパフォーマンスを改善したり、他の利点を提供したりすることがわかった特定の変更があります。そのような変更の1つは、文字レベルのBPEではなく、トークン化にバイトレベルのバイトペアエンコード(BPE)の使用です。もう1つは、ロバータは、次の文予測(NSP)タスクを無視しながら、マスクされた言語モデリング(MLM)目的のみで事前に訓練されていることです。 Self-Mormerには、(i)セルフィー表記によってエンコードされた小分子の簡潔かつ有益な表現のために変圧器エンコーダモジュールを利用する自己監督の事前訓練モデル、および(ii)事前に定められたモデルをベースとして使用し、多数の分類および退行ベースの分子特性予測上で微調整する監視された分類/回帰モデルを持っています。
事前に訓練されたエンコーダーモデルは、「RobertAmaskedlm」および「robertafor seckesenceclassification」として微調整モデルとして実装されています。微調整プロセスのために、セルフマーサーアーキテクチャには、事前に訓練されたロバータモデルをそのベースとして含め、次のレイヤーとして(分類と回帰のために)「ロバートアクラシフィケーションヘッド」クラスを含めます。 「Robertaclassificationhead」クラスは、ドロップアウト層、密な層、タン活性化関数、ドロップアウト層、最終的な線形層で構成されています。微調整プロセス中に、事前に訓練されたロバータベースモデルのシーケンス出力を分類器に転送します。
依存関係をインストールするために、Condaプラットフォームを強くお勧めします。 Condaのインストール後、以下に定義する依存関係を持つ環境を作成およびアクティブ化してください。
conda create -n SELFormer_env
conda activate SELFormer_env
conda env update --file data/requirements.yml
事前に訓練されたセルフマーモデルは、こちらからダウンロードできます。私たちの最良のパフォーマンスモデルによって生成されるChembl30およびchembl33からのすべての分子の埋め込みは、こちらから入手できます。
事前に訓練されたモデルを使用して、独自のデータセットの埋め込みを生成することもできます。そのためには、分子の自撮り表記が必要になります。以下のコマンドを使用して、Smilesデータセットのセルフィー表記を生成できます。
Chembl30データセットの埋め込みを生成するためのコードを再現したい場合は、 Data DirectoryのMolecule_dataset_smiles.zipおよび/またはmolecule_dataset_selfies.zipファイルをそれぞれ入力スマイルとセルフィーデータセットとして使用できます。
python3 generate_selfies.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv
事前に訓練されたモデルを使用してセルフィー分子データセットの埋め込みを生成するには、次のコマンドを実行してください。
python3 produce_embeddings.py --selfies_dataset=data/molecule_dataset_selfies.csv --model_file=data/pretrained_models/SELFormer --embed_file=data/embeddings.csv
MoleCulenetデータの最高のパフォーマンスの事前に訓練されたモデルによって生成される埋め込みは、ここから直接ダウンロードできます。
以下のコマンドを使用して、これらの埋め込みを再生することもできます。
python3 get_moleculenet_embeddings.py --dataset_path=data/finetuning_datasets --model_file=data/pretrained_models/SELFormer
モデルを事前にトレーニングするには、以下のコマンドを実行してください。セルフィーデータセットがある場合は、データセットのパスを-selfies_datasetに提供することで直接使用できます。 Smiles Datasetがある場合は、データセットのパスを-smiles_Datasetに与えることができ、 Selfies_Datasetに与えられたパスでセルフィー表現が作成されます。
python3 train_pretraining_model.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv --prepared_data_path=data/selfies_data.txt --bpe_path=data/BPETokenizer --roberta_fast_tokenizer_path=data/RobertaFastTokenizer --hyperparameters_path=data/pretraining_hyperparameters.yml --subset_size=100000
以下のコマンドを使用して、さまざまな分子特性予測タスクの事前に訓練されたモデルを微調整できます。これらのコマンドは、分子の笑顔表現を含むデータセットを処理するために利用されます。 Smilesの表現は、「Smiles」という名前のヘッダー付きの列に保存する必要があります。 Data/Finetuning_Datasetsディレクトリのデータセットの例を見ることができます。
バイナリ分類タスク
バイナリ分類データセットで事前に訓練されたモデルを微調整するには、以下のコマンドを実行してください。
python3 train_classification_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/classification/bbbp/bbbp.csv --save_to=data/finetuned_models/SELFormer_bbbp_classification --target_column_id=1 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
マルチラベル分類タスク
マルチラベル分類データセットで事前に訓練されたモデルを微調整するには、以下のコマンドを実行してください。 RobertafastTokenizerファイルは、事前に訓練されたモデルと同じディレクトリに保存する必要があります。
python3 train_classification_multilabel_model.py --model=data/saved_models/SELFormer --dataset=data/finetuning_datasets/classification/tox21/tox21.csv --save_to=data/finetuned_models/SELFormer_tox21_classification --use_scaffold=1 --batch_size=16 --num_epochs=25 --lr=5e-5 --wd=0
回帰タスク
回帰データセットで事前に訓練されたモデルを微調整するには、以下のコマンドを実行してください。
python3 train_regression_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/regression/esol/esol.csv --save_to=data/finetuned_models/SELFormer_esol_regression --target_column_id=-1 --scaler=2 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
微調整されたセルフマーモデルは、こちらからダウンロードできます。これらのモデルで予測するには、以下の指示に従ってください。
BACE、BBBP、およびHIVデータセットのいずれかについて予測するには、以下のコマンドを実行してください。さまざまなタスクの指定された引数を変更します。デフォルトのパラメーターは、BBBPに微調整されたモデルをロードします。
python3 binary_class_pred.py --task=bbbp --model_name=data/finetuned_models/SELFormer_bbbp_scaffold_optimized --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/bbbp/bbbp_mock.csv --training_args=data/finetuned_models/SELFormer_bbbp_scaffold_optimized/training_args.bin
tox21とsiderデータセットのいずれかの予測を行うには、以下のコマンドを実行してください。さまざまなタスクの指定された引数を変更します。デフォルトのパラメーターは、Siderに微調整されたモデルをロードします。
python3 multilabel_class_pred.py --task=sider --model_name=data/finetuned_models/SELFormer_sider_scaffold_optimized --pred_set=data/finetuning_datasets/classification/sider/sider_mock.csv --training_args=data/finetuned_models/SELFormer_sider_scaffold_optimized/training_args.bin --num_labels=27
ESOL、FreeSolv、親油性、PDBBINDデータセットのいずれかについて予測するには、以下のコマンドを実行してください。さまざまなタスクの指定された引数を変更します。デフォルトのパラメーターは、ESOLに微調整されたモデルをロードします。
python3 regression_pred.py --task=esol --model_name=data/finetuned_models/esol_regression --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/esol/esol_mock.csv --training_args=data/finetuned_models/esol_regression/training_args.bin
著作権(c)2023 hubiodatalab
このプログラムはフリーソフトウェアです。フリーソフトウェアファンデーションの条件、ライセンスのバージョン3、または(オプションで)後のバージョンのいずれかで公開されているように、GNU一般公開ライセンスの条件の下でそれを再配布したり、変更したりできます。
このプログラムは、それが有用であることを期待して配布されますが、保証はありません。商品性や特定の目的に対するフィットネスの暗黙の保証さえありません。詳細については、GNU一般公開ライセンスを参照してください。
このプログラムとともに、GNU一般公開ライセンスのコピーを受け取る必要があります。そうでない場合は、http://www.gnu.org/licenses/を参照してください。