L'analyse de calcul automatisée du vaste espace chimique est essentielle pour de nombreux domaines de recherche tels que la découverte de médicaments et la science des matériaux. Les techniques d'apprentissage de la représentation ont récemment été utilisées dans l'objectif principal de générer des expressions numériques compactes et informatives de données complexes. Une approche pour apprendre efficacement les représentations moléculaires consiste à traiter les notations basées sur des chaînes de produits chimiques via des algorithmes de traitement du langage naturel (NLP). La majorité des méthodes proposées jusqu'à présent utilisent les notations des sourires à cette fin; Cependant, Smiles est associé à de nombreux problèmes liés à la validité et à la robustesse, ce qui peut empêcher le modèle de découvrir efficacement les connaissances cachées dans les données. Dans cette étude, nous proposons Selformer, un modèle de langage chimique basé sur une architecture de transformateur qui utilise une notation 100% valide, compacte et expressive, les selfies, en entrée, afin d'apprendre des représentations moléculaires flexibles et de haute qualité. L'auto-tempête est pré-formé sur deux millions de composés de type médicament et ajustés pour diverses tâches de prédiction des propriétés moléculaires. Notre évaluation des performances a révélé que l'auto-tempête surpasse toutes les méthodes concurrentes, y compris les approches basées sur l'apprentissage graphique et les modèles de langage chimique basé sur les sourires, sur la prédiction de la solubilité aqueuse des molécules et des effets indésirables des médicaments. Nous avons également visualisé les représentations moléculaires apprises par l'auto-tempête via une réduction de la dimensionnalité, qui a indiqué que même le modèle pré-formé peut discriminer les molécules avec des propriétés structurelles différentes. Nous avons partagé l'auto-tempête en tant qu'outil programmatique, ainsi que ses ensembles de données et ses modèles pré-formés. Dans l'ensemble, nos recherches démontrent l'avantage de l'utilisation des notations des selfies dans le contexte de la modélisation du langage chimique et ouvre de nouvelles possibilités pour la conception et la découverte de nouveaux candidats médicamenteux avec des caractéristiques souhaitées.
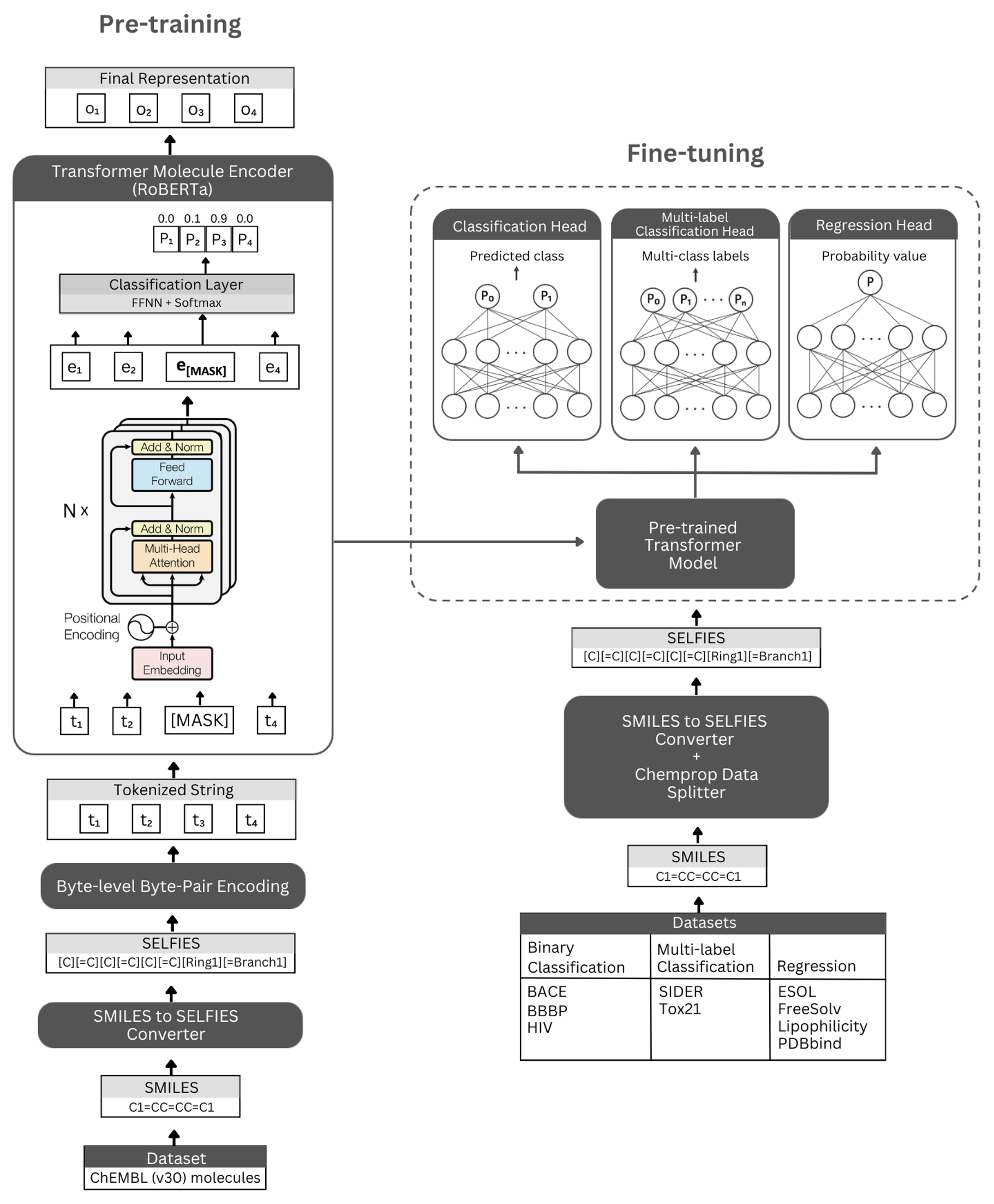
Chiffre. La représentation schématique de l'architecture auto-tempête et des expériences menées. Gauche: La pré-formation autopéralisée utilise le module de codeur de transformateur via la modélisation du langage masqué pour l'apprentissage des représentations concises et informatives de petites molécules codées par leur notation de selfies. À droite: le modèle pré-formé a été affiné indépendamment sur de nombreuses tâches de classification et de régression basées sur la propriété moléculaire.
Selformer est construit sur l'architecture Roberta Transformer, qui utilise la même architecture que Bert, mais avec certaines modifications qui ont été trouvées pour améliorer les performances du modèle ou offrir d'autres avantages. Une telle modification est l'utilisation du codage d'octets de niveau d'octets (BPE) pour la tokenisation au lieu du BPE au niveau du caractère. Un autre est que Roberta est pré-formé exclusivement sur l'objectif de modélisation du langage masqué (MLM) tout en ignorant la tâche de prédiction de phrase suivante (NSP). L'égodormer possède (i) des modèles pré-entraînés auto-supervisés qui utilisent le module de codeur de transformateur pour apprendre les représentations concises et informatives de petites molécules codées par leur notation de selfies, et (ii) des modèles de classification / régression supervisés qui utilisent le modèle pré-triageux comme base de tasks de prédiction moléculaire basée sur la propriété basée sur la propriété basée sur la propriété.
Nos modèles d'encodeur pré-formés sont mis en œuvre sous le nom de "RobertamaskEdlm" et les modèles affinés comme "RobertaRorseCenceClassification". Pour le processus de réglage fin, l'architecture auto-normale comprend le modèle Roberta pré-formé comme base et la classe "RobertAclassificationhead" comme les couches suivantes (pour la classification et la régression). La classe "RobertaClassificationhead" se compose d'une couche d'abandon, d'une couche dense, d'une fonction d'activation TANH, d'une couche d'abandon et d'une couche linéaire finale. Nous transmettant la sortie de séquence du modèle de base Roberta pré-formé au classificateur pendant le processus de réglage fin.
Nous recommandons fortement la plate-forme Conda pour installer des dépendances. Après l'installation de conda, veuillez créer et activer un environnement avec des dépendances telles que définies ci-dessous:
conda create -n SELFormer_env
conda activate SELFormer_env
conda env update --file data/requirements.yml
Des modèles auto-temporisés pré-formés sont disponibles en téléchargement ici. Des intégres de toutes les molécules de CHEMBL30 et CHEMBL33 générées par notre modèle le plus performant sont disponibles ici.
Vous pouvez également générer des intégres pour votre propre ensemble de données à l'aide des modèles pré-formés. Pour ce faire, vous aurez besoin de notations de selfies de vos molécules. Vous pouvez utiliser la commande ci-dessous pour générer des notations de selfies pour votre ensemble de données Smiles.
Si vous souhaitez reproduire notre code pour générer des intérêts de données CHEMBL30, vous pouvez dézip Molecule_dataset_Smiles.zip et / ou molécule_dataset_selties.zip Files dans le répertoire de données et les utiliser comme ensembles de données de sourires et de selfies, respectivement.
python3 generate_selfies.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv
Pour générer des intégres pour l'ensemble de données de molécules de selfies à l'aide d'un modèle pré-formé, veuillez exécuter la commande suivante:
python3 produce_embeddings.py --selfies_dataset=data/molecule_dataset_selfies.csv --model_file=data/pretrained_models/SELFormer --embed_file=data/embeddings.csv
Les intérêts générés par notre modèle pré-formé le plus performant pour les données de moléculénet peuvent être directement téléchargés ici.
Vous pouvez également générer ces intérêts en utilisant la commande ci-dessous.
python3 get_moleculenet_embeddings.py --dataset_path=data/finetuning_datasets --model_file=data/pretrained_models/SELFormer
Pour pré-entraîner un modèle, veuillez exécuter la commande ci-dessous. Si vous avez un ensemble de données de selfies, vous pouvez l'utiliser directement en donnant le chemin de l'ensemble de données à - selfies_dataset . Si vous avez un ensemble de données Smiles, vous pouvez donner le chemin de l'ensemble de données à --SMILS_DATASET et les représentations des selfies seront créées sur le chemin donné à --selries_dataset .
python3 train_pretraining_model.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv --prepared_data_path=data/selfies_data.txt --bpe_path=data/BPETokenizer --roberta_fast_tokenizer_path=data/RobertaFastTokenizer --hyperparameters_path=data/pretraining_hyperparameters.yml --subset_size=100000
Vous pouvez utiliser les commandes ci-dessous pour affiner un modèle pré-formé pour diverses tâches de prédiction des propriétés moléculaires. Ces commandes sont utilisées pour gérer des ensembles de données contenant des représentations de sourires des molécules. Les représentations des sourires doivent être stockées dans une colonne avec un en-tête nommé "Smiles". Vous pouvez voir l'exemple de jeux de données dans le répertoire Data / Finetuning_datasets .
Tâches de classification binaire
Pour affiner un modèle pré-formé sur un ensemble de données de classification binaire, veuillez exécuter la commande ci-dessous.
python3 train_classification_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/classification/bbbp/bbbp.csv --save_to=data/finetuned_models/SELFormer_bbbp_classification --target_column_id=1 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
Tâches de classification multi-étiquettes
Pour affiner un modèle pré-formé sur un ensemble de données de classification multi-étiquettes, veuillez exécuter la commande ci-dessous. Les fichiers RobertafastTokenizer doivent être stockés dans le même répertoire que le modèle pré-formé.
python3 train_classification_multilabel_model.py --model=data/saved_models/SELFormer --dataset=data/finetuning_datasets/classification/tox21/tox21.csv --save_to=data/finetuned_models/SELFormer_tox21_classification --use_scaffold=1 --batch_size=16 --num_epochs=25 --lr=5e-5 --wd=0
Tâches de régression
Pour affiner un modèle pré-formé sur un ensemble de données de régression, veuillez exécuter la commande ci-dessous.
python3 train_regression_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/regression/esol/esol.csv --save_to=data/finetuned_models/SELFormer_esol_regression --target_column_id=-1 --scaler=2 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
Des modèles auto-horribles affinés sont disponibles en téléchargement ici. Pour faire des prédictions avec ces modèles, veuillez suivre les instructions ci-dessous.
Pour faire des prédictions pour les ensembles de données BACE, BBBP et VIH, veuillez exécuter la commande ci-dessous. Modifiez les arguments indiqués pour différentes tâches. Les paramètres par défaut chargeront le modèle affiné sur BBBP.
python3 binary_class_pred.py --task=bbbp --model_name=data/finetuned_models/SELFormer_bbbp_scaffold_optimized --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/bbbp/bbbp_mock.csv --training_args=data/finetuned_models/SELFormer_bbbp_scaffold_optimized/training_args.bin
Pour faire des prédictions pour les ensembles de données TOX21 et SIDER, veuillez exécuter la commande ci-dessous. Modifiez les arguments indiqués pour différentes tâches. Les paramètres par défaut chargeront le modèle affiné sur Sider.
python3 multilabel_class_pred.py --task=sider --model_name=data/finetuned_models/SELFormer_sider_scaffold_optimized --pred_set=data/finetuning_datasets/classification/sider/sider_mock.csv --training_args=data/finetuned_models/SELFormer_sider_scaffold_optimized/training_args.bin --num_labels=27
Pour faire des prédictions pour les ensembles de données ESOL, Freesolv, Lipophilicité et PDBBIND, veuillez exécuter la commande ci-dessous. Modifiez les arguments indiqués pour différentes tâches. Les paramètres par défaut chargeront le modèle affiné sur ESOL.
python3 regression_pred.py --task=esol --model_name=data/finetuned_models/esol_regression --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/esol/esol_mock.csv --training_args=data/finetuned_models/esol_regression/training_args.bin
Copyright (C) 2023 Hubiodatalab
Ce programme est un logiciel gratuit: vous pouvez le redistribuer et / ou le modifier en vertu des termes de la licence publique générale GNU publiée par la Free Software Foundation, soit la version 3 de la licence, ou (à votre option) toute version ultérieure.
Ce programme est distribué dans l'espoir qu'il sera utile, mais sans aucune garantie; Sans même la garantie implicite de qualité marchande ou d'adéquation à un usage particulier. Voir la licence publique générale GNU pour plus de détails.
Vous devriez avoir reçu une copie de la licence publique générale GNU avec ce programme. Sinon, voir http://www.gnu.org/licenses/.