对广阔的化学空间的自动计算分析对于诸如药物发现和材料科学等众多研究领域至关重要。最近采用了表示学习技术,其主要目的是生成复杂数据的紧凑而有益的数值表达。有效学习分子表示的一种方法是通过自然语言处理(NLP)算法处理基于弦的化学物质的符号。到目前为止,大多数提出的方法都将微笑符号用于此目的;但是,微笑与与有效性和鲁棒性有关的许多问题有关,这可能会阻止该模型有效地发现隐藏数据中隐藏的知识。在这项研究中,我们提出了一种基于变压器体系结构的化学语言模型,该模型利用100%有效,紧凑和表达的符号,自拍照作为输入,以学习灵活和高质量的分子表示。自动型在200万种药物样化合物中进行了预训练,并针对各种分子财产预测任务进行了微调。我们的绩效评估表明,在预测分子的水溶性和不良药物反应的水溶性方面,自动士兵的表现优于所有竞争方法,包括基于图学习的方法和基于笑容的化学语言模型。我们还可以观察到通过降低维度学到的分子表示,这表明即使是预训练的模型也可以区分具有不同结构特性的分子。我们共享自动型物作为程序化工具,以及其数据集和预训练的模型。总体而言,我们的研究证明了在化学语言建模背景下使用自拍照符号的好处,并为设计和发现具有所需特征的新型药物候选者开辟了新的可能性。
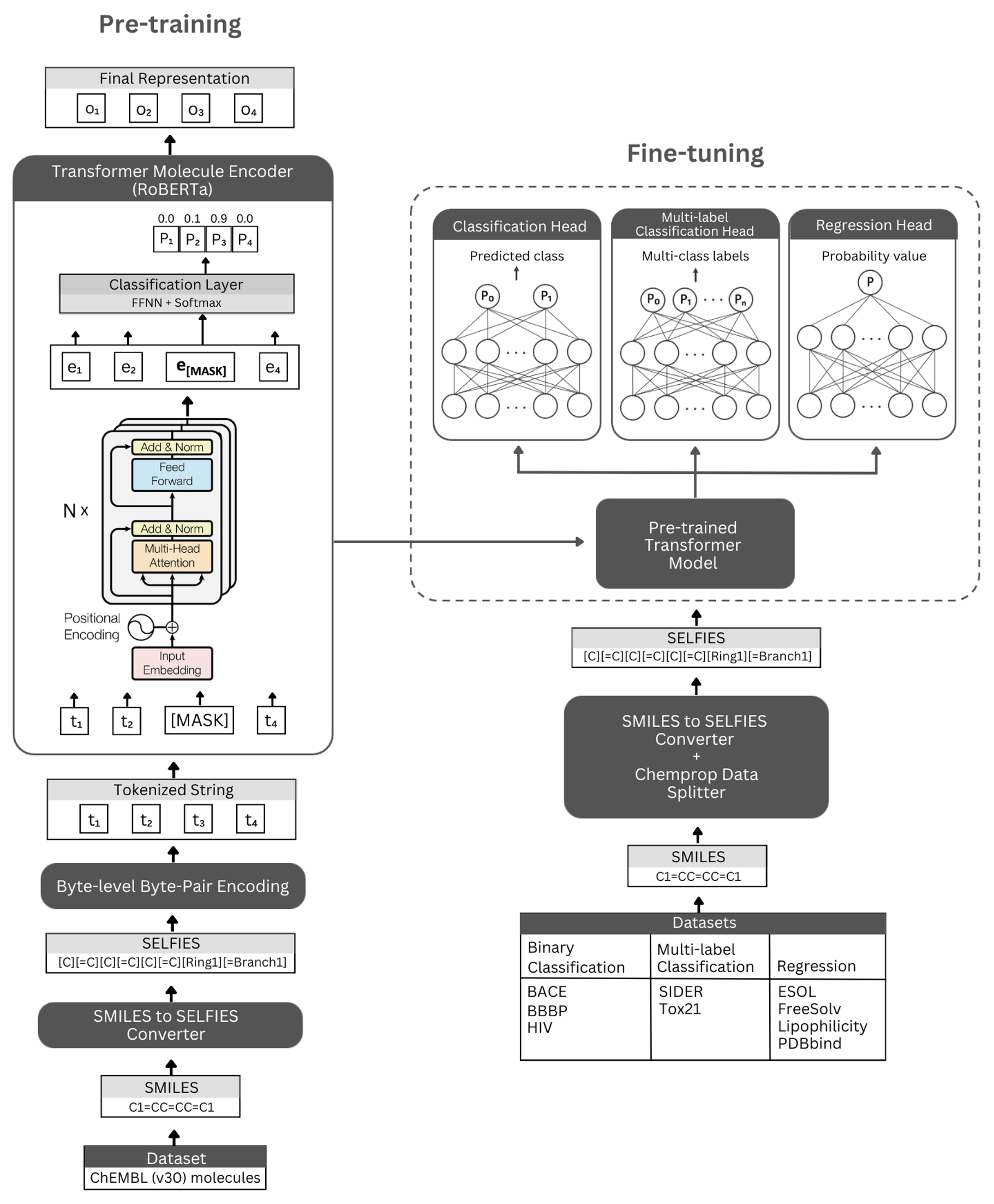
数字。自动架构的示意图和实验。左:自我监管的预训练利用蒙版语言建模利用变压器编码器模块来学习通过自拍照符号编码的小分子的简明和信息的表示。右:预先训练的模型已在众多基于分子属性的分类和回归任务上独立进行了微调。
自动师基于Roberta Transformer架构,该体系结构利用与Bert相同的体系结构,但已发现某些修改可以改善模型性能或提供其他好处。一种修改是使用字节级字节对编码(BPE)用于令牌化而不是字符级BPE。另一个是,罗伯塔(Roberta)在无视下一个句子预测(NSP)任务的同时,仅在蒙版语言建模(MLM)目标上进行预训练。自我监管的预培训模型(i)使用Transformer编码器模块来学习通过自拍照符号编码的小分子的简明和信息的代表性,以及(ii)使用预先修复模型的监督分类/回归模型作为基础作为基础,并对许多分类和回归基于基于回归的基于基于回归的分类物质预测性质。
我们的预训练的编码器模型被实现为“ Robertamaskedlm”,并以“ Robertafor-sequenceCeclassification”的形式实现。对于微调过程,自动架构包括预先培训的Roberta模型作为基础,将“ Robertacificationhead”类作为以下层(用于分类和回归)。 “ Robertacificationhead”类由辍学层,密集层,Tanh激活函数,辍学层和最终线性层组成。在微调过程中,我们将预训练的罗伯塔基本模型的序列输出转发到分类器。
我们强烈建议使用用于安装依赖关系的Conda平台。安装Conda后,请创建并激活具有依赖关系的环境,如下所示:
conda create -n SELFormer_env
conda activate SELFormer_env
conda env update --file data/requirements.yml
预先训练的自动型模型可在此处下载。在此处可用。
您还可以使用预训练的模型为自己的数据集生成嵌入式。为此,您需要分子的自拍照符号。您可以使用以下命令为您的微笑数据集生成自拍照符号。
如果要重现我们的代码以生成Chembl30数据集的嵌入,则可以启用Moalecule_dataset_smiles.zip和/或Morecule_dataset_selpies.zip文件在数据目录中并将它们用作输入Smiles和自拍照数据集。
python3 generate_selfies.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv
要使用预训练的模型为自拍照分子数据集生成嵌入式,请运行以下命令:
python3 produce_embeddings.py --selfies_dataset=data/molecule_dataset_selfies.csv --model_file=data/pretrained_models/SELFormer --embed_file=data/embeddings.csv
可以在此处直接下载由我们最佳性能预训练的分子数据模型生成的嵌入。
您还可以使用以下命令重新生成这些嵌入。
python3 get_moleculenet_embeddings.py --dataset_path=data/finetuning_datasets --model_file=data/pretrained_models/SELFormer
要预先训练模型,请在下面运行命令。如果您有自拍照数据集,则可以通过将数据集的路径直接使用到-selfies_dataset 。如果您有一个微笑数据集,则可以将数据集的路径提供给-Smiles_dataset ,并且将在给出的路径上创建自拍照表示。
python3 train_pretraining_model.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv --prepared_data_path=data/selfies_data.txt --bpe_path=data/BPETokenizer --roberta_fast_tokenizer_path=data/RobertaFastTokenizer --hyperparameters_path=data/pretraining_hyperparameters.yml --subset_size=100000
您可以使用下面的命令来微调各种分子属性预测任务的预训练模型。这些命令用于处理包含分子微笑表示的数据集。微笑表示形式应存储在列中,并带有名为“ Smiles”的标头。您可以在数据/finetuning_datasets目录中查看示例数据集。
二进制分类任务
要微调二进制分类数据集上的预训练模型,请在下面运行命令。
python3 train_classification_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/classification/bbbp/bbbp.csv --save_to=data/finetuned_models/SELFormer_bbbp_classification --target_column_id=1 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
多标签分类任务
要在多标签分类数据集上微调预训练的模型,请在下面运行命令。 RobertafastTokenizer文件应与预培训模型相同的目录存储。
python3 train_classification_multilabel_model.py --model=data/saved_models/SELFormer --dataset=data/finetuning_datasets/classification/tox21/tox21.csv --save_to=data/finetuned_models/SELFormer_tox21_classification --use_scaffold=1 --batch_size=16 --num_epochs=25 --lr=5e-5 --wd=0
回归任务
要在回归数据集上微调预训练的模型,请在下面运行命令。
python3 train_regression_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/regression/esol/esol.csv --save_to=data/finetuned_models/SELFormer_esol_regression --target_column_id=-1 --scaler=2 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
可以在此处下载微调的自动型模型。要使用这些模型进行预测,请按照下面的说明进行操作。
为了预测bace,BBBP和HIV数据集,请在下面运行命令。更改不同任务的指定参数。默认参数将在BBBP上加载微调模型。
python3 binary_class_pred.py --task=bbbp --model_name=data/finetuned_models/SELFormer_bbbp_scaffold_optimized --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/bbbp/bbbp_mock.csv --training_args=data/finetuned_models/SELFormer_bbbp_scaffold_optimized/training_args.bin
为了对TOX21和SIDE数据集进行预测,请在下面运行命令。更改不同任务的指定参数。默认参数将在SIDE上加载微调模型。
python3 multilabel_class_pred.py --task=sider --model_name=data/finetuned_models/SELFormer_sider_scaffold_optimized --pred_set=data/finetuning_datasets/classification/sider/sider_mock.csv --training_args=data/finetuned_models/SELFormer_sider_scaffold_optimized/training_args.bin --num_labels=27
为了对ESOL,FREESOLV,亲脂性和PDBBIND数据集进行预测,请在下面运行命令。更改不同任务的指定参数。默认参数将在ESOL上加载微调模型。
python3 regression_pred.py --task=esol --model_name=data/finetuned_models/esol_regression --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/esol/esol_mock.csv --training_args=data/finetuned_models/esol_regression/training_args.bin
版权(C)2023 Hubiodatalab
该程序是免费的软件:您可以根据自由软件基金会发布的GNU通用公共许可证的条款对其进行重新分配和/或修改它,该版本是该许可证的版本3,或(按您的选项)任何以后的版本。
该程序的分布是希望它将有用的,但没有任何保修;即使没有对特定目的的适销性或适合性的隐含保证。有关更多详细信息,请参见GNU通用公共许可证。
您应该已经收到了GNU通用公共许可证的副本以及此计划。如果没有,请参见http://www.gnu.org/licenses/。