Analisis komputasi otomatis ruang kimia yang luas sangat penting untuk berbagai bidang penelitian seperti penemuan obat dan ilmu material. Teknik pembelajaran representasi baru -baru ini telah digunakan dengan tujuan utama menghasilkan ekspresi numerik yang kompak dan informatif dari data kompleks. Salah satu pendekatan untuk mempelajari representasi molekuler secara efisien adalah memproses notasi berbasis string dari bahan kimia melalui algoritma pemrosesan bahasa alami (NLP). Mayoritas metode yang diusulkan sejauh ini menggunakan notasi senyum untuk tujuan ini; Namun, senyum dikaitkan dengan banyak masalah yang terkait dengan validitas dan ketahanan, yang dapat mencegah model dari secara efektif mengungkap pengetahuan yang tersembunyi dalam data. Dalam penelitian ini, kami mengusulkan Selformer, model bahasa kimia berbasis arsitektur transformator yang menggunakan notasi 100% yang valid, kompak dan ekspresif, selfie, sebagai input, untuk mempelajari representasi molekuler yang fleksibel dan berkualitas tinggi. Selformer dilatih sebelumnya pada dua juta senyawa seperti obat dan disesuaikan untuk beragam tugas prediksi properti molekuler. Evaluasi kinerja kami telah mengungkapkan bahwa, selformer mengungguli semua metode yang bersaing, termasuk pendekatan berbasis pembelajaran grafik dan model bahasa kimia berbasis senyum, pada memprediksi kelarutan molekul dan reaksi obat yang merugikan. Kami juga memvisualisasikan representasi molekuler yang dipelajari oleh sformer melalui reduksi dimensi, yang menunjukkan bahwa bahkan model pra-terlatih dapat membedakan molekul dengan sifat struktural yang berbeda. Kami berbagi Selformer sebagai alat terprogram, bersama dengan kumpulan data dan model pra-terlatih. Secara keseluruhan, penelitian kami menunjukkan manfaat menggunakan notasi selfie dalam konteks pemodelan bahasa kimia dan membuka kemungkinan baru untuk desain dan penemuan kandidat obat baru dengan fitur yang diinginkan.
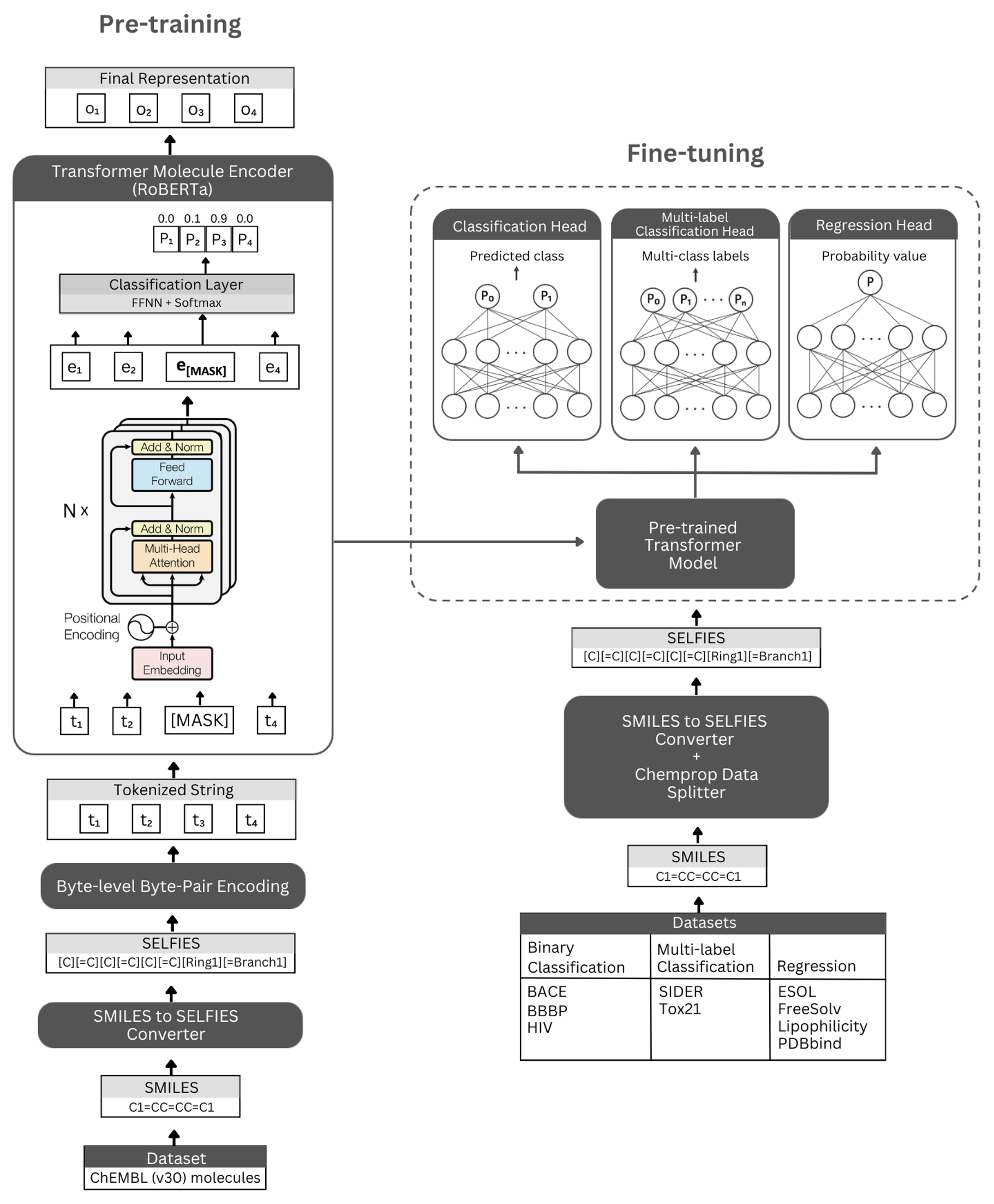
Angka. Representasi skematis dari arsitektur munafik dan eksperimen yang dilakukan. Kiri: Pra-pelatihan yang di-pelatihan sendiri menggunakan modul encoder transformator melalui pemodelan bahasa bertopeng untuk belajar secara ringkas dan representasi informatif dari molekul kecil yang dikodekan oleh notasi selfie mereka. Kanan: Model pra-terlatih telah disesuaikan secara independen pada berbagai tugas klasifikasi dan regresi berbasis properti.
Selformer dibangun di atas arsitektur Transformer Roberta, yang memanfaatkan arsitektur yang sama dengan Bert, tetapi dengan modifikasi tertentu yang telah ditemukan untuk meningkatkan kinerja model atau memberikan manfaat lainnya. Salah satu modifikasi tersebut adalah penggunaan encoding-byte-pair encoding (BPE) untuk tokenisasi alih-alih BPE tingkat karakter. Yang lain adalah bahwa, Roberta terlatih secara eksklusif pada tujuan pemodelan bahasa bertopeng (MLM) sambil mengabaikan tugas prediksi kalimat berikutnya (NSP). Selformer memiliki (i) model pra-terlatih yang di-latih sendiri yang memanfaatkan modul encoder transformator untuk belajar representasi ringkas dan informatif dari molekul kecil yang dikodekan oleh notasi selfie mereka, dan (ii) model klasifikasi/regresi yang diawasi yang menggunakan model prediksi pra-dasar.
Model Encoder pra-terlatih kami diimplementasikan sebagai "Robertamaskedlm" dan model penyempurnaan sebagai "RobertaforSeenceenceCeClasification". Untuk proses penyempurnaan, arsitektur selformer mencakup model Roberta terlatih sebagai basisnya, dan kelas "RobertacLasifificHead" sebagai lapisan berikut (untuk klasifikasi dan regresi). Kelas "RobertAcLasificifeHead" terdiri dari lapisan putus sekolah, lapisan padat, fungsi aktivasi tanh, lapisan putus sekolah, dan lapisan linier terakhir. Kami meneruskan output urutan model dasar Roberta yang sudah terlatih ke classifier selama proses penyempurnaan.
Kami sangat merekomendasikan platform Conda untuk menginstal dependensi. Mengikuti pemasangan Conda, silakan buat dan aktifkan lingkungan dengan dependensi sebagaimana didefinisikan di bawah ini:
conda create -n SELFormer_env
conda activate SELFormer_env
conda env update --file data/requirements.yml
Model selformer pra-terlatih tersedia untuk diunduh di sini. Embeddings dari semua molekul dari Chembl30 dan ChemBl33 yang dihasilkan oleh model kinerja terbaik kami tersedia di sini.
Anda juga dapat menghasilkan embeddings untuk dataset Anda sendiri menggunakan model pra-terlatih. Untuk melakukannya, Anda akan memerlukan notasi selfie tentang molekul Anda. Anda dapat menggunakan perintah di bawah ini untuk menghasilkan notasi selfie untuk dataset senyum Anda.
Jika Anda ingin mereproduksi kode kami untuk menghasilkan embeddings dari dataset ChemBl30, Anda dapat unzip molekul_dataset_smiles.zip dan/atau molekul_dataset_selfies.zip file dalam direktori data dan menggunakannya masing -masing sebagai input senyum dan dataset selfies.
python3 generate_selfies.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv
Untuk menghasilkan embeddings untuk dataset molekul selfie menggunakan model pra-terlatih, silakan jalankan perintah berikut:
python3 produce_embeddings.py --selfies_dataset=data/molecule_dataset_selfies.csv --model_file=data/pretrained_models/SELFormer --embed_file=data/embeddings.csv
Embeddings yang dihasilkan oleh model pra-terlatih terbaik kami untuk data molekulenet dapat langsung diunduh di sini.
Anda juga dapat menghasilkan kembali embeddings ini menggunakan perintah di bawah ini.
python3 get_moleculenet_embeddings.py --dataset_path=data/finetuning_datasets --model_file=data/pretrained_models/SELFormer
Untuk pra-pelatihan model, silakan jalankan perintah di bawah ini. Jika Anda memiliki dataset selfie, Anda dapat menggunakannya secara langsung dengan memberikan jalur dataset ke --selfies_dataset . Jika Anda memiliki dataset senyum, Anda dapat memberikan jalur dataset ke --smiles_dataset dan representasi selfie akan dibuat di jalur yang diberikan kepada --elfies_dataset .
python3 train_pretraining_model.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv --prepared_data_path=data/selfies_data.txt --bpe_path=data/BPETokenizer --roberta_fast_tokenizer_path=data/RobertaFastTokenizer --hyperparameters_path=data/pretraining_hyperparameters.yml --subset_size=100000
Anda dapat menggunakan perintah di bawah ini untuk menyempurnakan model pra-terlatih untuk berbagai tugas prediksi properti molekuler. Perintah -perintah ini digunakan untuk menangani set data yang berisi representasi senyum molekul. Representasi senyum harus disimpan dalam kolom dengan header bernama "Smiles". Anda dapat melihat contoh set data di direktori data/finetuning_datasets .
Tugas klasifikasi biner
Untuk menyempurnakan model pra-terlatih pada dataset klasifikasi biner, silakan jalankan perintah di bawah ini.
python3 train_classification_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/classification/bbbp/bbbp.csv --save_to=data/finetuned_models/SELFormer_bbbp_classification --target_column_id=1 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
Tugas klasifikasi multi-label
Untuk menyempurnakan model pra-terlatih pada dataset klasifikasi multi-label, silakan jalankan perintah di bawah ini. File RobertafastTokenizer harus disimpan di direktori yang sama dengan model pra-terlatih.
python3 train_classification_multilabel_model.py --model=data/saved_models/SELFormer --dataset=data/finetuning_datasets/classification/tox21/tox21.csv --save_to=data/finetuned_models/SELFormer_tox21_classification --use_scaffold=1 --batch_size=16 --num_epochs=25 --lr=5e-5 --wd=0
Tugas regresi
Untuk menyempurnakan model pra-terlatih pada dataset regresi, silakan jalankan perintah di bawah ini.
python3 train_regression_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/regression/esol/esol.csv --save_to=data/finetuned_models/SELFormer_esol_regression --target_column_id=-1 --scaler=2 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
Model selformer yang disempurnakan tersedia untuk diunduh di sini. Untuk membuat prediksi dengan model -model ini, silakan ikuti instruksi di bawah ini.
Untuk membuat prediksi untuk dataset BACE, BBBP, dan HIV, silakan jalankan perintah di bawah ini. Ubah argumen yang ditunjukkan untuk tugas yang berbeda. Parameter default akan memuat model fine-tuned pada BBBP.
python3 binary_class_pred.py --task=bbbp --model_name=data/finetuned_models/SELFormer_bbbp_scaffold_optimized --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/bbbp/bbbp_mock.csv --training_args=data/finetuned_models/SELFormer_bbbp_scaffold_optimized/training_args.bin
Untuk membuat prediksi untuk dataset Tox21 dan Sider, silakan jalankan perintah di bawah ini. Ubah argumen yang ditunjukkan untuk tugas yang berbeda. Parameter default akan memuat model fine-tuned pada Sider.
python3 multilabel_class_pred.py --task=sider --model_name=data/finetuned_models/SELFormer_sider_scaffold_optimized --pred_set=data/finetuning_datasets/classification/sider/sider_mock.csv --training_args=data/finetuned_models/SELFormer_sider_scaffold_optimized/training_args.bin --num_labels=27
Untuk membuat prediksi untuk dataset ESOL, Freesolv, Lipofilisitas, dan PDBBind, silakan jalankan perintah di bawah ini. Ubah argumen yang ditunjukkan untuk tugas yang berbeda. Parameter default akan memuat model yang disesuaikan dengan ESOL.
python3 regression_pred.py --task=esol --model_name=data/finetuned_models/esol_regression --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/esol/esol_mock.csv --training_args=data/finetuned_models/esol_regression/training_args.bin
Hak Cipta (C) 2023 Hubiodatalab
Program ini adalah perangkat lunak gratis: Anda dapat mendistribusikannya kembali dan/atau memodifikasinya berdasarkan ketentuan lisensi publik umum GNU seperti yang diterbitkan oleh Yayasan Perangkat Lunak Gratis, baik versi 3 lisensi, atau (pada opsi Anda) versi selanjutnya.
Program ini didistribusikan dengan harapan akan bermanfaat, tetapi tanpa jaminan apa pun; bahkan tanpa jaminan tersirat dari dapat diperjualbelikan atau kebugaran untuk tujuan tertentu. Lihat Lisensi Publik Umum GNU untuk lebih jelasnya.
Anda seharusnya menerima salinan Lisensi Publik Umum GNU bersama dengan program ini. Jika tidak, lihat http://www.gnu.org/licenses/.