يعد التحليل الحسابي الآلي للمساحة الكيميائية الشاسعة أمرًا بالغ الأهمية للعديد من مجالات الأبحاث مثل اكتشاف الأدوية وعلوم المواد. تم استخدام تقنيات تعلم التمثيل مؤخرًا بالهدف الأساسي المتمثل في توليد تعبيرات رقمية مضغوطة وغنية بالمعلومات للبيانات المعقدة. تتمثل إحدى الأساليب في تعلم التمثيل الجزيئي بكفاءة في معالجة الترميزات القائمة على السلسلة للمواد الكيميائية عبر خوارزميات معالجة اللغة الطبيعية (NLP). غالبية الأساليب المقترحة حتى الآن تستخدم رموز الابتسامات لهذا الغرض ؛ ومع ذلك ، ترتبط الابتسامات بالعديد من المشكلات المتعلقة بالصلاحية والمتانة ، مما قد يمنع النموذج من الكشف عن المعرفة المخفية بشكل فعال في البيانات. في هذه الدراسة ، نقترح العاصفة الذاتية ، وهو نموذج لغة كيميائية قائم على بنية المحولات يستخدم تدوينًا سيلفيًا صالحًا وضغوطًا ومضغوطًا بنسبة 100 ٪ ، كمدخلات ، من أجل تعلم التمثيل الجزيئي المرن وعالي الجودة. يتم تدريب Selformer مسبقًا على مليوني مركب يشبه المخدرات ويتم ضبطه جيدًا لمهام التنبؤ بالممتلكات الجزيئية المتنوعة. لقد كشف تقييم الأداء الخاص بنا بأنه يتفوق على جميع الأساليب المتنافسة ، بما في ذلك المقاربات القائمة على التعلم على الرسم البياني ونماذج اللغة الكيميائية القائمة على الابتسامات ، على التنبؤ بالذوبان المائي للجزيئات وتفاعلات الدواء الضارة. لقد تصورنا أيضًا التمثيل الجزيئي الذي تعلمته العاصفة الذاتية عن طريق الحد من الأبعاد ، والذي أشار إلى أنه حتى النموذج الذي تم تدريبه مسبقًا يمكن أن يميز الجزيئات ذات الخصائص الهيكلية المختلفة. شاركنا العاصفة الذاتية كأداة برمجية ، إلى جانب مجموعات البيانات والنماذج التي تم تدريبها مسبقًا. بشكل عام ، يوضح أبحاثنا الاستفادة من استخدام الرموز الشخصية في سياق نمذجة اللغة الكيميائية وتفتح إمكانيات جديدة لتصميم واكتشاف مرشحين مخدرات جديدة مع الميزات المرغوبة.
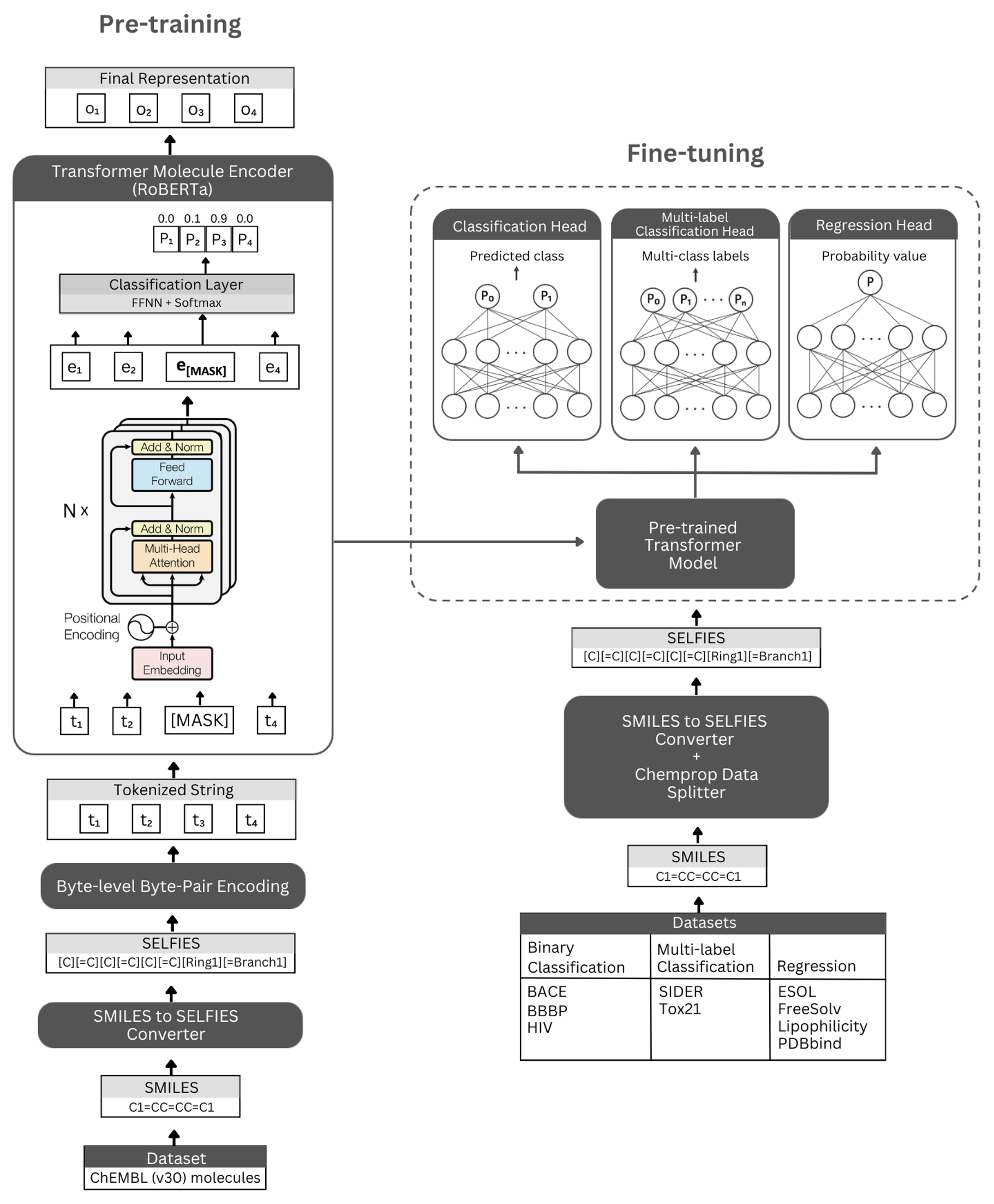
شكل. التمثيل التخطيطي للهندسة المعمارية للثمن الذاتي والتجارب التي أجريت. اليسار: يستخدم التدريب المسبق للإشراف ذاتيًا وحدة تشفير المحولات عبر نمذجة اللغة المقنعة لتعلم تمثيلات موجزة وغنية بالمعلومات للجزيئات الصغيرة المشفرة بواسطة تدوين صورهم الشخصية. إلى اليمين: تم ضبط النموذج الذي تم تدريبه مسبقًا بشكل مستقل على العديد من مهام التصنيف والانحدار الجزيئي.
تم بناء Selformer على بنية Transformer Roberta ، التي تستخدم نفس الهندسة المعمارية مثل Bert ، ولكن مع بعض التعديلات التي تم العثور عليها لتحسين أداء النموذج أو توفير فوائد أخرى. أحد هذه التعديلات هو استخدام ترميز زوج البايت على مستوى البايت (BPE) للرمز المميز بدلاً من BPE على مستوى الأحرف. آخر هو أن روبرتا تدرب مسبقًا على وجه الحصر على هدف نمذجة اللغة المقنعة (MLM) مع تجاهل مهمة التنبؤ بالجملة التالية (NSP). يحتوي العاصف الذاتي على نماذج (1) التي يتم تدريبها على ذاتيا والتي تستخدم وحدة تشفير المحولات لتعلم تمثيلات موجزة وغنية بالمعلومات للجزيئات الصغيرة المشفرة بواسطة تدوين صورهم الشخصية ، و (2) نماذج التصنيف/الانحدار الخاضعة للإشراف التي تستخدم النموذج المسبق لتنسيق الأساس والتواطؤ المتمثل في إمكانية الإبلاغ عن الموجهات القائمة على التصنيف والانتعاش.
يتم تنفيذ نماذج التشفير المدربة قبل التدريب على أنها "RobertamaskedLM" ونماذج صقلها مثل "RobertaforsequencyClassification". بالنسبة لعملية الضبط ، تتضمن بنية العاصفة الذاتية نموذج Roberta الذي تم تدريبه مسبقًا كقاعدته ، وفئة "RobertAclassificationhead" كطبقات التالية (للتصنيف والانحدار). تتكون فئة "RobertAclassificationhead" من طبقة التسرب ، وطبقة كثيفة ، ووظيفة تنشيط TANH ، وطبقة التسرب ، وطبقة خطية نهائية. نرسل توجيه الناتج التسلسل لنموذج قاعدة روبرتا المدربة مسبقًا إلى المصنف أثناء عملية التأكيد.
نوصي بشدة بمنصة كوندا لتثبيت التبعيات. بعد تثبيت كوندا ، يرجى إنشاء وتنشيط بيئة مع تبعيات كما هو موضح أدناه:
conda create -n SELFormer_env
conda activate SELFormer_env
conda env update --file data/requirements.yml
تتوفر نماذج العاصفة الذاتية التي تم تدريبها مسبقًا للتنزيل هنا. تضمينات جميع الجزيئات من Chembl30 و Chembl33 التي يتم إنشاؤها بواسطة أفضل نموذج أداء لدينا متوفرة هنا.
يمكنك أيضًا إنشاء تضمينات لمجموعة البيانات الخاصة بك باستخدام النماذج التي تم تدريبها مسبقًا. للقيام بذلك ، ستحتاج إلى رموز صور شخصية للجزيئات الخاصة بك. يمكنك استخدام الأمر أدناه لإنشاء رموز صور شخصية لمجموعة بيانات الابتسامات الخاصة بك.
إذا كنت ترغب في إعادة إنتاج التعليمات البرمجية الخاصة بنا لإنشاء تضمينات من مجموعة بيانات ChemBl30 ، فيمكنك إلغاء الضغط على molecule_dataset_smiles.zip و/أو molecule_dataset_selfies.zip في دليل البيانات واستخدامها كمجموعات بيانات مبتسمات و Seflies ، على التوالي.
python3 generate_selfies.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv
لإنشاء التضمينات لمجموعة بيانات جزيء Selfies باستخدام نموذج تم تدريبه مسبقًا ، يرجى تشغيل الأمر التالي:
python3 produce_embeddings.py --selfies_dataset=data/molecule_dataset_selfies.csv --model_file=data/pretrained_models/SELFormer --embed_file=data/embeddings.csv
يمكن تنزيل التضمينات التي تم إنشاؤها بواسطة أفضل نموذج لدينا مسبقًا لبيانات الجزيئات مباشرة هنا.
يمكنك أيضًا إعادة توحيد هذه التضمينات باستخدام الأمر أدناه.
python3 get_moleculenet_embeddings.py --dataset_path=data/finetuning_datasets --model_file=data/pretrained_models/SELFormer
لتدريب نموذج مسبقًا ، يرجى تشغيل الأمر أدناه. إذا كان لديك مجموعة بيانات صور شخصية ، فيمكنك استخدامها مباشرة من خلال منح مسار مجموعة البيانات إلى -seelfies_dataset . إذا كان لديك مجموعة بيانات للابتسامات ، فيمكنك إعطاء مسار مجموعة البيانات إلى -smiles_dataset وسيتم إنشاء تمثيلات صور سيلفي على المسار المقدم -seelfies_dataset .
python3 train_pretraining_model.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv --prepared_data_path=data/selfies_data.txt --bpe_path=data/BPETokenizer --roberta_fast_tokenizer_path=data/RobertaFastTokenizer --hyperparameters_path=data/pretraining_hyperparameters.yml --subset_size=100000
يمكنك استخدام الأوامر أدناه لضبط نموذج تم تدريبه مسبقًا لمختلف مهام التنبؤ بالممتلكات الجزيئية. يتم استخدام هذه الأوامر للتعامل مع مجموعات البيانات التي تحتوي على تمثيلات للابتسامات للجزيئات. يجب تخزين تمثيلات الابتسامات في عمود برأس يسمى "الابتسامات". يمكنك رؤية مثال مجموعات البيانات في دليل البيانات/finetuning_datasets .
مهام التصنيف الثنائية
لضبط نموذج تدريب مسبقًا على مجموعة بيانات التصنيف الثنائية ، يرجى تشغيل الأمر أدناه.
python3 train_classification_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/classification/bbbp/bbbp.csv --save_to=data/finetuned_models/SELFormer_bbbp_classification --target_column_id=1 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
مهام التصنيف متعددة العلامات
لضبط نموذج تدريب مسبقًا على مجموعة بيانات تصنيف متعددة العلامات ، يرجى تشغيل الأمر أدناه. يجب تخزين ملفات RobertaFastTokenizer في نفس الدليل مثل النموذج الذي تم تدريبه مسبقًا.
python3 train_classification_multilabel_model.py --model=data/saved_models/SELFormer --dataset=data/finetuning_datasets/classification/tox21/tox21.csv --save_to=data/finetuned_models/SELFormer_tox21_classification --use_scaffold=1 --batch_size=16 --num_epochs=25 --lr=5e-5 --wd=0
مهام الانحدار
لضبط نموذج تدريب مسبقًا على مجموعة بيانات الانحدار ، يرجى تشغيل الأمر أدناه.
python3 train_regression_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/regression/esol/esol.csv --save_to=data/finetuned_models/SELFormer_esol_regression --target_column_id=-1 --scaler=2 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
تتوفر نماذج العواصف الذاتية التي تم ضبطها للتنزيل هنا. لتقديم تنبؤات مع هذه النماذج ، يرجى اتباع التعليمات أدناه.
لتقديم تنبؤات إما لبياس و BBBP وفيروس نقص المناعة البشرية ، يرجى تشغيل الأمر أدناه. تغيير الحجج المشار إليها للمهام المختلفة. سوف تقوم المعلمات الافتراضية بتحميل نموذج ضبط دقيق على BBBP.
python3 binary_class_pred.py --task=bbbp --model_name=data/finetuned_models/SELFormer_bbbp_scaffold_optimized --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/bbbp/bbbp_mock.csv --training_args=data/finetuned_models/SELFormer_bbbp_scaffold_optimized/training_args.bin
لتقديم تنبؤات لمجموعات بيانات Tox21 و Sider ، يرجى تشغيل الأمر أدناه. تغيير الحجج المشار إليها للمهام المختلفة. ستقوم المعلمات الافتراضية بتحميل نموذج ضبط دقيق على Sider.
python3 multilabel_class_pred.py --task=sider --model_name=data/finetuned_models/SELFormer_sider_scaffold_optimized --pred_set=data/finetuning_datasets/classification/sider/sider_mock.csv --training_args=data/finetuned_models/SELFormer_sider_scaffold_optimized/training_args.bin --num_labels=27
لعمل تنبؤات إما esol و freesolv و lipophilicity و pdbbind ، يرجى تشغيل الأمر أدناه. تغيير الحجج المشار إليها للمهام المختلفة. ستقوم المعلمات الافتراضية بتحميل نموذج ضبط دقيق على ESOL.
python3 regression_pred.py --task=esol --model_name=data/finetuned_models/esol_regression --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/esol/esol_mock.csv --training_args=data/finetuned_models/esol_regression/training_args.bin
حقوق الطبع والنشر (C) 2023 Hubiodatalab
هذا البرنامج هو برنامج مجاني: يمكنك إعادة توزيعه و/أو تعديله بموجب شروط رخصة GNU العامة العامة كما تم نشرها من قبل مؤسسة البرمجيات المجانية ، إما الإصدار 3 من الترخيص ، أو (في خيارك) أي إصدار لاحق.
يتم توزيع هذا البرنامج على أمل أن يكون مفيدًا ، ولكن بدون أي ضمان ؛ بدون حتى الضمان الضمني للتسويق أو اللياقة لغرض معين. راجع رخصة GNU العامة لمزيد من التفاصيل.
يجب أن تكون قد تلقيت نسخة من رخصة GNU العامة العامة مع هذا البرنامج. إذا لم يكن الأمر كذلك ، راجع http://www.gnu.org/licenses/.