Die automatisierte rechnerische Analyse des riesigen chemischen Raums ist für zahlreiche Forschungsbereiche wie Arzneimittelentdeckung und Materialwissenschaft von entscheidender Bedeutung. Repräsentationslerntechniken wurden kürzlich mit dem Hauptziel der Generierung kompakter und informativer numerischer Ausdrücke komplexer Daten eingesetzt. Ein Ansatz zum effizienten Erlernen molekularer Darstellungen ist die Verarbeitungssting-basierte Notationen von Chemikalien über natürliche Sprachverarbeitung (NLP) -Algorithmen. Die Mehrheit der bisher vorgeschlagenen Methoden nutzen für diesen Zweck das Lächeln. Lächeln ist jedoch mit zahlreichen Problemen im Zusammenhang mit Gültigkeit und Robustheit verbunden, was verhindern kann, dass das Modell das in den Daten verborgene Wissen effektiv aufdeckt. In dieser Studie schlagen wir Selformer vor, ein auf Transformator architekturbasierter chemischer Sprachmodell, das eine 100% gültige, kompakte und ausdrucksstarke Notation, Selfies, als Input verwendet, um flexible und qualitativ hochwertige molekulare Darstellungen zu lernen. Selformer ist auf zwei Millionen medikamentenähnliche Verbindungen vorgeschrieben und für verschiedene Vorhersageaufgaben für molekulare Immobilienfunktionen fein abgestimmt. Unsere Leistungsbewertung hat gezeigt, dass Selbstormer alle konkurrierenden Methoden übertrifft, einschließlich graphisch-lernbasierter Ansätze und lächelnder chemischer Sprachmodelle, bei der Vorhersage der wässrigen Löslichkeit von Molekülen und unerwünschten Arzneimittelreaktionen. Wir haben auch molekulare Darstellungen visualisiert, die von Selbstormer durch Dimensionalitätsreduktion erlernt wurden, was darauf hinwies, dass selbst das vorgebreitete Modell Moleküle mit unterschiedlichen strukturellen Eigenschaften unterscheiden kann. Wir haben Selformer als programmatisches Tool zusammen mit seinen Datensätzen und vorgeborenen Modellen geteilt. Insgesamt zeigt unsere Forschung den Vorteil der Verwendung der Selfies -Notationen im Kontext der modellierten chemischen Sprachmodellierung und eröffnet neue Möglichkeiten für das Design und die Entdeckung neuartiger Arzneimittelkandidaten mit gewünschten Merkmalen.
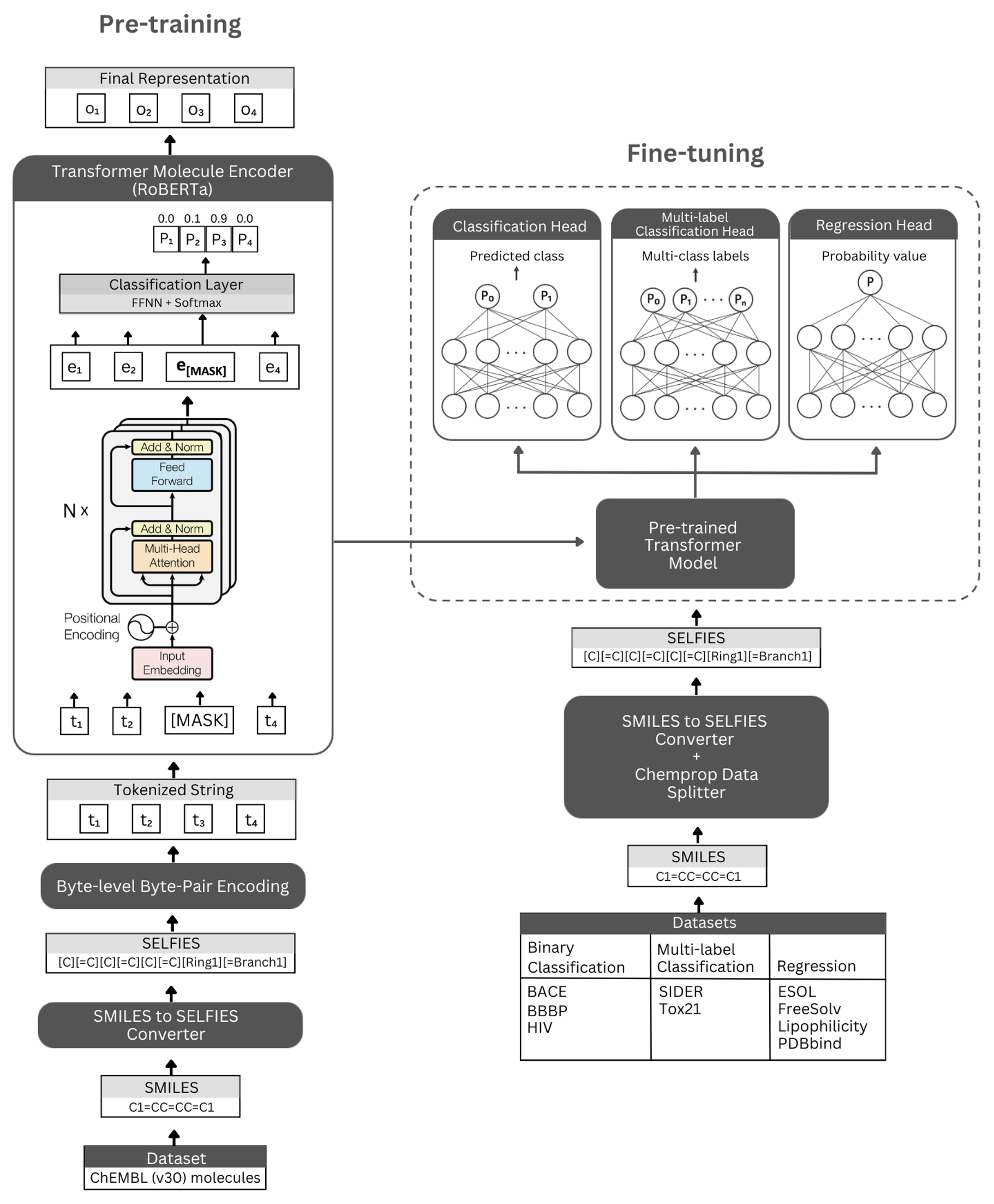
Figur. Die schematische Darstellung der Selbstormer -Architektur und der durchgeführten Experimente. Links: Die selbstverletzte Vorausbildung verwendet das Transformator-Encoder-Modul über maskierte Sprachmodellierung zum Lernen präziser und informativer Darstellungen kleiner Moleküle, die von ihren Selfies-Notation codiert werden. Rechts: Das vorgebreitete Modell wurde unabhängig von zahlreichen klassifizierenden und Regressionsaufgaben auf molekularer Eigenschaft auf der Basis von Molekulareigenschaften fein abgestimmt.
Selformer basiert auf der Roberta Transformator Architecture, die dieselbe Architektur wie Bert verwendet, aber mit bestimmten Modifikationen, die sich zur Verbesserung der Modellleistung oder anderen Vorteilen befinden. Eine solche Modifikation ist die Verwendung von Byte-Pair-Codierung (BPE) auf Byte-Ebene für Tokenisierung anstelle von BPE auf Zeichenebene. Eine andere ist, dass Roberta ausschließlich auf das Ziel der maskierten Sprachmodellierung (MLM) ausgebildet ist, während die Aufgabe der nächsten Satzvorhersage (NSP) nicht berücksichtigt wird. Selformer hat (i) selbst überträgte vorgebrachte Modelle, die das Transformator-Encoder-Modul zum Lernen präziser und informativer Darstellungen von kleinen Molekülen verwenden, die durch ihre Selfies-Notation codiert sind, und (ii) beaufsichtigte Klassifizierungs-/Regressionsmodelle, die das vorgezogene Modell als Basis und feinstimmig auf zahlreichen Klassifizierungs- und Regressions-basierten molekularen molekularen Molekulations-Molekulations-Molekulations-Taschen verwenden.
Unsere vorgebauten Encoder-Modelle werden als "Robertamaskedlm" und Feinabstimmungsmodelle als "Robertaforsequencklassifizierung" implementiert. Für den Feinabstimmungsprozess umfasst die SelfOrmer-Architektur das vorgebildete Roberta-Modell als Basis und die Klasse "Robertaclassificationhead" als folgende Schichten (zur Klassifizierung und Regression). Die Klasse "Robertaclassificationhead" besteht aus einer Tropfenschicht, einer dichten Schicht, einer TANH -Aktivierungsfunktion, einer Dropout -Schicht und einer endgültigen linearen Schicht. Wir leiten die Sequenzausgabe des vorgeborenen Roberta-Basismodells während des Feinabstimmungsprozesses an den Klassifikator weiter.
Wir empfehlen die Conda -Plattform für die Installation von Abhängigkeiten. Nach der Installation von Conda erstellen und aktivieren Sie eine Umgebung mit Abhängigkeiten wie unten definiert:
conda create -n SELFormer_env
conda activate SELFormer_env
conda env update --file data/requirements.yml
Vorausgebildete Selbstormer-Modelle können hier heruntergeladen werden. Hier sind Einbettungen aller Moleküle aus Chembl30 und Chembl33, die durch unser Best -Performing -Modell erzeugt werden, sind hier erhältlich.
Mit den vorgebreiteten Modellen können Sie auch Einbettungen für Ihren eigenen Datensatz generieren. Um dies zu tun, brauchen Sie Selfies -Notationen Ihrer Moleküle. Sie können den folgenden Befehl verwenden, um Selfies -Notationen für Ihren Smiles -Datensatz zu generieren.
Wenn Sie unseren Code für die Generierung von Einbettungen von Chembl30 -Datensatz reproduzieren möchten, können Sie Molekule_Dataset_Smiles.zip und/oder Molecule_Dataset_Selies.zip -Dateien im Datenverzeichnis entpucken und als Eingabedatensätze verwenden.
python3 generate_selfies.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv
Um Emetten für den Selfies-Molekül-Datensatz mit einem vorgebildeten Modell zu generieren, führen Sie bitte den folgenden Befehl aus:
python3 produce_embeddings.py --selfies_dataset=data/molecule_dataset_selfies.csv --model_file=data/pretrained_models/SELFormer --embed_file=data/embeddings.csv
Die Einbettungsdings, die durch unser am besten ausgebildeter Modell für Molekulenetdaten erzeugt werden, können hier direkt heruntergeladen werden.
Sie können diese Einbettungsdings auch mit dem folgenden Befehl neu generieren.
python3 get_moleculenet_embeddings.py --dataset_path=data/finetuning_datasets --model_file=data/pretrained_models/SELFormer
Um ein Modell vorzuverüben, führen Sie bitte den Befehl unten aus. Wenn Sie einen Selfies -Datensatz haben, können Sie ihn direkt verwenden, indem Sie den Pfad des Datensatzes zu -Selfies_Dataset geben. Wenn Sie einen lächelnden Datensatz haben, können Sie den Pfad des Datensatzes zu --Smiles_Dataset angeben und die Selfies -Darstellungen werden auf dem Pfad erstellt, der zu -Selfies_Dataset gegeben wurde.
python3 train_pretraining_model.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv --prepared_data_path=data/selfies_data.txt --bpe_path=data/BPETokenizer --roberta_fast_tokenizer_path=data/RobertaFastTokenizer --hyperparameters_path=data/pretraining_hyperparameters.yml --subset_size=100000
Sie können unten Befehle verwenden, um ein vorgebildetes Modell für verschiedene Vorhersageaufgaben für molekulare Eigenschaften zu optimieren. Diese Befehle werden verwendet, um Datensätze zu verarbeiten, die Smiles -Darstellungen von Molekülen enthalten. Smiles -Darstellungen sollten in einer Spalte mit einem Header namens "Smiles" gespeichert werden. Sie können die Beispieldatensätze im Verzeichnis data/finetuning_datasets sehen.
Binärklassifizierungsaufgaben
Um ein vorgebildetes Modell in einem binären Klassifizierungsdatensatz zu optimieren, führen Sie bitte den folgenden Befehl aus.
python3 train_classification_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/classification/bbbp/bbbp.csv --save_to=data/finetuned_models/SELFormer_bbbp_classification --target_column_id=1 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
Multi-Label-Klassifizierungsaufgaben
Um ein vorgebildetes Modell auf einem Multi-Label-Klassifizierungsdatensatz zu optimieren, führen Sie bitte den folgenden Befehl aus. Die RobertafastTokenizer-Dateien sollten im selben Verzeichnis wie das vorgebildete Modell gespeichert werden.
python3 train_classification_multilabel_model.py --model=data/saved_models/SELFormer --dataset=data/finetuning_datasets/classification/tox21/tox21.csv --save_to=data/finetuned_models/SELFormer_tox21_classification --use_scaffold=1 --batch_size=16 --num_epochs=25 --lr=5e-5 --wd=0
Regressionsaufgaben
Um ein vorgebildetes Modell in einem Regressionsdatensatz zu optimieren, führen Sie bitte den folgenden Befehl aus.
python3 train_regression_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/regression/esol/esol.csv --save_to=data/finetuned_models/SELFormer_esol_regression --target_column_id=-1 --scaler=2 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
Hier können feine Selbstormer-Modelle hier heruntergeladen werden. Um Vorhersagen mit diesen Modellen zu treffen, befolgen Sie bitte die folgenden Anweisungen.
Um Vorhersagen für BACE-, BBBP- und HIV -Datensätze zu treffen, führen Sie bitte den folgenden Befehl aus. Ändern Sie die angegebenen Argumente für verschiedene Aufgaben. Die Standardparameter laden ein feines abgestimmtes Modell auf BBBP.
python3 binary_class_pred.py --task=bbbp --model_name=data/finetuned_models/SELFormer_bbbp_scaffold_optimized --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/bbbp/bbbp_mock.csv --training_args=data/finetuned_models/SELFormer_bbbp_scaffold_optimized/training_args.bin
Um Vorhersagen für TOX21- und SIDER -Datensätze zu treffen, führen Sie bitte den folgenden Befehl aus. Ändern Sie die angegebenen Argumente für verschiedene Aufgaben. Die Standardparameter laden ein feines abgestimmtes Modell auf Sider.
python3 multilabel_class_pred.py --task=sider --model_name=data/finetuned_models/SELFormer_sider_scaffold_optimized --pred_set=data/finetuning_datasets/classification/sider/sider_mock.csv --training_args=data/finetuned_models/SELFormer_sider_scaffold_optimized/training_args.bin --num_labels=27
Um Vorhersagen für ESOL-, Freesolv-, Lipophilicity- und PDBBind -Datensätze zu treffen, führen Sie bitte den Befehl unten aus. Ändern Sie die angegebenen Argumente für verschiedene Aufgaben. Die Standardparameter laden ein feines abgestimmtes Modell auf ESOL.
python3 regression_pred.py --task=esol --model_name=data/finetuned_models/esol_regression --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/esol/esol_mock.csv --training_args=data/finetuned_models/esol_regression/training_args.bin
Copyright (C) 2023 Hubiodatalab
Dieses Programm ist kostenlose Software: Sie können es neu verteilt und/oder unter den Bestimmungen der GNU General Public Lizenz wie von der Free Software Foundation, entweder Version 3 der Lizenz veröffentlicht, oder (nach Ihrer Option) jede spätere Version ändern.
Dieses Programm wird in der Hoffnung verteilt, dass es nützlich sein wird, jedoch ohne Garantie; Ohne die implizite Garantie für Handelsfähigkeit oder Eignung für einen bestimmten Zweck. Weitere Informationen finden Sie in der GNU General Public Lizenz.
Sie hätten zusammen mit diesem Programm eine Kopie der GNU General Public Lizenz erhalten haben. Wenn nicht, siehe http://www.gnu.org/licenses/.