광대 한 화학 공간의 자동화 된 전산 분석은 약물 발견 및 재료 과학과 같은 수많은 연구 분야에 중요합니다. 표현 학습 기술은 최근 복잡한 데이터의 작고 유익한 수치 표현을 생성하는 주요 목표와 함께 사용되었습니다. 분자 표현을 효율적으로 학습하기위한 한 가지 방법은 NLP (Natural Language Processing) 알고리즘을 통해 화학 물질의 문자열 기반 표기법을 처리하는 것입니다. 지금까지 제안 된 대부분의 방법은이 목적을 위해 미소 표기법을 활용합니다. 그러나 Smiles는 유효성과 견고성과 관련된 수많은 문제와 관련이 있으며, 이는 모델이 데이터에 숨겨진 지식을 효과적으로 발견하지 못하게 할 수 있습니다. 이 연구에서 우리는 유연하고 고품질 분자 표현을 배우기 위해 100% 유효하고 작고 표현적인 표기법 인 셀카를 입력하는 변압기 아키텍처 기반 화학 언어 모델 인 Selformer를 제안합니다. Selformer는 2 백만 개의 약물 유사 화합물에서 미리 훈련되고 다양한 분자 특성 예측 작업을 위해 미세 조정됩니다. 우리의 성능 평가는 셀프 오르메르 (Selformer)가 그래프 학습 기반 접근법 및 미소 기반 화학 언어 모델을 포함한 모든 경쟁 방법을 분자 및 약물 반응의 수성 용해도 예측에 능숙하다는 것을 밝혀 냈습니다. 우리는 또한 차원 감소를 통해 셀프러머에 의해 학습 된 분자 표현을 시각화했으며, 이는 미리 훈련 된 모델조차도 다른 구조적 특성으로 분자를 구별 할 수 있음을 나타냅니다. 우리는 데이터 세트 및 미리 훈련 된 모델과 함께 프로그램 도구로서 Selformer를 공유했습니다. 전반적으로, 우리의 연구는 화학 언어 모델링의 맥락에서 셀카 표기법을 사용하는 이점을 보여주고 원하는 기능을 가진 새로운 약물 후보의 설계 및 발견을위한 새로운 가능성을 열어줍니다.
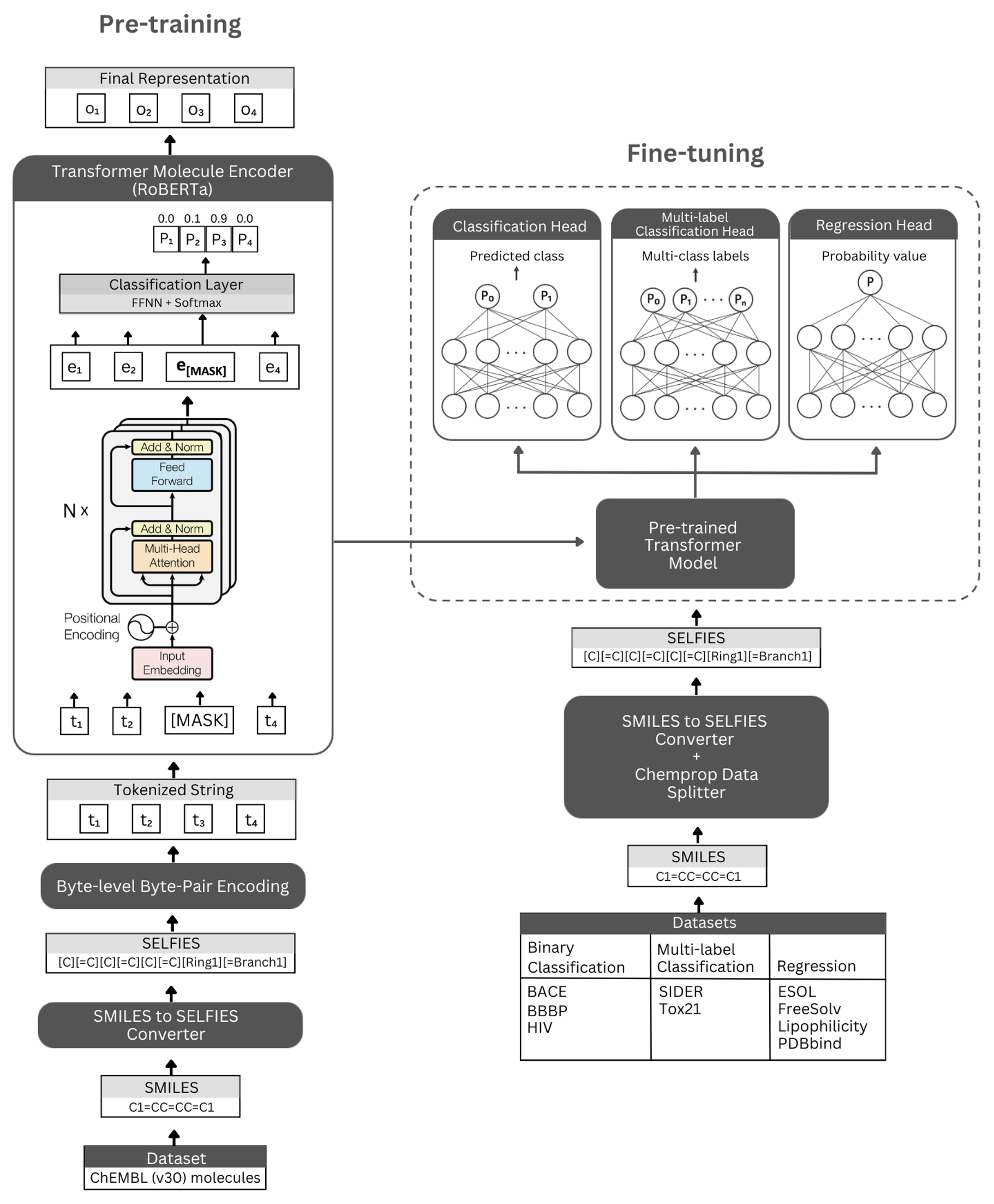
수치. Selformer Architecture의 개략도 및 실험. 왼쪽 : 자체 감독 된 사전 훈련은 셀카에 의해 인코딩 된 소분자의 간결하고 유익한 표현을 학습하기 위해 마스크 언어 모델링을 통해 변압기 인코더 모듈을 사용합니다. 오른쪽 : 미리 훈련 된 모델은 수많은 분자 특성 기반 분류 및 회귀 작업에서 독립적으로 미세 조정되었습니다.
Selformer는 Bert와 동일한 아키텍처를 사용하는 Roberta Transformer Architecture를 기반으로하지만 모델 성능을 향상 시키거나 다른 이점을 제공하는 것으로 밝혀진 특정 수정으로 사용됩니다. 이러한 수정 중 하나는 문자 수준 BPE 대신 토큰 화에 바이트 레벨 바이트 패밀 인코딩 (BPE)을 사용하는 것입니다. 또 다른 하나는 Roberta가 다음 문장 예측 (NSP) 작업을 무시하면서 MLM (Masked Language Modeling) 목표에 대해 독점적으로 미리 훈련된다는 것입니다. Selformer는 (i) 셀카에 의해 인코딩 된 소분자의 간결하고 유익한 표현을 학습하기 위해 변압기 인코더 모듈을 사용하는 (i) 자체 감독 된 미리 훈련 된 모델을 가지고 있으며, (ii) 수많은 분류 및 회귀 기반 분자 특성 예측 작업에서 사전 트리 된 모델을 기본 및 미세 튜닝으로 사용하는 감독 분류/회귀 모델을 감독합니다.
미리 훈련 된 인코더 모델은 "RobertamaskEdlm"으로 구현되고 미세 조정 모델은 "RobertaForeceenceClassification"으로 구현됩니다. 미세 조정 프로세스의 경우, 셀프 오르메르 아키텍처에는 미리 훈련 된 Roberta 모델이 기본으로, "RobertaclassificationHead"클래스는 다음 계층 (분류 및 회귀 용)을 포함합니다. "RobertaclassificationHead"클래스는 드롭 아웃 층, 조밀 한 층, TANH 활성화 기능, 드롭 아웃 층 및 최종 선형 층으로 구성됩니다. 미세 조정 프로세스 동안 미리 훈련 된 Roberta Base 모델의 서열 출력을 분류기로 전달합니다.
종속성을 설치하기위한 Conda 플랫폼을 적극 권장합니다. Conda를 설치 한 후 아래 정의 된대로 종속성을 가진 환경을 만들고 활성화하십시오.
conda create -n SELFormer_env
conda activate SELFormer_env
conda env update --file data/requirements.yml
사전 훈련 된 셀프 모머 모델은 여기에서 다운로드 할 수 있습니다. Best Performing 모델에 의해 생성되는 Chembl30 및 Chembl33의 모든 분자의 임베딩은 여기에서 제공됩니다.
미리 훈련 된 모델을 사용하여 자신의 데이터 세트에 대한 임베딩을 생성 할 수도 있습니다. 그렇게하려면 분자의 셀카 표기법이 필요합니다. 아래 명령을 사용하여 Smiles 데이터 세트에 대한 셀카 표기법을 생성 할 수 있습니다.
Chembl30 데이터 세트 의 임베딩을 생성하기위한 코드를 재현하려면 Molecule_dataset_smiles.zip 및/또는 molecule_dataset_selfies.zip 파일을 각각 입력 스마일 및 자체 데이터 세트로 사용할 수 있습니다.
python3 generate_selfies.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv
미리 훈련 된 모델을 사용하여 셀카 분자 데이터 세트에 대한 임베딩을 생성하려면 다음 명령을 실행하십시오.
python3 produce_embeddings.py --selfies_dataset=data/molecule_dataset_selfies.csv --model_file=data/pretrained_models/SELFormer --embed_file=data/embeddings.csv
분자 데이터에 대한 가장 잘 수행되는 미리 훈련 된 모델에 의해 생성 된 임베딩은 여기에서 직접 다운로드 할 수 있습니다.
아래 명령을 사용하여 이러한 임베딩을 다시 생성 할 수도 있습니다.
python3 get_moleculenet_embeddings.py --dataset_path=data/finetuning_datasets --model_file=data/pretrained_models/SELFormer
모델을 사전 훈련하려면 아래 명령을 실행하십시오. 셀카 데이터 세트가있는 경우 데이터 세트의 경로를 -selfies_dataset 에 제공하여 직접 사용할 수 있습니다. 미소 데이터 세트가있는 경우 데이터 세트의 경로를 -smiles_dataset 로 제공 할 수 있으며 셀카 표현은 -selfies_dataset 에 제공된 경로에서 생성됩니다.
python3 train_pretraining_model.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv --prepared_data_path=data/selfies_data.txt --bpe_path=data/BPETokenizer --roberta_fast_tokenizer_path=data/RobertaFastTokenizer --hyperparameters_path=data/pretraining_hyperparameters.yml --subset_size=100000
아래 명령을 사용하여 다양한 분자 특성 예측 작업에 대한 미리 훈련 된 모델을 미세 조정할 수 있습니다. 이 명령은 분자의 미소 표현을 포함하는 데이터 세트를 처리하는 데 사용됩니다. Smiles 표현은 "Smiles"라는 헤더가있는 열에 저장해야합니다. 데이터/FINETUNING_DATASETS 디렉토리에서 예제 데이터 세트를 볼 수 있습니다.
이진 분류 작업
이진 분류 데이터 세트에서 미리 훈련 된 모델을 미세 조정하려면 아래 명령을 실행하십시오.
python3 train_classification_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/classification/bbbp/bbbp.csv --save_to=data/finetuned_models/SELFormer_bbbp_classification --target_column_id=1 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
멀티 라벨 분류 작업
멀티 라벨 분류 데이터 세트에서 미리 훈련 된 모델을 미세 조정하려면 아래 명령을 실행하십시오. RobertaFastTokenizer 파일은 미리 훈련 된 모델과 동일한 디렉토리에 저장해야합니다.
python3 train_classification_multilabel_model.py --model=data/saved_models/SELFormer --dataset=data/finetuning_datasets/classification/tox21/tox21.csv --save_to=data/finetuned_models/SELFormer_tox21_classification --use_scaffold=1 --batch_size=16 --num_epochs=25 --lr=5e-5 --wd=0
회귀 작업
회귀 데이터 세트에서 미리 훈련 된 모델을 미세 조정하려면 아래 명령을 실행하십시오.
python3 train_regression_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/regression/esol/esol.csv --save_to=data/finetuned_models/SELFormer_esol_regression --target_column_id=-1 --scaler=2 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
미세 조정 된 Selformer 모델은 여기에서 다운로드 할 수 있습니다. 이 모델로 예측하려면 아래 지침을 따르십시오.
BACE, BBBP 및 HIV 데이터 세트에 대해 예측하려면 아래 명령을 실행하십시오. 다른 작업에 대한 표시된 인수를 변경하십시오. 기본 매개 변수는 BBBP에 미세 조정 모델을로드합니다.
python3 binary_class_pred.py --task=bbbp --model_name=data/finetuned_models/SELFormer_bbbp_scaffold_optimized --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/bbbp/bbbp_mock.csv --training_args=data/finetuned_models/SELFormer_bbbp_scaffold_optimized/training_args.bin
Tox21 및 Sider 데이터 세트에 대한 예측을하려면 아래 명령을 실행하십시오. 다른 작업에 대한 표시된 인수를 변경하십시오. 기본 매개 변수는 Sider에 미세 조정 모델을로드합니다.
python3 multilabel_class_pred.py --task=sider --model_name=data/finetuned_models/SELFormer_sider_scaffold_optimized --pred_set=data/finetuning_datasets/classification/sider/sider_mock.csv --training_args=data/finetuned_models/SELFormer_sider_scaffold_optimized/training_args.bin --num_labels=27
ESOL, FREESOLV, 친 유성 및 PDBBind 데이터 세트에 대한 예측을하려면 아래 명령을 실행하십시오. 다른 작업에 대한 표시된 인수를 변경하십시오. 기본 매개 변수는 ESOL에 미세 조정 모델을로드합니다.
python3 regression_pred.py --task=esol --model_name=data/finetuned_models/esol_regression --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/esol/esol_mock.csv --training_args=data/finetuned_models/esol_regression/training_args.bin
저작권 (C) 2023 Hubiodatalab
이 프로그램은 무료 소프트웨어입니다. Free Software Foundation, 라이센스의 버전 3 또는 이후 버전에서 게시 한 GNU 일반 공개 라이센스의 조건에 따라 재분배 및/또는 수정할 수 있습니다.
이 프로그램은 유용 할 것이지만 보증이 없다는 희망으로 배포됩니다. 상업성 또는 특정 목적에 대한 적합성에 대한 묵시적 보증조차 없습니다. 자세한 내용은 GNU 일반 공개 라이센스를 참조하십시오.
이 프로그램과 함께 GNU 일반 공개 라이센스 사본을 받았어야합니다. 그렇지 않은 경우 http://www.gnu.org/licenses/를 참조하십시오.