Автоматизированный вычислительный анализ обширного химического пространства имеет решающее значение для многочисленных областей исследований, таких как обнаружение лекарств и материальная наука. Методы обучения представительства недавно использовались с основной целью генерации компактных и информативных численных выражений сложных данных. Одним из подходов к эффективному изучению молекулярных представлений является обработка обозначений на основе строк химических веществ с помощью алгоритмов обработки естественного языка (NLP). Большинство методов, предложенных до сих пор, используют улыбки для этой цели; Тем не менее, улыбки связаны с многочисленными проблемами, связанными с достоверностью и надежностью, которые могут помешать модели эффективно раскрыть знания, скрытые в данных. В этом исследовании мы предлагаем Selformer, модель химического языка на основе архитектуры трансформатора, которая использует 100% достоверную, компактную и выразительную нотацию, селфи, в качестве входных данных, для изучения гибких и высококачественных молекулярных представлений. Selformer предварительно обучает двух миллионов лекарственных соединений и тонко настроен для различных задач прогнозирования молекулярного свойства. Наша оценка эффективности показала, что Selformer превосходит все конкурирующие методы, включая подходы, основанные на графиках и модели химического языка на основе улыбок, о прогнозировании растворимости водных молекул и побочных реакций лекарств. Мы также визуализировали молекулярные представления, изученные Selformer, посредством уменьшения размерности, что указывало на то, что даже предварительно обученная модель может различать молекулы с различными структурными свойствами. Мы поделились Selformer как программным инструментом вместе с его наборами данных и предварительно обученными моделями. В целом, наше исследование демонстрирует преимущество использования нотаций селфи в контексте моделирования химического языка и открывает новые возможности для проектирования и обнаружения новых кандидатов на наркотики с желаемыми особенностями.
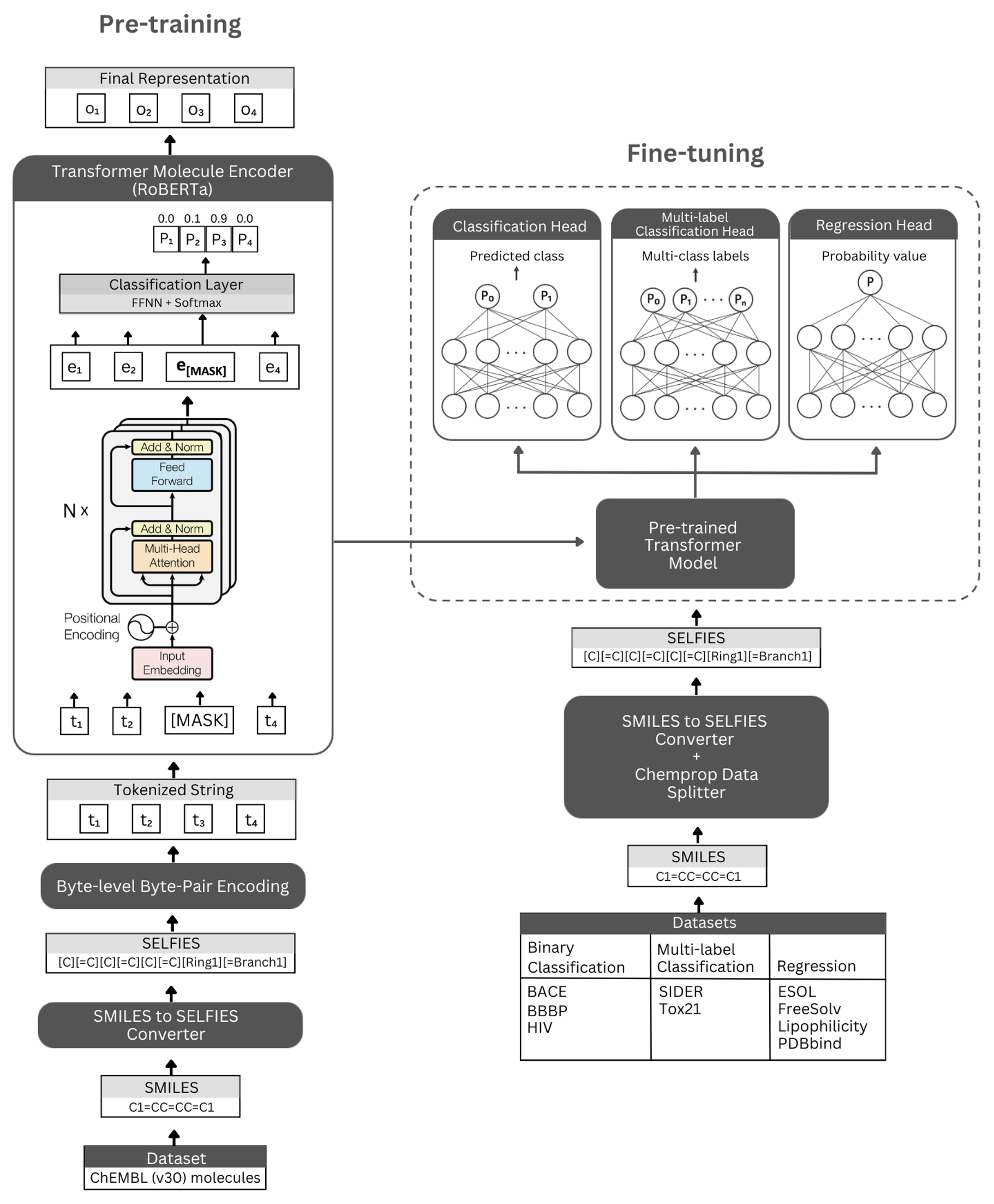
Фигура. Схематическое представление архитектуры самостоятельно и проведенных экспериментов. Слева: самоотверженное предварительное обучение использует модуль энкодера трансформатора посредством моделирования в масках для изучения кратких и информативных представлений о мелких молекулах, кодируемых их нотацией селфи. Справа: предварительно обученная модель была точно настроена на многочисленные задачи классификации и регрессии на основе молекулярных свойств.
Selformer построен на архитектуре Transformer Roberta, которая использует ту же архитектуру, что и BERT, но с определенными модификациями, которые, как было установлено, улучшают производительность модели или обеспечивают другие преимущества. Одной из таких модификаций является использование кодирования байтовых паев на уровне байтов (BPE) для токенизации вместо BPE уровня символов. Другим является то, что Роберта предварительно обучена исключительно по объективам маскированного языкового моделирования (MLM), игнорируя задачу предсказания следующего предложения (NSP). Selformer имеет (i) самоотверженные предварительно обученные модели, которые используют модуль энкодера трансформатора для обучения кратким и информативным представлениям мелких молекул, кодируемых их нотацией селфи, и (ii) контролируемые классификационные/регрессионные модели, которые используют предварительную модель в качестве базовой и тонкой настройки на многочисленные молекулярные свойства на основе классификации и регрессии.
Наши предварительно обученные модели энкодера реализованы как «Robertamaskedlm» и модели с тонкой настройкой как «robertaforsequenceclassication». Для процесса тонкой настройки архитектура Selformer включает в себя предварительно обученную модель Роберты в качестве своей базы, а также класс «robertaclassificationhead» в качестве следующих слоев (для классификации и регрессии). Класс "RobertaclassificationHead" состоит из выпадающего слоя, плотного слоя, функции активации TANH, выпадающего слоя и окончательного линейного слоя. Мы пересылаем выход последовательности предварительно обученной базовой модели Роберты в классификатор во время процесса тонкой настройки.
Мы настоятельно рекомендуем платформу Conda для установки зависимостей. После установки Conda, пожалуйста, создайте и активируйте среду с зависимостями, как определено ниже:
conda create -n SELFormer_env
conda activate SELFormer_env
conda env update --file data/requirements.yml
Предварительно обученные модели Selformer доступны для загрузки здесь. Внедрение всех молекул от Chembl30 и Chembl33, которые генерируются нашей лучшей моделью, доступны здесь.
Вы также можете генерировать Entgeddings для вашего собственного набора данных, используя предварительно обученные модели. Для этого вам понадобятся селфи обозначения ваших молекул. Вы можете использовать команду ниже, чтобы создать селфи обозначения для вашего набора данных Smiles.
Если вы хотите воспроизвести наш код для генерации встроенных набора данных ChemBL30, вы можете UNZIP MOLECULE_DATASET_SMILES.ZIP и/или MOLECULE_DATASET_SESWERES.ZIP в каталоге данных и используйте их в качестве входных наборов и наборов селени соответственно.
python3 generate_selfies.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv
Чтобы сгенерировать вставки для набора данных молекул селфи с использованием предварительно обученной модели, пожалуйста, запустите следующую команду:
python3 produce_embeddings.py --selfies_dataset=data/molecule_dataset_selfies.csv --model_file=data/pretrained_models/SELFormer --embed_file=data/embeddings.csv
Внедрения, сгенерированные нашей наиболее эффективной предварительно обученной моделью для данных Moleculenet, могут быть непосредственно загружены здесь.
Вы также можете переиграть эти встроения, используя команду ниже.
python3 get_moleculenet_embeddings.py --dataset_path=data/finetuning_datasets --model_file=data/pretrained_models/SELFormer
Чтобы предварительно готовить модель, пожалуйста, запустите команду ниже. Если у вас есть набор данных селфи, вы можете использовать его непосредственно, предоставив путь набора данных -selfies_dataset . Если у вас есть набор данных Smiles, вы можете дать путь набора данных -SMILES_DATASET , и представления селфи будут созданы на пути, данном -SELFIES_DATASET .
python3 train_pretraining_model.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv --prepared_data_path=data/selfies_data.txt --bpe_path=data/BPETokenizer --roberta_fast_tokenizer_path=data/RobertaFastTokenizer --hyperparameters_path=data/pretraining_hyperparameters.yml --subset_size=100000
Вы можете использовать команды ниже, чтобы точно настроить предварительно обученную модель для различных задач прогнозирования молекулярного свойства. Эти команды используются для обработки наборов данных, содержащих улыбки молекул. Представления улыбков должны храниться в столбце с заголовком под названием «Улыбки». Вы можете увидеть пример наборов данных в каталоге Data/Fenetuning_Datasets .
Бинарные классификационные задачи
Чтобы точно настроить предварительно обученную модель в бинарном наборе данных классификации, пожалуйста, запустите команду ниже.
python3 train_classification_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/classification/bbbp/bbbp.csv --save_to=data/finetuned_models/SELFormer_bbbp_classification --target_column_id=1 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
Задачи классификации с несколькими маршрутами
Чтобы точно настроить предварительно обученную модель в многопользовательском наборе данных классификации, пожалуйста, запустите команду ниже. Файлы RobertafastTokenizer должны храниться в том же каталоге, что и предварительно обученная модель.
python3 train_classification_multilabel_model.py --model=data/saved_models/SELFormer --dataset=data/finetuning_datasets/classification/tox21/tox21.csv --save_to=data/finetuned_models/SELFormer_tox21_classification --use_scaffold=1 --batch_size=16 --num_epochs=25 --lr=5e-5 --wd=0
Регрессионные задачи
Чтобы точно настроить предварительно обученную модель в наборе данных регрессии, пожалуйста, запустите команду ниже.
python3 train_regression_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/regression/esol/esol.csv --save_to=data/finetuned_models/SELFormer_esol_regression --target_column_id=-1 --scaler=2 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
Модели Selformer Suplemermer доступны для загрузки здесь. Чтобы сделать прогнозы с этими моделями, пожалуйста, следуйте инструкциям ниже.
Чтобы сделать прогнозы для наборов данных BACE, BBBP и ВИЧ, запустите команду ниже. Измените указанные аргументы для различных задач. Параметры по умолчанию будут загружать тонкую модель на BBBP.
python3 binary_class_pred.py --task=bbbp --model_name=data/finetuned_models/SELFormer_bbbp_scaffold_optimized --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/bbbp/bbbp_mock.csv --training_args=data/finetuned_models/SELFormer_bbbp_scaffold_optimized/training_args.bin
Чтобы сделать прогнозы для наборов данных TOX21 и Sider, запустите команду ниже. Измените указанные аргументы для различных задач. Параметры по умолчанию загружат тонкую модель на Sider.
python3 multilabel_class_pred.py --task=sider --model_name=data/finetuned_models/SELFormer_sider_scaffold_optimized --pred_set=data/finetuning_datasets/classification/sider/sider_mock.csv --training_args=data/finetuned_models/SELFormer_sider_scaffold_optimized/training_args.bin --num_labels=27
Чтобы сделать прогнозы для наборов данных ESOL, Freesolv, липофильности и PDBBind, запустите команду ниже. Измените указанные аргументы для различных задач. Параметры по умолчанию будут загружать тонкую модель на ESOL.
python3 regression_pred.py --task=esol --model_name=data/finetuned_models/esol_regression --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/esol/esol_mock.csv --training_args=data/finetuned_models/esol_regression/training_args.bin
Авторские права (C) 2023 Hubiodatalab
Эта программа является бесплатным программным обеспечением: вы можете перераспределить его и/или изменить ее в соответствии с условиями общей публичной лицензии GNU, опубликованных Фондом Free Software, либо версией 3 лицензии, либо (по варианту) любой более поздней версии.
Эта программа распространяется в надежде, что она будет полезна, но без каких -либо гарантий; даже без подразумеваемой гарантии торговой точки зрения или пригодности для определенной цели. Смотрите общую публичную лицензию GNU для получения более подробной информации.
Вы должны были получить копию общей публичной лицензии GNU вместе с этой программой. Если нет, см. Http://www.gnu.org/licenses/.