A análise computacional automatizada do vasto espaço químico é fundamental para vários campos de pesquisa, como descoberta de medicamentos e ciência do material. As técnicas de aprendizado de representação foram recentemente empregadas com o objetivo principal de gerar expressões numéricas compactas e informativas de dados complexos. Uma abordagem para aprender com eficiência representações moleculares é o processamento de notações baseadas em cordas de produtos químicos por meio de algoritmos de processamento de linguagem natural (PNL). A maioria dos métodos propostos até agora utiliza notações de sorrisos para esse fim; No entanto, o Smiles está associado a vários problemas relacionados à validade e robustez, o que pode impedir que o modelo descubra efetivamente o conhecimento oculto nos dados. Neste estudo, propomos Selformer, um modelo de linguagem química baseada em arquitetura de transformadores que utiliza uma notação 100% válida, compacta e expressiva, selfies, como entrada, a fim de aprender representações moleculares flexíveis e de alta qualidade. O Selformer é pré-treinado em dois milhões de compostos semelhantes a drogas e ajustado para diversas tarefas de previsão de propriedades moleculares. Nossa avaliação de desempenho revelou que, o Selformer supera todos os métodos concorrentes, incluindo abordagens baseadas em aprendizado de gráfico e modelos de linguagem química baseados em sorrisos, na previsão da solubilidade aquosa de moléculas e reações adversas a medicamentos. Também visualizamos representações moleculares aprendidas pelo auto-formador por redução da dimensionalidade, que indicaram que mesmo o modelo pré-treinado pode discriminar moléculas com diferentes propriedades estruturais. Compartilhamos o Selformer como uma ferramenta programática, juntamente com seus conjuntos de dados e modelos pré-treinados. No geral, nossa pesquisa demonstra o benefício de usar as anotações de selfies no contexto da modelagem de linguagem química e abre novas possibilidades para o design e a descoberta de novos candidatos a medicamentos com os recursos desejados.
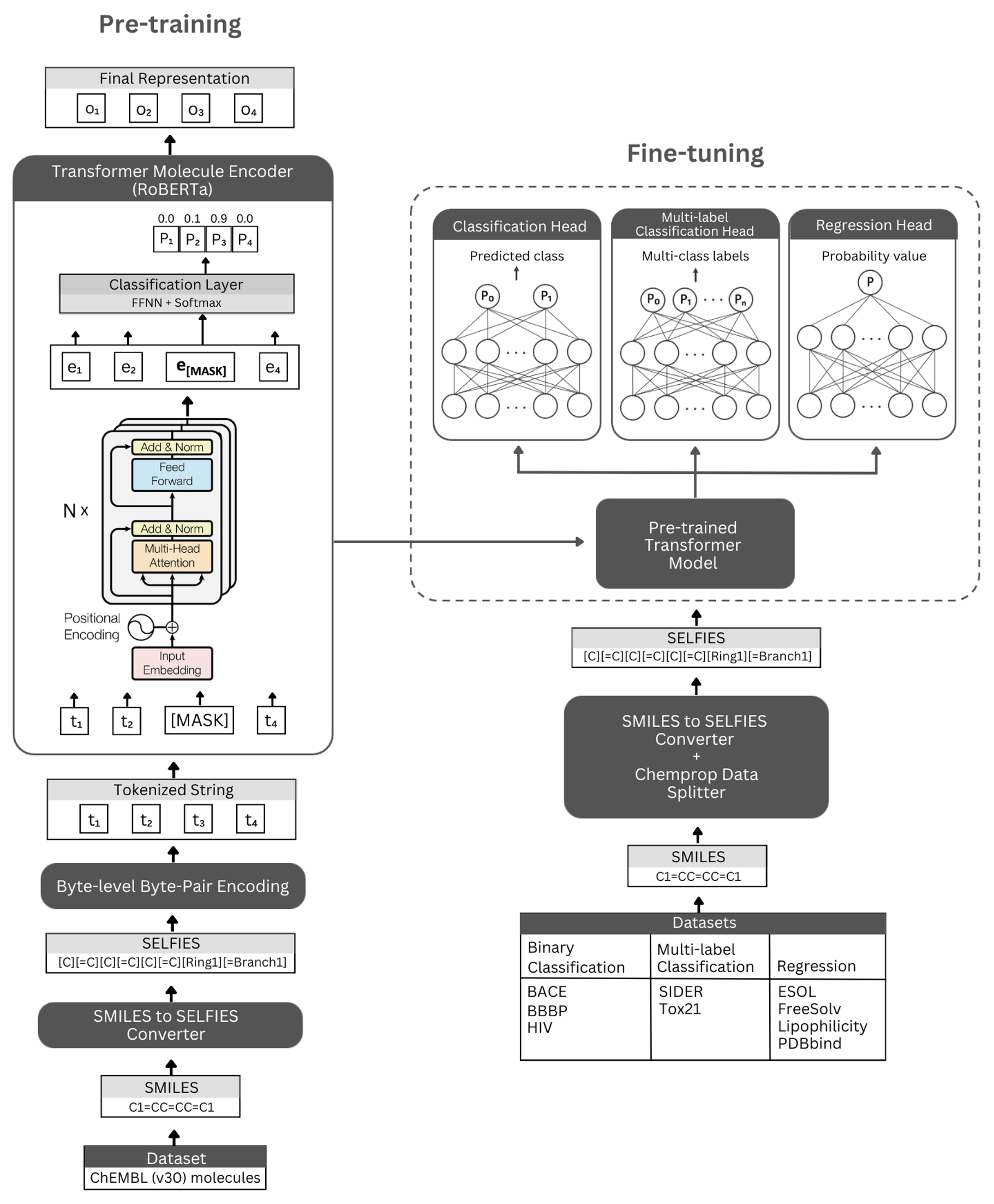
Figura. A representação esquemática da arquitetura autônoma e os experimentos realizados. Esquerda: o pré-treinamento auto-supervisionado utiliza o módulo do codificador do transformador via modelagem de linguagem mascarada para aprender representações concisas e informativas de pequenas moléculas codificadas por suas selfies notação. Direita: O modelo pré-treinado foi ajustado de forma independente em inúmeras tarefas de classificação e regressão moleculares baseadas em propriedades.
O Selformer é construído sobre a arquitetura Roberta Transformer, que utiliza a mesma arquitetura que Bert, mas com certas modificações que foram encontradas para melhorar o desempenho do modelo ou fornecer outros benefícios. Uma dessas modificações é o uso da codificação de par-pares de bytes (BPE) para tokenização em vez de BPE no nível do caractere. Outro é que, Roberta é pré-treinado exclusivamente com o objetivo de modelagem de idiomas mascarado (MLM), e desconsiderando a próxima tarefa de previsão de frases (NSP). O Selformer possui (i) modelos pré-treinados auto-supervisionados que utilizam o módulo do codificador do transformador para aprender representações concisas e informativas de pequenas moléculas codificadas por suas selfies notação e (ii) modelos de classificação/regressão supervisionados que usam o modelo pré-treinado como base e tune fino em numerosas classificação e regressão baseadas em regressão baseadas em regressão.
Nossos modelos de codificadores pré-treinados são implementados como "Robertamaskedlm" e modelos de ajuste fino como "RobertforeNencEnceclassification". Para o processo de ajuste fino, a arquitetura Selformer inclui o modelo Roberta pré-treinado como sua base, e a classe "Robertaclassificationhead" como as seguintes camadas (para classificação e regressão). A classe "Robertaclassificationhead" consiste em uma camada de abandono, uma camada densa, função de ativação de Tanh, uma camada de abandono e uma camada linear final. Encaminhamos a saída de sequência do modelo base Roberta pré-treinado para o classificador durante o processo de ajuste fino.
Recomendamos altamente a plataforma do CONDA para instalar dependências. Após a instalação do CONDA, crie e ative um ambiente com dependências, conforme definido abaixo:
conda create -n SELFormer_env
conda activate SELFormer_env
conda env update --file data/requirements.yml
Os modelos de auto-atuação pré-treinados estão disponíveis para download aqui. As incorporações de todas as moléculas do ChemBl30 e ChemBl33 que são geradas pelo nosso modelo de melhor desempenho estão disponíveis aqui.
Você também pode gerar incorporação para seu próprio conjunto de dados usando os modelos pré-treinados. Para fazer isso, você precisará de anotações selfies de suas moléculas. Você pode usar o comando abaixo para gerar notações selfies para o seu conjunto de dados Smiles.
Se você deseja reproduzir nosso código para gerar incorporações do conjunto de dados ChemBl30, poderá descompactar molecule_dataset_smiles.zip e/ou molecule_dataset_elfies.zip arquivos no diretório de dados e os use como sorrisos de entrada e dados de slowies, respectivamente.
python3 generate_selfies.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv
Para gerar incorporações para o conjunto de dados da molécula de selfies usando um modelo pré-treinado, execute o seguinte comando:
python3 produce_embeddings.py --selfies_dataset=data/molecule_dataset_selfies.csv --model_file=data/pretrained_models/SELFormer --embed_file=data/embeddings.csv
As incorporações geradas pelo nosso modelo pré-treinado com melhor desempenho para dados de moleculenet podem ser baixadas diretamente aqui.
Você também pode gerar novamente essas incorporações usando o comando abaixo.
python3 get_moleculenet_embeddings.py --dataset_path=data/finetuning_datasets --model_file=data/pretrained_models/SELFormer
Para pré-treinar um modelo, execute o comando abaixo. Se você possui um conjunto de dados Selfies, poderá usá -lo diretamente, fornecendo o caminho do conjunto de dados para - -Selfies_dataset . Se você tiver um conjunto de dados Smiles, poderá fornecer o caminho do conjunto de dados para - -Smiles_dataset e as representações de selfies serão criadas no caminho dado a - -Selfies_dataset .
python3 train_pretraining_model.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv --prepared_data_path=data/selfies_data.txt --bpe_path=data/BPETokenizer --roberta_fast_tokenizer_path=data/RobertaFastTokenizer --hyperparameters_path=data/pretraining_hyperparameters.yml --subset_size=100000
Você pode usar os comandos abaixo para ajustar um modelo pré-treinado para várias tarefas de previsão de propriedades moleculares. Esses comandos são utilizados para lidar com conjuntos de dados contendo representações de moléculas de sorrisos. As representações de sorrisos devem ser armazenadas em uma coluna com um cabeçalho chamado "Smiles". Você pode ver os conjuntos de dados de exemplo no diretório Data/Finetuning_Datasets .
Tarefas de classificação binária
Para ajustar um modelo pré-treinado em um conjunto de dados de classificação binária, execute o comando abaixo.
python3 train_classification_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/classification/bbbp/bbbp.csv --save_to=data/finetuned_models/SELFormer_bbbp_classification --target_column_id=1 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
Tarefas de classificação com vários rótulos
Para ajustar um modelo pré-treinado em um conjunto de dados de classificação com vários rótulos, execute o comando abaixo. Os arquivos RobertAfastTokenizer devem ser armazenados no mesmo diretório que o modelo pré-treinado.
python3 train_classification_multilabel_model.py --model=data/saved_models/SELFormer --dataset=data/finetuning_datasets/classification/tox21/tox21.csv --save_to=data/finetuned_models/SELFormer_tox21_classification --use_scaffold=1 --batch_size=16 --num_epochs=25 --lr=5e-5 --wd=0
Tarefas de regressão
Para ajustar um modelo pré-treinado em um conjunto de dados de regressão, execute o comando abaixo.
python3 train_regression_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/regression/esol/esol.csv --save_to=data/finetuned_models/SELFormer_esol_regression --target_column_id=-1 --scaler=2 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
Modelos de auto-atualização de ajuste fino estão disponíveis para download aqui. Para fazer previsões com esses modelos, siga as instruções abaixo.
Para fazer previsões para conjuntos de dados BACE, BBBP e HIV, execute o comando abaixo. Altere os argumentos indicados para diferentes tarefas. Os parâmetros padrão carregarão o modelo de ajuste fino no BBBP.
python3 binary_class_pred.py --task=bbbp --model_name=data/finetuned_models/SELFormer_bbbp_scaffold_optimized --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/bbbp/bbbp_mock.csv --training_args=data/finetuned_models/SELFormer_bbbp_scaffold_optimized/training_args.bin
Para fazer previsões para os conjuntos de dados Tox21 e Sider, execute o comando abaixo. Altere os argumentos indicados para diferentes tarefas. Os parâmetros padrão carregarão o modelo de ajuste fino no sider.
python3 multilabel_class_pred.py --task=sider --model_name=data/finetuned_models/SELFormer_sider_scaffold_optimized --pred_set=data/finetuning_datasets/classification/sider/sider_mock.csv --training_args=data/finetuned_models/SELFormer_sider_scaffold_optimized/training_args.bin --num_labels=27
Para fazer previsões para os conjuntos de dados de ESOL, Freesolv, Lipofilicidade e PDBBind, execute o comando abaixo. Altere os argumentos indicados para diferentes tarefas. Os parâmetros padrão carregarão o modelo de ajuste fino na ESOL.
python3 regression_pred.py --task=esol --model_name=data/finetuned_models/esol_regression --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/esol/esol_mock.csv --training_args=data/finetuned_models/esol_regression/training_args.bin
Copyright (C) 2023 Hubiodatalab
Este programa é um software livre: você pode redistribuí -lo e/ou modificá -lo nos termos da licença pública geral da GNU, conforme publicado pela Free Software Foundation, versão 3 da licença ou (por sua opção) qualquer versão posterior.
Este programa é distribuído na esperança de que seja útil, mas sem garantia; sem a garantia implícita de comercialização ou aptidão para uma finalidade específica. Veja a licença pública geral da GNU para obter mais detalhes.
Você deveria ter recebido uma cópia da licença pública geral da GNU junto com este programa. Caso contrário, consulte http://www.gnu.org/license/.