การวิเคราะห์การคำนวณอัตโนมัติของพื้นที่เคมีขนาดใหญ่เป็นสิ่งสำคัญสำหรับการวิจัยจำนวนมากเช่นการค้นพบยาและวิทยาศาสตร์วัสดุ เทคนิคการเรียนรู้การเป็นตัวแทนเพิ่งได้รับการใช้กับวัตถุประสงค์หลักของการสร้างการแสดงออกเชิงตัวเลขขนาดกะทัดรัดและให้ข้อมูลของข้อมูลที่ซับซ้อน วิธีหนึ่งในการเรียนรู้การเป็นตัวแทนระดับโมเลกุลได้อย่างมีประสิทธิภาพคือการประมวลผลสัญลักษณ์ของสารเคมีผ่านอัลกอริทึมการประมวลผลภาษาธรรมชาติ (NLP) วิธีการส่วนใหญ่ที่เสนอจนถึงตอนนี้ใช้สัญลักษณ์รอยยิ้มเพื่อจุดประสงค์นี้ อย่างไรก็ตามรอยยิ้มนั้นเกี่ยวข้องกับปัญหามากมายที่เกี่ยวข้องกับความถูกต้องและความทนทานซึ่งอาจป้องกันไม่ให้แบบจำลองการเปิดเผยความรู้ที่ซ่อนอยู่ในข้อมูลได้อย่างมีประสิทธิภาพ ในการศึกษานี้เราเสนอ Selformer ซึ่งเป็นแบบจำลองภาษาเคมีที่ใช้สถาปัตยกรรมของหม้อแปลงที่ใช้สัญลักษณ์ที่ถูกต้องขนาดกะทัดรัดและแสดงออกถึง 100% เซลฟี่เป็นอินพุตเพื่อเรียนรู้การเป็นตัวแทนโมเลกุลที่ยืดหยุ่นและคุณภาพสูง Selformer ได้รับการฝึกอบรมล่วงหน้าเกี่ยวกับสารประกอบที่มีลักษณะคล้ายยาสองล้านตัว การประเมินผลการปฏิบัติงานของเราได้เปิดเผยว่า Selformer มีประสิทธิภาพสูงกว่าวิธีการแข่งขันทั้งหมดรวมถึงวิธีการเรียนรู้แบบกราฟและแบบจำลองภาษาเคมีที่อิงกับรอยยิ้มในการทำนายความสามารถในการละลายน้ำของโมเลกุลและปฏิกิริยาของยาที่ไม่พึงประสงค์ นอกจากนี้เรายังแสดงให้เห็นถึงการเป็นตัวแทนของโมเลกุลที่เรียนรู้โดย Selformer ผ่านการลดขนาดมิติซึ่งแสดงให้เห็นว่าแม้แต่แบบจำลองที่ผ่านการฝึกอบรมมาก่อนสามารถแยกแยะโมเลกุลที่มีคุณสมบัติโครงสร้างที่แตกต่างกัน เราแบ่งปัน Selformer เป็นเครื่องมือเชิงโปรแกรมพร้อมกับชุดข้อมูลและรุ่นที่ผ่านการฝึกอบรมมาก่อน โดยรวมการวิจัยของเราแสดงให้เห็นถึงประโยชน์ของการใช้สัญลักษณ์เซลฟี่ในบริบทของการสร้างแบบจำลองภาษาเคมีและเปิดโอกาสใหม่สำหรับการออกแบบและการค้นพบผู้สมัครยาใหม่ที่มีคุณสมบัติที่ต้องการ
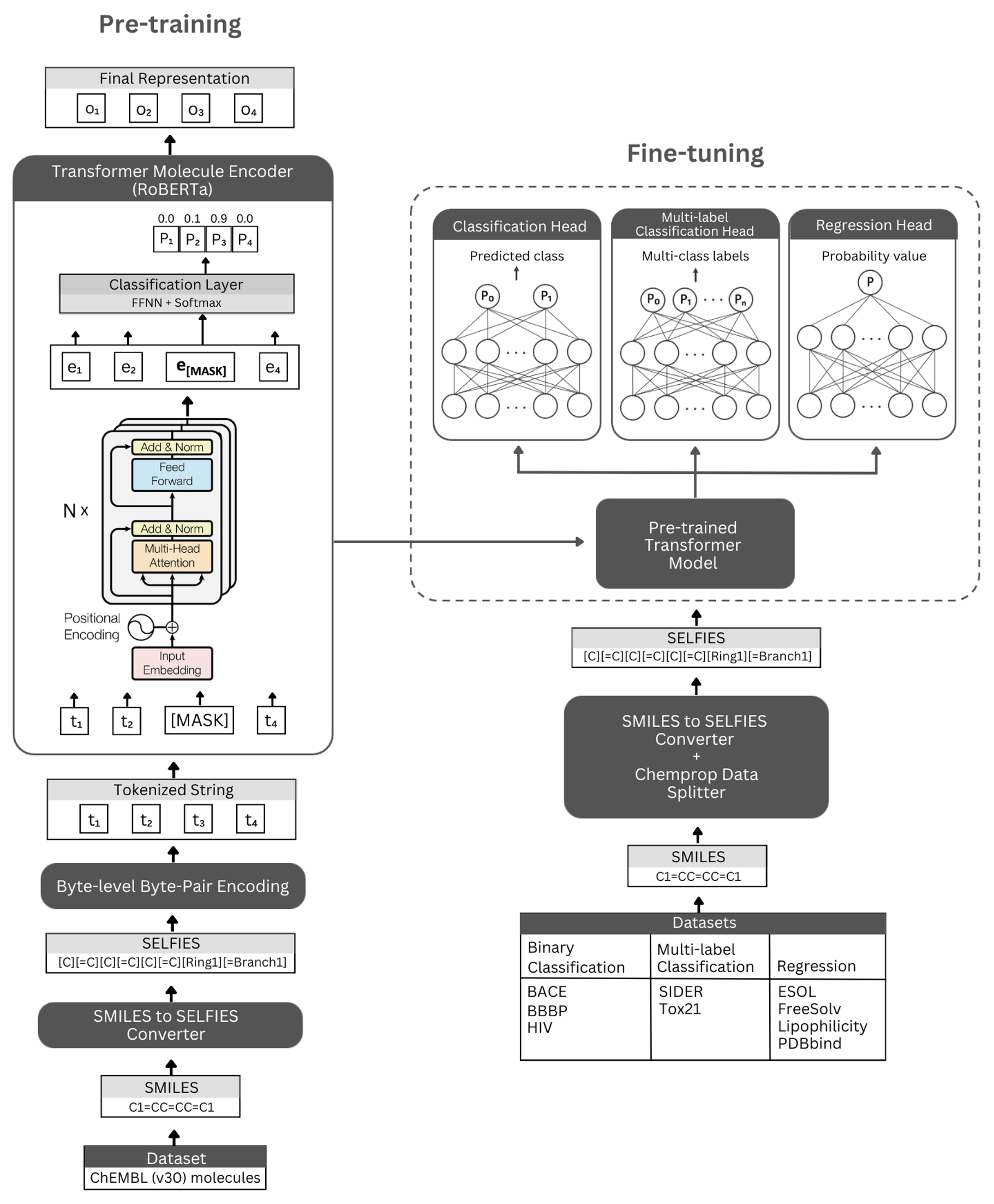
รูป. การแสดงแผนผังของสถาปัตยกรรม Selformer และการทดลองที่ดำเนินการ ซ้าย: การฝึกอบรมก่อนการดูแลตนเองใช้โมดูลตัวเข้ารหัสหม้อแปลงผ่านการสร้างแบบจำลองภาษาที่สวมหน้ากากเพื่อการเรียนรู้ที่กระชับและเป็นตัวแทนของโมเลกุลขนาดเล็กที่เข้ารหัสโดยสัญกรณ์เซลฟี่ของพวกเขา ขวา: โมเดลที่ผ่านการฝึกอบรมมาก่อนได้รับการปรับแต่งอย่างดีอย่างอิสระในการจำแนกประเภทของโมเลกุลตามอณูและงานการถดถอย
Selformer ถูกสร้างขึ้นบนสถาปัตยกรรม Roberta Transformer ซึ่งใช้สถาปัตยกรรมเช่นเดียวกับ Bert แต่ด้วยการดัดแปลงบางอย่างที่พบเพื่อปรับปรุงประสิทธิภาพของโมเดลหรือให้ประโยชน์อื่น ๆ การดัดแปลงอย่างหนึ่งคือการใช้การเข้ารหัสไบต์ระดับไบต์ (BPE) สำหรับโทเค็นแทนระดับ BPE ระดับอักขระ อีกประการหนึ่งคือโรเบอร์ต้าได้รับการฝึกอบรมล่วงหน้าโดยเฉพาะในวัตถุประสงค์การสร้างแบบจำลองภาษาที่สวมหน้ากาก (MLM) ในขณะที่ไม่สนใจงานการทำนายประโยคถัดไป (NSP) Selformer มี (i) โมเดลที่ได้รับการฝึกฝนมาก่อนที่ได้รับการฝึกฝนตนเองซึ่งใช้โมดูลตัวเข้ารหัสหม้อแปลงสำหรับการเรียนรู้การเป็นตัวแทนที่กระชับและให้ข้อมูลของโมเลกุลขนาดเล็กที่เข้ารหัสโดยสัญกรณ์เซลฟี่ของพวกเขาและ (ii) แบบจำลองการจำแนก/การถดถอยแบบควบคุมการถดถอย
โมเดลเข้ารหัสที่ผ่านการฝึกอบรมมาก่อนของเราถูกนำมาใช้เป็น "Robertamaskedlm" และโมเดลการปรับแต่งอย่างละเอียดว่า "RobertaforenceCecleScification" สำหรับกระบวนการปรับแต่งอย่างละเอียดสถาปัตยกรรม Selformer รวมโมเดล Roberta ที่ผ่านการฝึกอบรมมาล่วงหน้าเป็นฐานและคลาส "Robertaclassification Head" เป็นเลเยอร์ต่อไปนี้ (สำหรับการจำแนกและการถดถอย) คลาส "Robertaclassificationhead" ประกอบด้วยชั้นออกกลางคัน, ชั้นหนาแน่น, ฟังก์ชั่นการเปิดใช้งาน TANH, ชั้นการออกกลางคันและชั้นเชิงเส้นสุดท้าย เราส่งต่อลำดับผลลัพธ์ของโมเดลฐานโรเบอร์ต้าที่ผ่านการฝึกอบรมมาก่อนไปยังตัวจําแนกในระหว่างกระบวนการปรับจูน
เราขอแนะนำแพลตฟอร์ม Conda สำหรับการติดตั้งการพึ่งพา หลังจากการติดตั้ง conda โปรดสร้างและเปิดใช้งานสภาพแวดล้อมที่มีการอ้างอิงตามที่กำหนดไว้ด้านล่าง:
conda create -n SELFormer_env
conda activate SELFormer_env
conda env update --file data/requirements.yml
รุ่น Selformer ที่ผ่านการฝึกอบรมมาก่อนมีให้ดาวน์โหลดที่นี่ การฝังตัวของโมเลกุลทั้งหมดจาก ChemBl30 และ ChemBL33 ที่สร้างขึ้นโดยแบบจำลองที่มีประสิทธิภาพดีที่สุดของเรามีให้ที่นี่
นอกจากนี้คุณยังสามารถสร้าง EMBEDDINGS สำหรับชุดข้อมูลของคุณเองโดยใช้โมเดลที่ผ่านการฝึกอบรมมาก่อน ในการทำเช่นนั้นคุณจะต้องมีสัญลักษณ์เซลฟี่ของโมเลกุลของคุณ คุณสามารถใช้คำสั่งด้านล่างเพื่อสร้างสัญลักษณ์เซลฟี่สำหรับชุดข้อมูลรอยยิ้มของคุณ
หากคุณต้องการทำซ้ำรหัสของเราสำหรับการสร้างชุดข้อมูล ChemBl30 คุณสามารถคลายซิป Molecule_Dataset_Smiles.zip และ/หรือ Molecule_Dataset_Selfies.zip ในไดเรกทอรี ข้อมูล และใช้เป็นรอยยิ้มอินพุต
python3 generate_selfies.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv
ในการสร้างชุดข้อมูลโมเลกุลเซลฟี่โดยใช้โมเดลที่ผ่านการฝึกอบรมมาก่อนโปรดเรียกใช้คำสั่งต่อไปนี้:
python3 produce_embeddings.py --selfies_dataset=data/molecule_dataset_selfies.csv --model_file=data/pretrained_models/SELFormer --embed_file=data/embeddings.csv
Embeddings ที่สร้างขึ้นโดยโมเดลที่ได้รับการฝึกฝนมาก่อนที่มีประสิทธิภาพดีที่สุดของเราสำหรับข้อมูล moleculenet สามารถดาวน์โหลดได้โดยตรงที่นี่
นอกจากนี้คุณยังสามารถสร้าง embeddings เหล่านี้อีกครั้งโดยใช้คำสั่งด้านล่าง
python3 get_moleculenet_embeddings.py --dataset_path=data/finetuning_datasets --model_file=data/pretrained_models/SELFormer
หากต้องการฝึกอบรมแบบจำลองล่วงหน้าโปรดเรียกใช้คำสั่งด้านล่าง หากคุณมีชุดข้อมูลเซลฟี่คุณสามารถใช้มันโดยตรงโดยให้เส้นทางของชุดข้อมูลไปยัง -selfies_dataset หากคุณมีชุดข้อมูล Smiles คุณสามารถให้เส้นทางของชุดข้อมูลไปยัง -Smiles_Dataset และการแสดงเซลฟี่จะถูกสร้างขึ้นที่เส้นทางที่มอบให้ -SELFIES_DATASET
python3 train_pretraining_model.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv --prepared_data_path=data/selfies_data.txt --bpe_path=data/BPETokenizer --roberta_fast_tokenizer_path=data/RobertaFastTokenizer --hyperparameters_path=data/pretraining_hyperparameters.yml --subset_size=100000
คุณสามารถใช้คำสั่งด้านล่างเพื่อปรับแบบจำลองที่ผ่านการฝึกอบรมล่วงหน้าสำหรับงานการทำนายคุณสมบัติโมเลกุลต่างๆ คำสั่งเหล่านี้ใช้เพื่อจัดการชุดข้อมูลที่มีการแสดงรอยยิ้มของโมเลกุล การแสดงรอยยิ้มควรเก็บไว้ในคอลัมน์ที่มีส่วนหัวชื่อ "Smiles" คุณสามารถดูชุดข้อมูลตัวอย่างในไดเรกทอรี ข้อมูล/finetuning_datasets
งานจำแนกประเภทไบนารี
ในการปรับแต่งโมเดลที่ผ่านการฝึกอบรมล่วงหน้าบนชุดข้อมูลการจำแนกแบบไบนารีโปรดเรียกใช้คำสั่งด้านล่าง
python3 train_classification_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/classification/bbbp/bbbp.csv --save_to=data/finetuned_models/SELFormer_bbbp_classification --target_column_id=1 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
งานการจำแนกประเภทหลายฉลาก
ในการปรับแต่งโมเดลที่ผ่านการฝึกอบรมล่วงหน้าบนชุดข้อมูลการจำแนกหลายฉลากโปรดเรียกใช้คำสั่งด้านล่าง ควรเก็บไฟล์ RobertaFastTokenizer ไว้ในไดเรกทอรีเดียวกับรุ่นที่ผ่านการฝึกอบรมมาก่อน
python3 train_classification_multilabel_model.py --model=data/saved_models/SELFormer --dataset=data/finetuning_datasets/classification/tox21/tox21.csv --save_to=data/finetuned_models/SELFormer_tox21_classification --use_scaffold=1 --batch_size=16 --num_epochs=25 --lr=5e-5 --wd=0
งานถดถอย
หากต้องการปรับแต่งโมเดลที่ผ่านการฝึกอบรมล่วงหน้าบนชุดข้อมูลการถดถอยโปรดเรียกใช้คำสั่งด้านล่าง
python3 train_regression_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/regression/esol/esol.csv --save_to=data/finetuned_models/SELFormer_esol_regression --target_column_id=-1 --scaler=2 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
รุ่น Selformer ที่ปรับแต่งได้พร้อมให้ดาวน์โหลดที่นี่ ในการคาดการณ์กับโมเดลเหล่านี้โปรดทำตามคำแนะนำด้านล่าง
ในการคาดการณ์สำหรับชุดข้อมูล BACE, BBBP และ HIV โปรดเรียกใช้คำสั่งด้านล่าง เปลี่ยนอาร์กิวเมนต์ที่ระบุสำหรับงานที่แตกต่างกัน พารามิเตอร์เริ่มต้นจะโหลดโมเดลที่ปรับแต่งอย่างละเอียดบน BBBP
python3 binary_class_pred.py --task=bbbp --model_name=data/finetuned_models/SELFormer_bbbp_scaffold_optimized --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/bbbp/bbbp_mock.csv --training_args=data/finetuned_models/SELFormer_bbbp_scaffold_optimized/training_args.bin
ในการทำนายชุดข้อมูล TOX21 และ SIDER โปรดเรียกใช้คำสั่งด้านล่าง เปลี่ยนอาร์กิวเมนต์ที่ระบุสำหรับงานที่แตกต่างกัน พารามิเตอร์เริ่มต้นจะโหลดโมเดลที่ปรับจูนอย่างละเอียดบน Sider
python3 multilabel_class_pred.py --task=sider --model_name=data/finetuned_models/SELFormer_sider_scaffold_optimized --pred_set=data/finetuning_datasets/classification/sider/sider_mock.csv --training_args=data/finetuned_models/SELFormer_sider_scaffold_optimized/training_args.bin --num_labels=27
เพื่อทำการคาดการณ์สำหรับชุดข้อมูล ESOL, FREESOLV, lipophilicity และ PDBBIND โปรดเรียกใช้คำสั่งด้านล่าง เปลี่ยนอาร์กิวเมนต์ที่ระบุสำหรับงานที่แตกต่างกัน พารามิเตอร์เริ่มต้นจะโหลดโมเดลที่ปรับแต่งอย่างละเอียดบน ESOL
python3 regression_pred.py --task=esol --model_name=data/finetuned_models/esol_regression --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/esol/esol_mock.csv --training_args=data/finetuned_models/esol_regression/training_args.bin
ลิขสิทธิ์ (c) 2023 hubiodatalab
โปรแกรมนี้เป็นซอฟต์แวร์ฟรี: คุณสามารถแจกจ่ายใหม่และ/หรือแก้ไขภายใต้ข้อกำหนดของใบอนุญาตสาธารณะ GNU ทั่วไปที่เผยแพร่โดย Free Software Foundation ไม่ว่าจะเป็นเวอร์ชัน 3 ของใบอนุญาตหรือ (ตามตัวเลือกของคุณ) รุ่นใหม่ ๆ
โปรแกรมนี้มีการแจกจ่ายด้วยความหวังว่าจะมีประโยชน์ แต่ไม่มีการรับประกันใด ๆ โดยไม่มีการรับประกันโดยนัยเกี่ยวกับความสามารถในการค้าหรือความเหมาะสมสำหรับวัตถุประสงค์เฉพาะ ดูใบอนุญาตสาธารณะ GNU ทั่วไปสำหรับรายละเอียดเพิ่มเติม
คุณควรได้รับสำเนาใบอนุญาตสาธารณะ GNU ทั่วไปพร้อมกับโปรแกรมนี้ ถ้าไม่ดู http://www.gnu.org/licenses/