El análisis computacional automatizado del vasto espacio químico es fundamental para numerosos campos de investigación, como el descubrimiento de fármacos y la ciencia de los materiales. Recientemente se han empleado técnicas de aprendizaje de representación con el objetivo principal de generar expresiones numéricas compactas e informativas de datos complejos. Un enfoque para aprender de manera eficiente representaciones moleculares es el procesamiento de anotaciones de productos químicos basados en cadenas a través de algoritmos de procesamiento del lenguaje natural (PNL). La mayoría de los métodos propuestos hasta ahora utilizan anotaciones de sonrisas para este propósito; Sin embargo, las sonrisas se asocian con numerosos problemas relacionados con la validez y la robustez, lo que puede evitar que el modelo descubra efectivamente el conocimiento oculto en los datos. En este estudio, proponemos el autoormador, un modelo de lenguaje químico basado en la arquitectura del transformador que utiliza una notación 100% válida, compacta y expresiva, selfies, como entrada, para aprender representaciones moleculares flexibles y de alta calidad. El autoormer se captura previamente en dos millones de compuestos similares a fármacos y ajustados para diversas tareas de predicción de propiedades moleculares. Nuestra evaluación del rendimiento ha revelado que el autoormador supera todos los métodos competitivos, incluidos los enfoques de aprendizaje gráfico y los modelos de lenguaje químico basados en sonrisas, en la predicción de la solubilidad acuosa de las moléculas y las reacciones adversas a los medicamentos. También visualizamos representaciones moleculares aprendidas por el autoormador a través de la reducción de la dimensionalidad, lo que indicó que incluso el modelo previamente capacitado puede discriminar las moléculas con diferentes propiedades estructurales. Compartimos el autoormador como una herramienta programática, junto con sus conjuntos de datos y modelos previamente capacitados. En general, nuestra investigación demuestra el beneficio de utilizar las anotaciones de selfies en el contexto del modelado de lenguaje químico y abre nuevas posibilidades para el diseño y el descubrimiento de nuevos candidatos de drogas con las características deseadas.
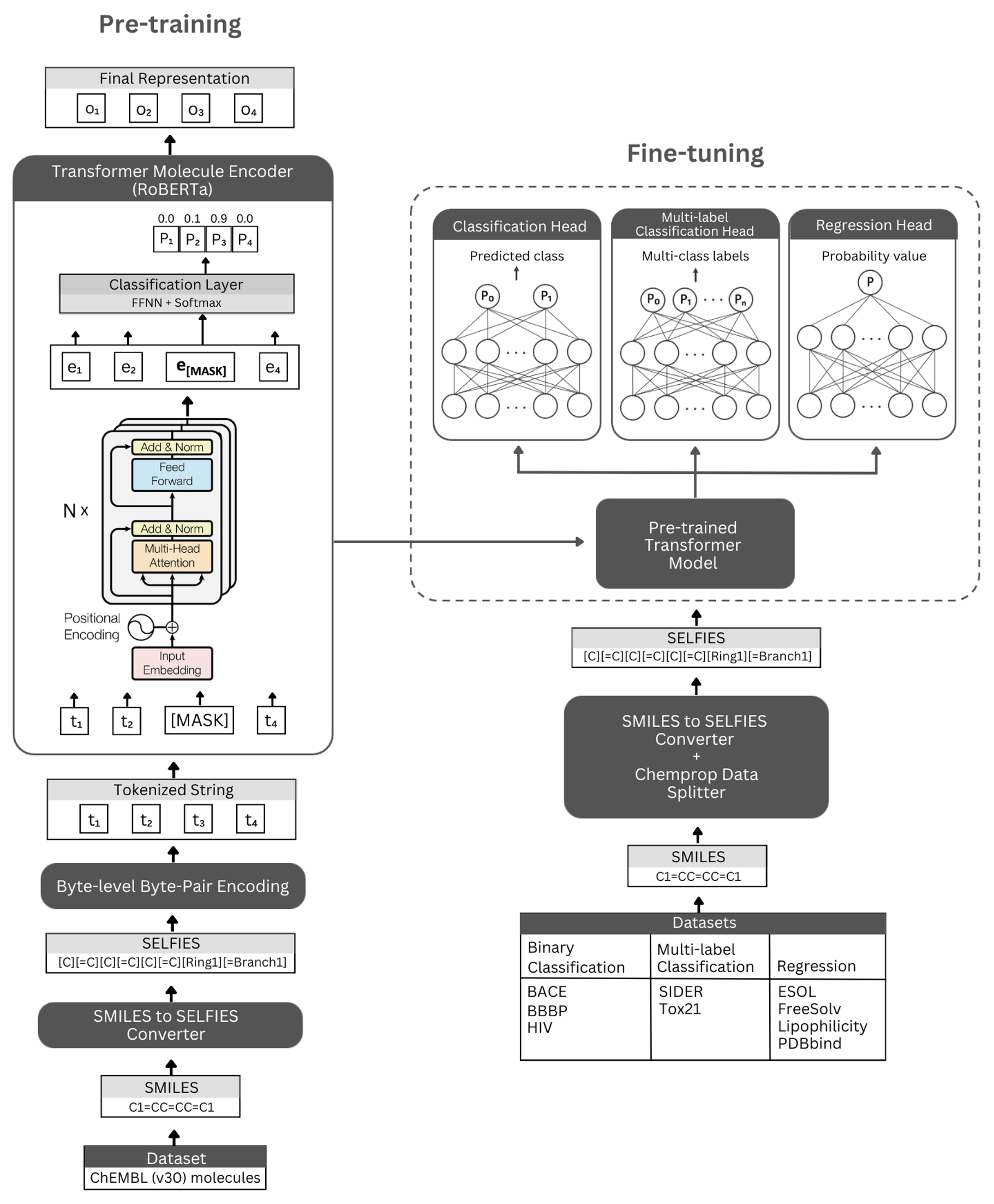
Cifra. La representación esquemática de la arquitectura del autoormador y los experimentos realizados. Izquierda: el pre-entrenador auto-supervisado utiliza el módulo de codificador del transformador a través del modelado de lenguaje enmascarado para el aprendizaje de representaciones concisas e informativas de moléculas pequeñas codificadas por la notación de sus selfies. Derecho: el modelo previamente capacitado se ha ajustado independientemente en numerosas tareas de clasificación y regresión basadas en propiedades moleculares.
Selformer se basa en la arquitectura de Roberta Transformer, que utiliza la misma arquitectura que Bert, pero con ciertas modificaciones que mejoran el rendimiento del modelo o proporcionan otros beneficios. Una de esas modificaciones es el uso de la codificación de pares de bytes a nivel de byte (BPE) para la tokenización en lugar de BPE a nivel de carácter. Otro es que, Roberta se entrena previamente exclusivamente en el objetivo de modelado de lenguaje enmascarado (MLM) al no tener en cuenta la siguiente tarea de predicción de oraciones (NSP). El autoormador tiene (i) modelos pre-capacitados auto-supervisados que utilizan el módulo de codificador del transformador para aprender representaciones concisas e informativas de moléculas pequeñas codificadas por su notación de selfies, y (ii) modelos de clasificación/regresión supervisados que utilizan el modelo previamente intensivo como base y fina en numerosas tareas de predicción de propiedades moleculares basadas en la clasificación y regresión.
Nuestros modelos de codificadores previamente capacitados se implementan como "Robertamaskedlm" y modelos de ajuste fino como "RobertAforsequenceClassification". Para el proceso de ajuste fino, la arquitectura del autoormer incluye el modelo Roberta previamente entrenado como su base, y la clase "RobertAclassificationhead" como las siguientes capas (para clasificación y regresión). La clase "RobertAclassificationhead" consiste en una capa de abandono, una capa densa, función de activación de tanh, una capa de deserción y una capa lineal final. Reenviamos la salida de secuencia del modelo de base Roberta previamente entrenado al clasificador durante el proceso de ajuste fino.
Recomendamos encarecidamente la plataforma de conda para instalar dependencias. Después de la instalación de Conda, cree y active un entorno con dependencias como se define a continuación:
conda create -n SELFormer_env
conda activate SELFormer_env
conda env update --file data/requirements.yml
Los modelos de autoormador previamente capacitados están disponibles para descargar aquí. Los incrustaciones de todas las moléculas de ChemBL30 y ChemBL33 que generan nuestro modelo de mejor rendimiento están disponibles aquí.
También puede generar incrustaciones para su propio conjunto de datos utilizando los modelos previamente capacitados. Para hacerlo, necesitará anotaciones de selfies de sus moléculas. Puede usar el comando a continuación para generar anotaciones de selfies para su conjunto de datos Smiles.
Si desea reproducir nuestro código para generar incrustaciones del conjunto de datos Chembl30, puede descifrar moléculas_dataset_smiles.zip y/o moléculas_dataset_selfies.zip archivos en el directorio de datos y usarlos como conjuntos de datos de entradas y selfies, respectivamente.
python3 generate_selfies.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv
Para generar incrustaciones para el conjunto de datos de la molécula de selfies utilizando un modelo previamente capacitado, ejecute el siguiente comando:
python3 produce_embeddings.py --selfies_dataset=data/molecule_dataset_selfies.csv --model_file=data/pretrained_models/SELFormer --embed_file=data/embeddings.csv
Los incrustaciones generados por nuestro modelo previamente realizado de mejor rendimiento para los datos de Moleculenet se pueden descargar directamente aquí.
También puede volver a generar estos incrustaciones utilizando el comando a continuación.
python3 get_moleculenet_embeddings.py --dataset_path=data/finetuning_datasets --model_file=data/pretrained_models/SELFormer
Para pre-entrenar un modelo, ejecute el comando a continuación. Si tiene un conjunto de datos de selfies, puede usarlo directamente dando la ruta del conjunto de datos a --elfies_dataset . Si tiene un conjunto de datos de sonrisas, puede dar la ruta del conjunto de datos a --smiles_dataset y las representaciones de selfies se crearán en la ruta dada a - -selfies_dataset .
python3 train_pretraining_model.py --smiles_dataset=data/molecule_dataset_smiles.txt --selfies_dataset=data/molecule_dataset_selfies.csv --prepared_data_path=data/selfies_data.txt --bpe_path=data/BPETokenizer --roberta_fast_tokenizer_path=data/RobertaFastTokenizer --hyperparameters_path=data/pretraining_hyperparameters.yml --subset_size=100000
Puede usar comandos a continuación para ajustar un modelo previamente capacitado para varias tareas de predicción de propiedades moleculares. Estos comandos se utilizan para manejar conjuntos de datos que contienen representaciones de sonrisas de moléculas. Las representaciones de las sonrisas deben almacenarse en una columna con un encabezado llamado "Smiles". Puede ver los conjuntos de datos de ejemplo en el directorio de datos/finetuning_datasets .
Tareas de clasificación binaria
Para ajustar un modelo previamente capacitado en un conjunto de datos de clasificación binaria, ejecute el comando a continuación.
python3 train_classification_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/classification/bbbp/bbbp.csv --save_to=data/finetuned_models/SELFormer_bbbp_classification --target_column_id=1 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
Tareas de clasificación de múltiples etiquetas
Para ajustar un modelo previamente capacitado en un conjunto de datos de clasificación de múltiples etiquetas, ejecute el comando a continuación. Los archivos de RoberAfastTokenizer deben almacenarse en el mismo directorio que el modelo previamente capacitado.
python3 train_classification_multilabel_model.py --model=data/saved_models/SELFormer --dataset=data/finetuning_datasets/classification/tox21/tox21.csv --save_to=data/finetuned_models/SELFormer_tox21_classification --use_scaffold=1 --batch_size=16 --num_epochs=25 --lr=5e-5 --wd=0
Tareas de regresión
Para ajustar un modelo previamente capacitado en un conjunto de datos de regresión, ejecute el comando a continuación.
python3 train_regression_model.py --model=data/saved_models/SELFormer --tokenizer=data/RobertaFastTokenizer --dataset=data/finetuning_datasets/regression/esol/esol.csv --save_to=data/finetuned_models/SELFormer_esol_regression --target_column_id=-1 --scaler=2 --use_scaffold=1 --train_batch_size=16 --validation_batch_size=8 --num_epochs=25 --lr=5e-5 --wd=0
Los modelos de autoormador ajustados están disponibles para descargar aquí. Para hacer predicciones con estos modelos, siga las instrucciones a continuación.
Para hacer predicciones para conjuntos de datos BACE, BBBP y VIH, ejecute el comando a continuación. Cambie los argumentos indicados para diferentes tareas. Los parámetros predeterminados cargarán el modelo ajustado en BBBP.
python3 binary_class_pred.py --task=bbbp --model_name=data/finetuned_models/SELFormer_bbbp_scaffold_optimized --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/bbbp/bbbp_mock.csv --training_args=data/finetuned_models/SELFormer_bbbp_scaffold_optimized/training_args.bin
Para hacer predicciones para conjuntos de datos Tox21 y Sider, ejecute el comando a continuación. Cambie los argumentos indicados para diferentes tareas. Los parámetros predeterminados cargarán el modelo ajustado en Sider.
python3 multilabel_class_pred.py --task=sider --model_name=data/finetuned_models/SELFormer_sider_scaffold_optimized --pred_set=data/finetuning_datasets/classification/sider/sider_mock.csv --training_args=data/finetuned_models/SELFormer_sider_scaffold_optimized/training_args.bin --num_labels=27
Para hacer predicciones para ESOL, Freesolv, lipofilia y conjuntos de datos PDBBind, ejecute el comando a continuación. Cambie los argumentos indicados para diferentes tareas. Los parámetros predeterminados cargarán el modelo ajustado en ESOL.
python3 regression_pred.py --task=esol --model_name=data/finetuned_models/esol_regression --tokenizer=data/RobertaFastTokenizer --pred_set=data/finetuning_datasets/classification/esol/esol_mock.csv --training_args=data/finetuned_models/esol_regression/training_args.bin
Copyright (c) 2023 Hubiodatalab
Este programa es un software gratuito: puede redistribuirlo y/o modificarlo bajo los términos de la Licencia Pública General de GNU publicada por Free Software Foundation, ya sea la versión 3 de la licencia o (a su opción) cualquier versión posterior.
Este programa se distribuye con la esperanza de que sea útil, pero sin ninguna garantía; Sin siquiera la garantía implícita de comerciabilidad o estado físico para un propósito particular. Vea la Licencia Pública General de GNU para más detalles.
Debería haber recibido una copia de la Licencia Pública General de GNU junto con este programa. Si no, consulte http://www.gnu.org/licenses/.