PIA是用于基于MS的蛋白质推理和鉴定分析的工具箱。
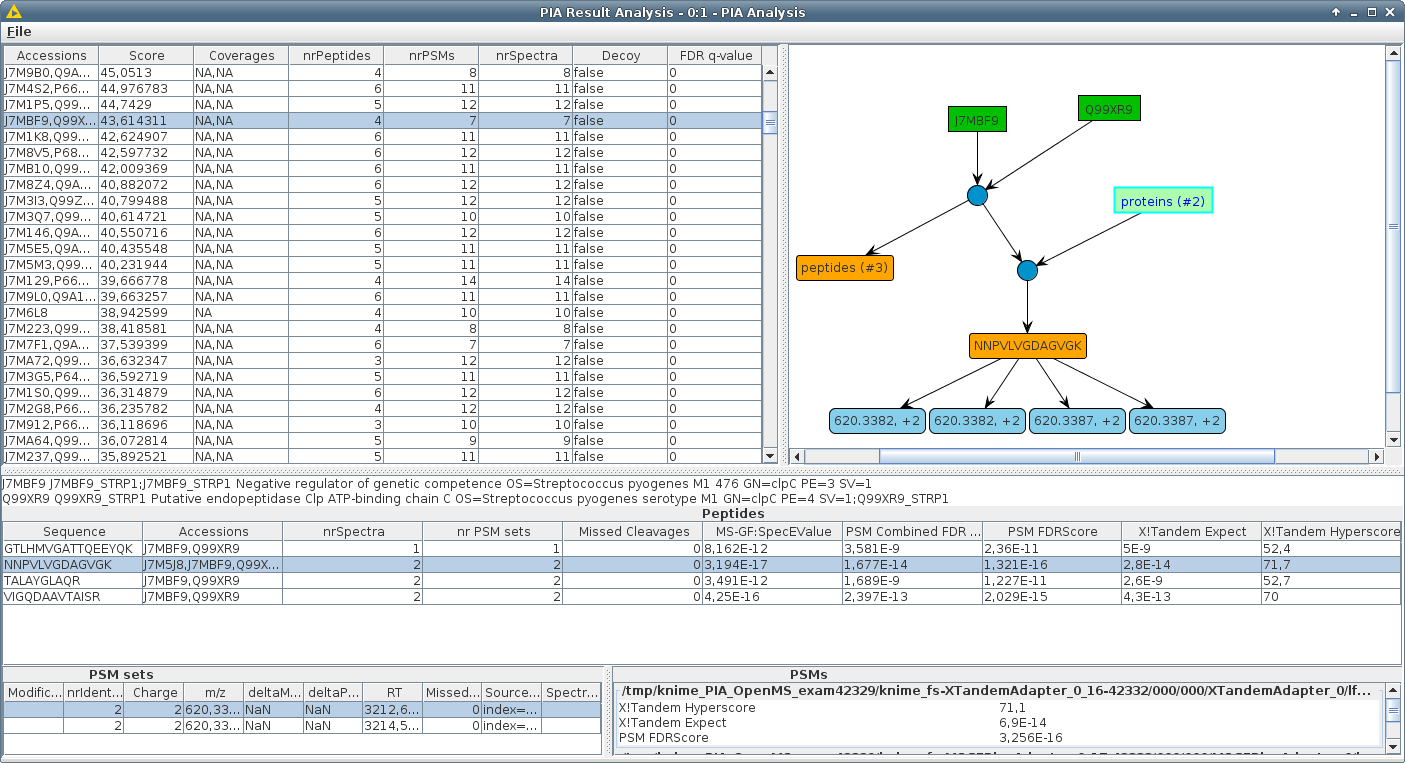
PIA允许您检查常见的蛋白质组学识别搜索引擎的结果,无缝地组合并进行统计分析。 PIA的主要重点放在综合的推理算法上,即从一组确定的光谱中得出结论蛋白质。但这也使您可以检查肽光谱匹配,计算不同搜索引擎结果的FDR值,并可视化PSM,肽和蛋白质之间的对应关系。

尽管实际搜索产生了一组肽光谱匹配(PSMS),但大多数搜索MS/MS实验中蛋白质识别的引擎都会返回蛋白质列表。从PSM到蛋白质的步骤称为“蛋白质推理”。如果一组确定的PSM支持在搜索数据库中检测多个蛋白质(“蛋白质歧义”),则通常只报告一个代表性登录。这些代表可能会根据使用的搜索引擎和设置而有所不同。因此,通常无法将不同搜索引擎的蛋白质列表相互比较。互补搜索引擎的PSM通常被合并以增强报告的蛋白质数量或验证肽的证据,该证据通过使用不同算法的检测来改善。
我们开发了一个用Java编写的算法套件,包括完全可参数的knime节点,该节点结合了来自不同实验和/或搜索引擎的PSM,并报告一致,因此结果一致。没有像先前的方法那样固定的参数,例如过滤或评分,但要保持尽可能灵活,以允许用户需要进行任何调整。
PIA可以通过命令行(也在Docker容器中)或工作流环境knime中调用,这允许无缝集成到OpenMS工作流程中。
对于命令行,您可以使用conda(分别bioconda)下载最新发布的版本,也可以在此处下载构建版本。
PIA也被整合到刀片中。您可以轻松地从可信赖的社区贡献存储库中安装它,该存储库都有所有较新的刀具版本。在提交任何错误或问题之前,请始终使用最新版本的knime。在Wiki中可以找到有关如何在Knime中找到有关PIA内部PIA的更多信息。
可以在此处下载的教程AS PDF,可在此处找到所需的数据以及此处的工作流(所有数据也可以在https://github.com/mpc-bioinformics/pia-tutorial/的教程存储库中可用)。
有关更多文档,请参阅Github上的Wiki(https://github.com/medbioinf/pia/wiki)。
如果您在PIA有任何问题或查找错误和其他问题,请使用GitHub的问题跟踪器(https://github.com/medbioinf/pia/issues)。
如果您发现PIA对您的工作有用,请引用以下出版物:
https://www.ncbi.nlm.nih.gov/pubmed/25938255
https://www.ncbi.nlm.nih.gov/pubmed/30474983
PIA的发展由德国生物信息学基础设施网络Elixir / De.NBI资助。
PIA的编程工作由Julian Uszkoreit(Ruhr University Bochum,医学生物信息学)和Yasset Perez-Riverol(欧洲生物信息学研究所(EMBL-EBI),剑桥)进行