PIA هو صندوق أدوات لاستدلال البروتين على أساس MS وتحليل تحديد الهوية.
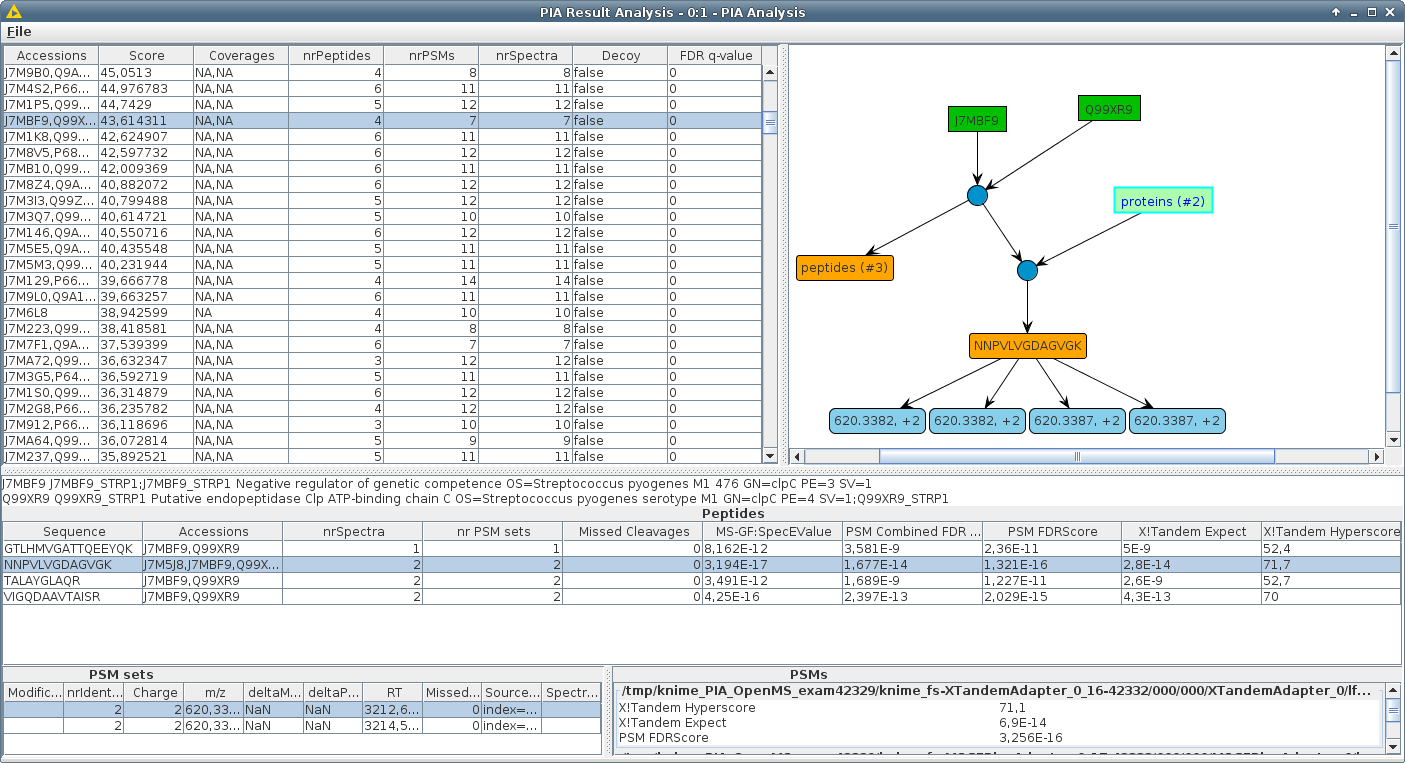
يتيح لك PIA فحص نتائج محركات البحث في تحديد طيف البروتينات الشائعة ، ودمجها بسلاسة وإجراء تحليلات إحصائية. يركز التركيز الرئيسي لـ PIA على خوارزميات الاستدلال المتكاملة ، أي إبرام البروتينات من مجموعة من الأطياف المحددة. ولكنه يتيح لك أيضًا فحص تطابق طيف الببتيد الخاص بك وحساب قيم FDR عبر نتائج محرك البحث المختلفة وتصور المراسلات بين PSM و Peptides والبروتينات.

معظم محركات البحث لتحديد البروتين في تجارب MS/MS لإرجاع قوائم البروتين ، على الرغم من أن البحث الفعلي يعطي مجموعة من تطابق طيف الببتيد (PSMS). وتسمى الخطوة من PSMs إلى البروتينات "استنتاج البروتين". إذا كانت مجموعة من PSMs المحددة تدعم اكتشاف أكثر من بروتين واحد في قاعدة البيانات التي تم تفتيشها ("غموض البروتين") ، عادة ما يتم الإبلاغ عن انضمام تمثيلي واحد فقط. قد يختلف هؤلاء الممثلون وفقًا لمحرك البحث والإعدادات المستخدمة. وبالتالي ، لا يمكن مقارنة قوائم البروتين بمحركات البحث المختلفة بشكل عام مع بعضها البعض. غالبًا ما يتم دمج PSMS لمحركات البحث التكميلية لتعزيز عدد البروتينات المبلغ عنها أو للتحقق من أدلة الببتيد ، والتي يتم تحسينها عن طريق الكشف مع خوارزميات مميزة.
لقد قمنا بتطوير مجموعة خوارزمية مكتوبة في Java ، بما في ذلك عقد الركبتين القابلة للتصوير بالكامل ، والتي تجمع بين PSM من تجارب مختلفة و/أو محركات البحث ، وتقارير متسقة وبالتالي نتائج مماثلة. لا يتم إصلاح أي من المعلمات ، مثل التصفية أو التسجيل ، كما هو الحال في الأساليب السابقة ، ولكنها مرنة بقدر الإمكان ، للسماح بأي تعديلات يحتاجها المستخدم.
يمكن استدعاء PIA عبر سطر الأوامر (أيضًا في حاويات Docker) أو في ركبة بيئة سير العمل ، مما يتيح تكاملًا سلسًا في سير العمل OpenMs.
بالنسبة لخط الأوامر ، يمكنك تنزيل أحدث إصدار تم إصداره باستخدام Conda (على التوالي Bioconda) أو تنزيل Build هنا.
تم دمج PIA أيضًا في Knime. يمكنك بسهولة تثبيته من مستودع مساهمات المجتمع الموثوق به ، والذي يتوفر في جميع إصدارات الركبتين الأحدث. يرجى دائمًا استخدام أحدث إصدار من Knime قبل تقديم أي أخطاء أو مشكلات. يمكن العثور على مزيد من المعلومات حول كيفية تثبيت وتشغيل PIA داخل Knime في الويكي حول PIA في Knime.
يمكن تنزيل البرنامج التعليمي كـ PDF هنا ، وتتوفر البيانات المطلوبة هنا وسير العمل هنا (جميع البيانات متوفرة أيضًا في مستودع البرنامج التعليمي على https://github.com/mpc-bioinforformic/PIA-TUTORALL/).
لمزيد من الوثائق ، يرجى الرجوع إلى wiki (https://github.com/medbioinf/pia/wiki) على github.
إذا كانت لديك أي مشاكل مع PIA أو تجد الأخطاء وغيرها من المشكلات ، فيرجى استخدام متتبع GitHub (https://github.com/medbioinf/pia/issues).
إذا وجدت PIA مفيدًا لعملك ، فيرجى الاستشهاد بالمنشورات التالية:
https://www.ncbi.nlm.nih.gov/pubmed/25938255
https://www.ncbi.nlm.nih.gov/pubmed/30474983
يتم تمويل تطوير PIA بواسطة Elixir / De.NBI ، الشبكة الألمانية للبنية التحتية للمعلوماتية الحيوية.
تم تنفيذ أعمال البرمجة على PIA بواسطة جوليان أوزيكوريت (جامعة رور بوشوم ، والمعلوماتية الحيوية الطبية) ، و Yasset Perez-Riverol (معهد المعلوماتية الحيوية الأوروبية (EMBL-EBI) ، كامبريدج)