PIA는 MS 기반 단백질 추론 및 식별 분석을위한 도구 상자입니다.
PIA를 사용하면 일반적인 Proteomics Spectrum 식별 검색 엔진의 결과를 검사하고 원활하게 결합하여 통계 분석을 수행 할 수 있습니다. PIA의 주요 초점은 통합 된 추론 알고리즘에 놓여 있으며, 즉, 식별 된 스펙트럼 세트에서 단백질을 결론 지었다. 그러나 펩티드 스펙트럼 일치를 검사하고 다양한 검색 엔진 결과에서 FDR 값을 계산하며 PSM, 펩티드 및 단백질 사이의 대응을 시각화 할 수 있습니다.

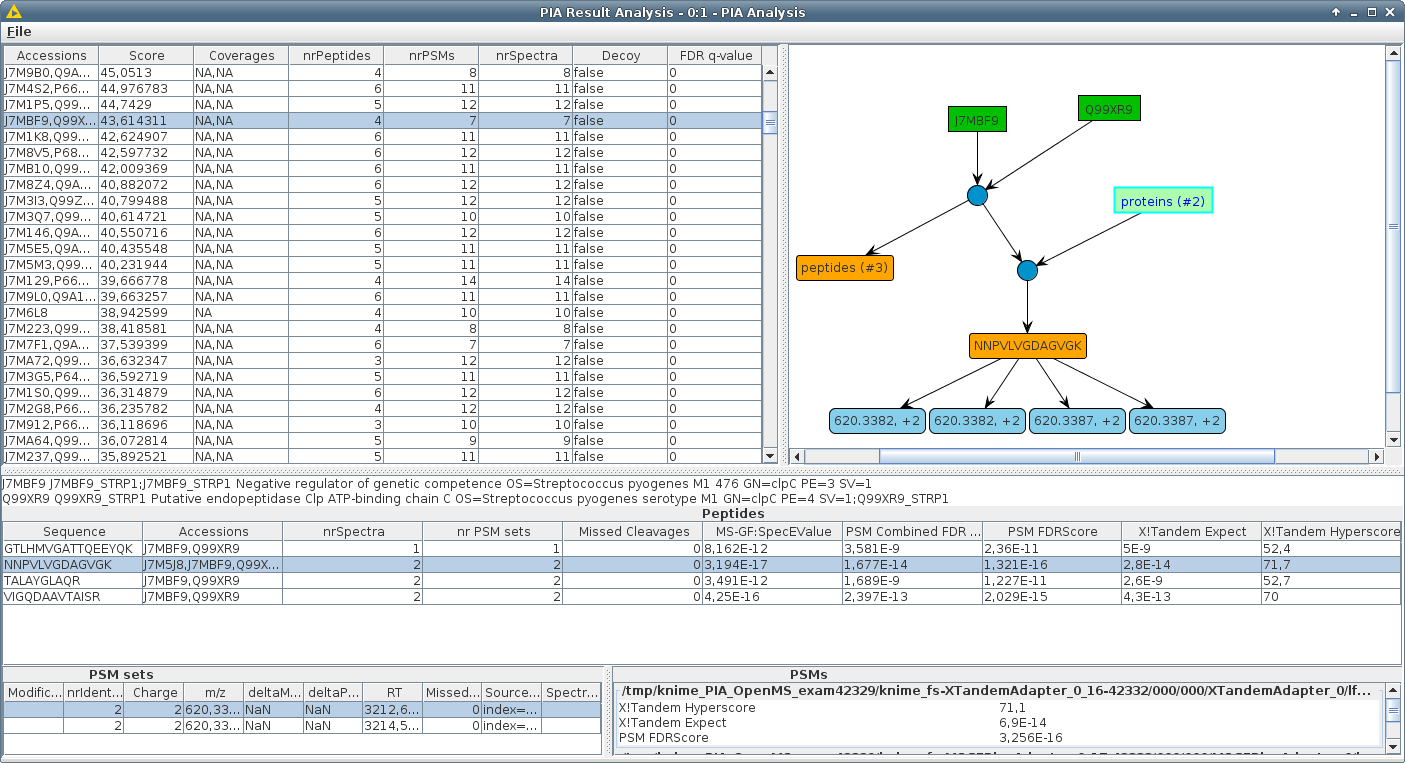
MS/MS 실험에서 단백질 식별을위한 대부분의 검색 엔진은 단백질 목록을 반환하지만, 실제 검색은 펩티드 스펙트럼 일치 (PSM) 세트를 생성합니다. PSM에서 단백질로의 단계를 "단백질 추론"이라고합니다. 식별 된 PSM 세트가 검색 된 데이터베이스 ( "단백질 모호성")에서 하나 이상의 단백질의 검출을 지원하는 경우 일반적으로 하나의 대표적인 가입 만보 고됩니다. 이 담당자는 중고 검색 엔진 및 설정에 따라 다를 수 있습니다. 따라서 다른 검색 엔진의 단백질 목록은 일반적으로 서로 비교할 수 없습니다. 보완적인 검색 엔진의 PSM은 종종보고 된 단백질의 수를 향상 시키거나 펩티드의 증거를 검증하기 위해 결합되며, 이는 별개의 알고리즘으로 검출하여 개선됩니다.
우리는 다양한 실험 및/또는 검색 엔진의 PSM을 결합한 완전히 매개 변수성 KNIME 노드를 포함하여 Java로 작성된 알고리즘 제품군을 개발하여 일관되고 비슷한 결과를보고했습니다. 필터링 또는 스코어링과 같은 매개 변수 중 어느 것도 이전 접근법에서와 같이 고정되어 있지 않지만 사용자가 필요한 조정을 허용하기 위해 가능한 한 유연하게 유지됩니다.
PIA는 명령 줄 (도커 컨테이너) 또는 워크 플로 환경에서 호출 할 수 있으며, 이로 인해 OpenMS 워크 플로우에 원활한 통합이 가능합니다.
명령 줄의 경우 Conda (각각 BioConda)를 사용하여 최신 릴리스 버전을 다운로드하거나 여기에서 빌드를 다운로드 할 수 있습니다.
PIA는 또한 Knime에 통합되어 있습니다. 모든 새로운 KNIME 버전으로 제공되는 신뢰할 수있는 Community Contributions Repository에서 쉽게 설치할 수 있습니다. 버그 나 문제를 제출하기 전에 항상 최신 KNIME 버전을 사용하십시오. Knime 내부의 PIA를 설치하고 실행하는 방법에 대한 자세한 내용은 Wiki에서 Knime의 PIA에 대한 정보를 찾을 수 있습니다.
PDF로서의 튜토리얼은 여기에서 다운로드 할 수 있으며 여기에서 필요한 데이터를 사용할 수 있으며 여기에서 워크 플로우 (모든 데이터는 https://github.com/mpc-bioinformatics/pia-tutorial/)에서도 사용할 수 있습니다).
추가 문서는 Github의 Wiki (https://github.com/medbioinf/pia/wiki)를 참조하십시오.
PIA에 문제가 있거나 버그 및 기타 문제를 찾으면 GitHub (https://github.com/medbioinf/pia/issues)의 문제 추적기를 사용하십시오.
PIA가 작업에 유용한 것을 발견하면 다음 간행물을 인용하십시오.
https://www.ncbi.nlm.nih.gov/pubmed/25938255
https://www.ncbi.nlm.nih.gov/pubmed/30474983
PIA의 개발은 독일 생물 정보 인프라 네트워크 인 Elixir / de.nbi가 자금을 지원합니다.
PIA에 대한 프로그래밍 작업은 Julian Uszkoreit (Ruhr University Bochum, Medical Bioinformatics) 및 Yasset Perez-Riverol (European Bioinformatics Institute (Embl-Ebi), Cambridge)에 의해 수행되었습니다.