โดย Rahmatullah Roche, Sumit Tarafder และ Debswapna Bhattacharya
[Biorxiv] [PDF]
Codebase สำหรับวิธีการทำนายโครงสร้างโปรตีน -RNA ที่ซับซ้อนลำดับเดียวของเรา PRORNA3D-Single
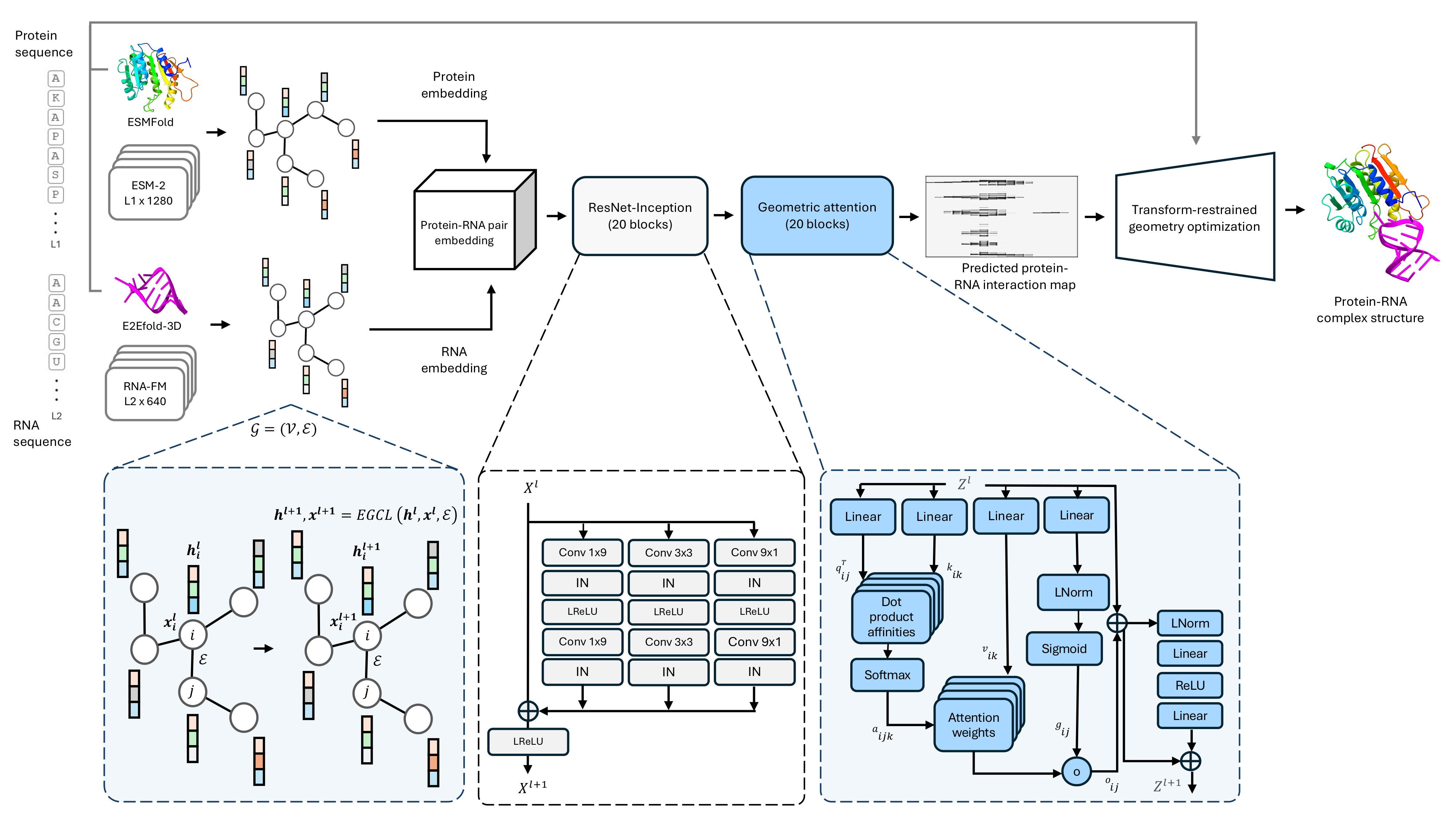
1. ) เราขอแนะนำสภาพแวดล้อมเสมือนจริงของ Conda เพื่อติดตั้งการพึ่งพาสำหรับ ProRNA3D-Single คำสั่งต่อไปนี้จะสร้างสภาพแวดล้อมเสมือนจริงชื่อ 'ProRNA3D-Single'
conda env create -f ProRNA3D-single_environment.yml
2. ) จากนั้นเปิดใช้งานสภาพแวดล้อมเสมือนจริง
conda activate ProRNA3D-single
3. ) สคริปต์การพับต้องใช้ไพโรเซ็ตต้าซึ่งสามารถติดตั้งได้หลังจากการเกิดเหล่านี้
4. ) ดาวน์โหลดโมเดลที่ผ่านการฝึกอบรมจากที่นี่และสถานที่ภายใน ProRNA3D_model/
curl --output ProRNA3D_model/model.pt "https://zenodo.org/records/11477127/files/model.pt?download=1"
แค่ไหน! ProRNA3D-Single พร้อมที่จะใช้
1. ) วางโปรตีนและโมโนเมอร์ RNA (PDBs) ภายใน inputs/
2. ) วาง ESM2 EMBEDDINGS ภายใน inputs/ (ดูตัวอย่าง inputs/7ZLQB.rep_1280.npy ) และวาง RNA-FM EMBEDDINGs ภายใน inputs/ (ดูตัวอย่างที่นี่ inputs/7ZLQC_RNA.npy )
3. ) วางโปรตีน (C α -C α ) แผนที่ระยะทางภายใน prot_dist/ (ดูตัวอย่าง prot_dist/7ZLQB_prot.dist ) และวาง RNA (C ' 4 -C' 4 ) แผนที่ระยะทางภายใน rna_dist/ (ดูตัวอย่าง rna_dist/7ZLQC_RNA.c4p.dist )
4. ) ใส่รายการเป้าหมายในไฟล์ inputs.list รายการภายใน inputs/
5. ) วิ่ง
python run_predictions.py
สคริปต์นี้จะเรียกใช้การอนุมานและสร้างการโต้ตอบระหว่างโปรตีน -RNA ภายใน out_inter_rr/ จากนั้นมันจะเปลี่ยนการคาดการณ์เป็นโครงสร้างที่ซับซ้อนโปรตีน 3 มิติที่พับได้ภายใน predictions/
Datasets/