por Rahmatullah Roche, Sumit Tarafder y Debswapna Bhattacharya
[Biorxiv] [PDF]
CodeBase para nuestro método de predicción de estructura de complejo de proteína de proteína de secuencia única, Prorna3D-Single.
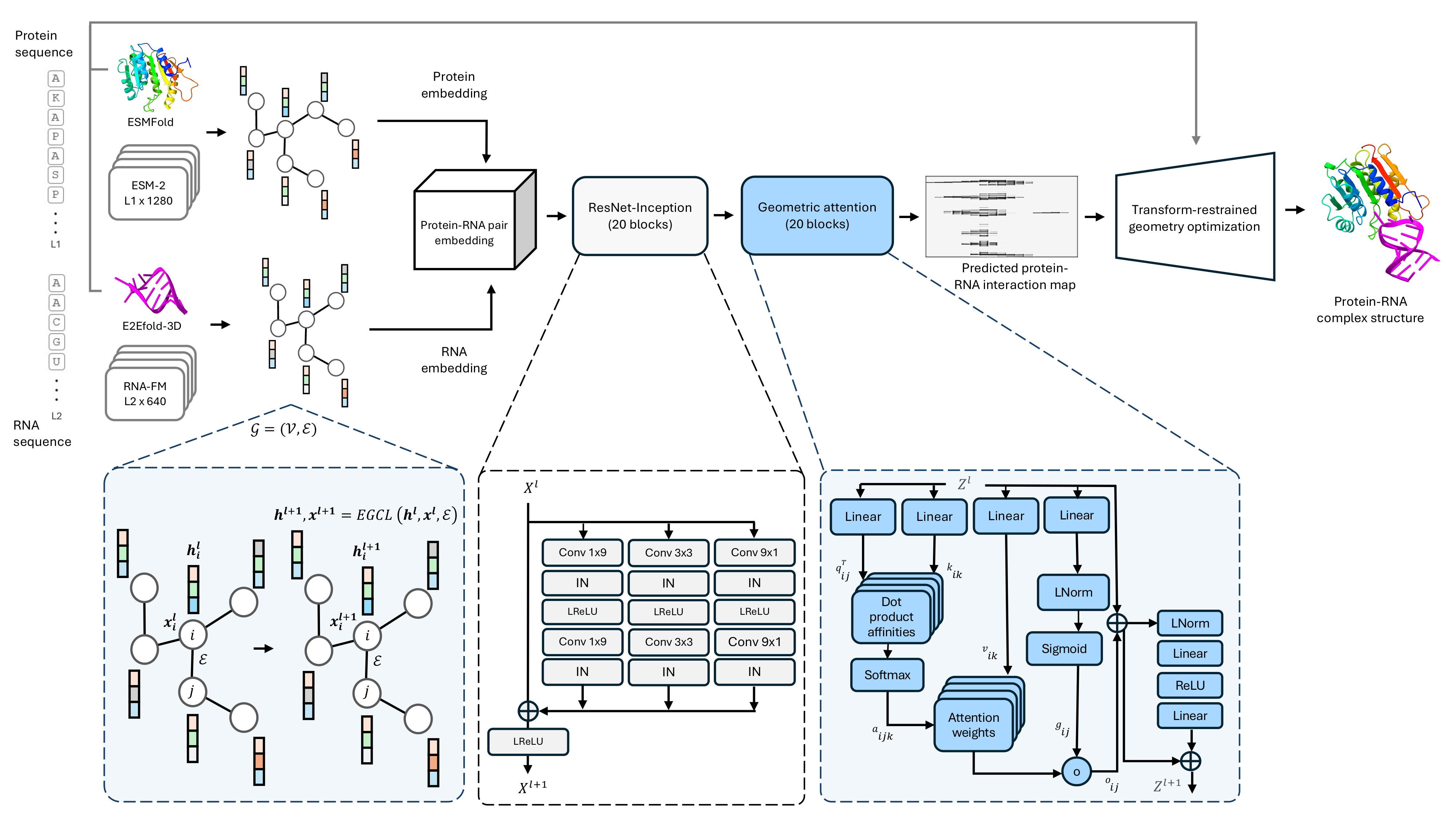
1.) Recomendamos el entorno virtual de Conda para instalar dependencias para Prorna3d-Single. El siguiente comando creará un entorno virtual llamado 'Prorna3d-Single'
conda env create -f ProRNA3D-single_environment.yml
2.) luego active el entorno virtual
conda activate ProRNA3D-single
3.) El script plegable requiere Pyrosetta, que se puede instalar después de estas instucción.
4.) Descargue el modelo capacitado desde aquí y colóquelo dentro de ProRNA3D_model/
curl --output ProRNA3D_model/model.pt "https://zenodo.org/records/11477127/files/model.pt?download=1"
¡Eso es todo! Prorna3D-Single está listo para ser utilizado.
1.) Coloque los monómeros de proteína y ARN (PDB) dentro de inputs/
2.) Coloque las incrustaciones de ESM2 dentro de inputs/ (ver Ejemplo inputs/7ZLQB.rep_1280.npy ) y coloque las incrustaciones de ARN-FM dentro de inputs/ (consulte el ejemplo aquí inputs/7ZLQC_RNA.npy ).
3.) Coloque los mapas de distancia de proteína (C α -C α ) dentro de prot_dist/ (ver Ejemplo prot_dist/7ZLQB_prot.dist ), y coloque los mapas de distancia de ARN (C ' 4 -c' 4 ) dentro de rna_dist/ (ver Ejemplo rna_dist/7ZLQC_RNA.c4p.dist ).
4.) Ponga la lista de objetivos en las inputs.list del archivo. Lista dentro de inputs/ .
5.) Run
python run_predictions.py
Este script ejecutará inferencia y generará interacciones entre proteínas-ARNt dentro de out_inter_rr/ . Luego transformará las predicciones en estructuras complejas de ARN de proteína 3D plegadas dentro de predictions/
Datasets/ .