Rahmatullah Roche, Sumit Tarafder 및 Debspapna Bhattacharya
[Biorxiv] [PDF]
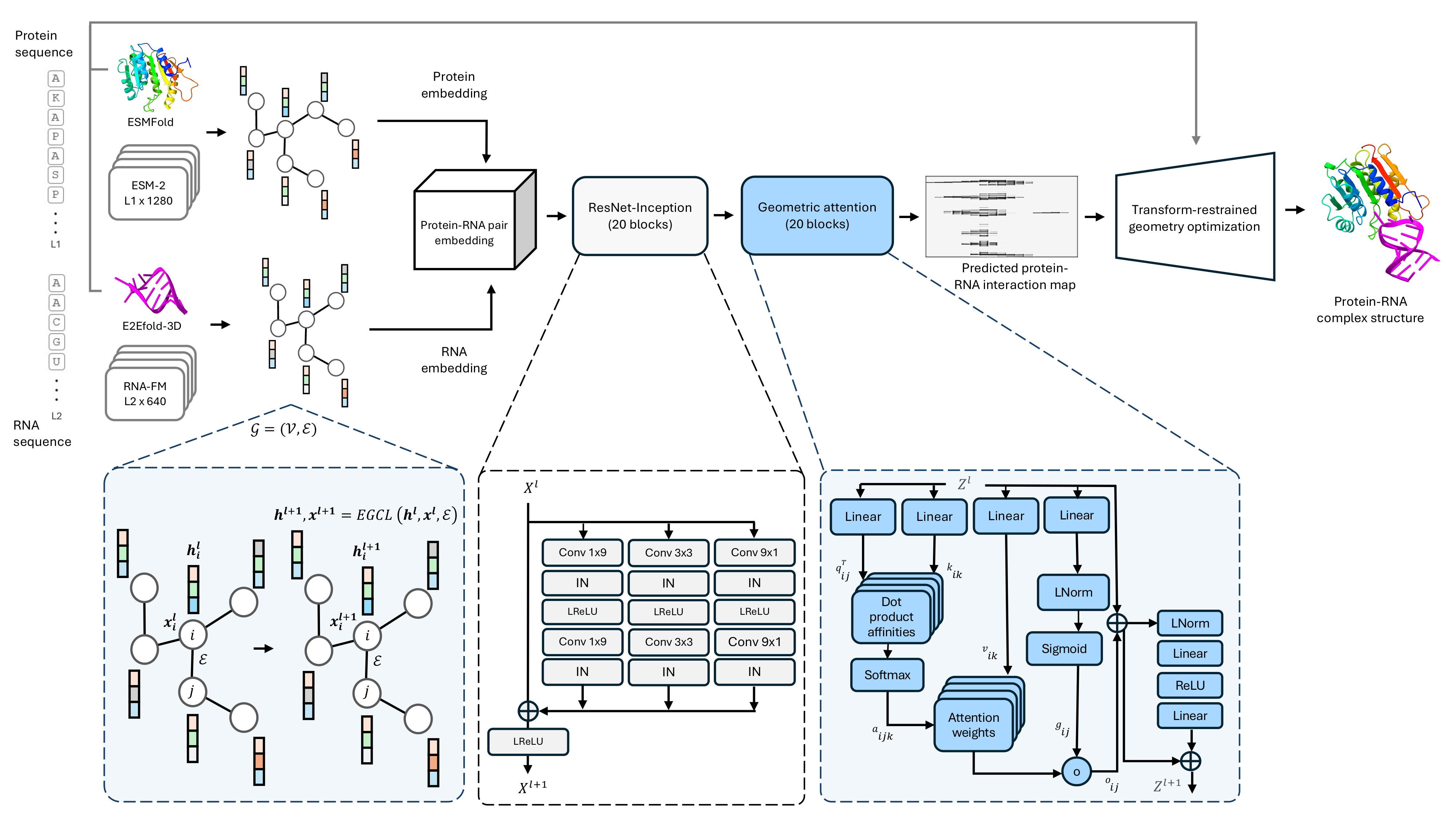
단일 시퀀스 단백질 -RNA 복잡한 구조 예측 방법, prorna3d-single에 대한 코드베이스.
1.) PRORNA3D 단일의 종속성을 설치하려면 Conda Virtual Environment를 권장합니다. 다음 명령은 'prorna3d-single'이라는 가상 환경을 만듭니다.
conda env create -f ProRNA3D-single_environment.yml
2.) 그런 다음 가상 환경을 활성화하십시오
conda activate ProRNA3D-single
3.) 접이식 스크립트에는 Pyrosetta가 필요하며, 이러한 인stuctions에 따라 설치할 수 있습니다.
4.) 여기에서 훈련 된 모델을 다운로드하고 ProRNA3D_model/ 내부에 배치하십시오.
curl --output ProRNA3D_model/model.pt "https://zenodo.org/records/11477127/files/model.pt?download=1"
그게 다야! PRORNA3D-SILLE을 사용할 준비가되었습니다.
1.) 단백질 및 RNA 단량체 (PDBS)를 inputs/ 내부에 놓습니다.
2.) inputs/ inputs/7ZLQB.rep_1280.npy 예제 참조)에 ESM2 임베딩을 배치하고 RNA-FM 임베딩을 inputs/ 내부에 배치하십시오 (여기서 inputs/7ZLQC_RNA.npy 예제 참조).
3.) prot_dist/ ( prot_dist/7ZLQB_prot.dist 예제 참조)에 단백질 (C α -C α ) 거리 맵을 놓고 RNA (C ' 4 -C'4 ) 거리지도를 rna_dist/ (예제 rna_dist/7ZLQC_RNA.c4p.dist 참조) 배치하십시오.
4.) 대상 목록을 파일 inputs.list 내부 inputs/ 에 넣습니다.
5.) 실행
python run_predictions.py
이 스크립트는 추론을 실행하고 out_inter_rr/ 내에서 단백질 간 RNA 상호 작용을 생성합니다. 그런 다음 예측을 접힌 3D 단백질 -RNA 복합체 구조로 변환합니다 predictions/
Datasets/ 내부에서 사용할 수 있습니다.