Rahmatullah Roche、Sumit Tarafder、およびDebswapna Bhattacharya
[biorxiv] [pdf]
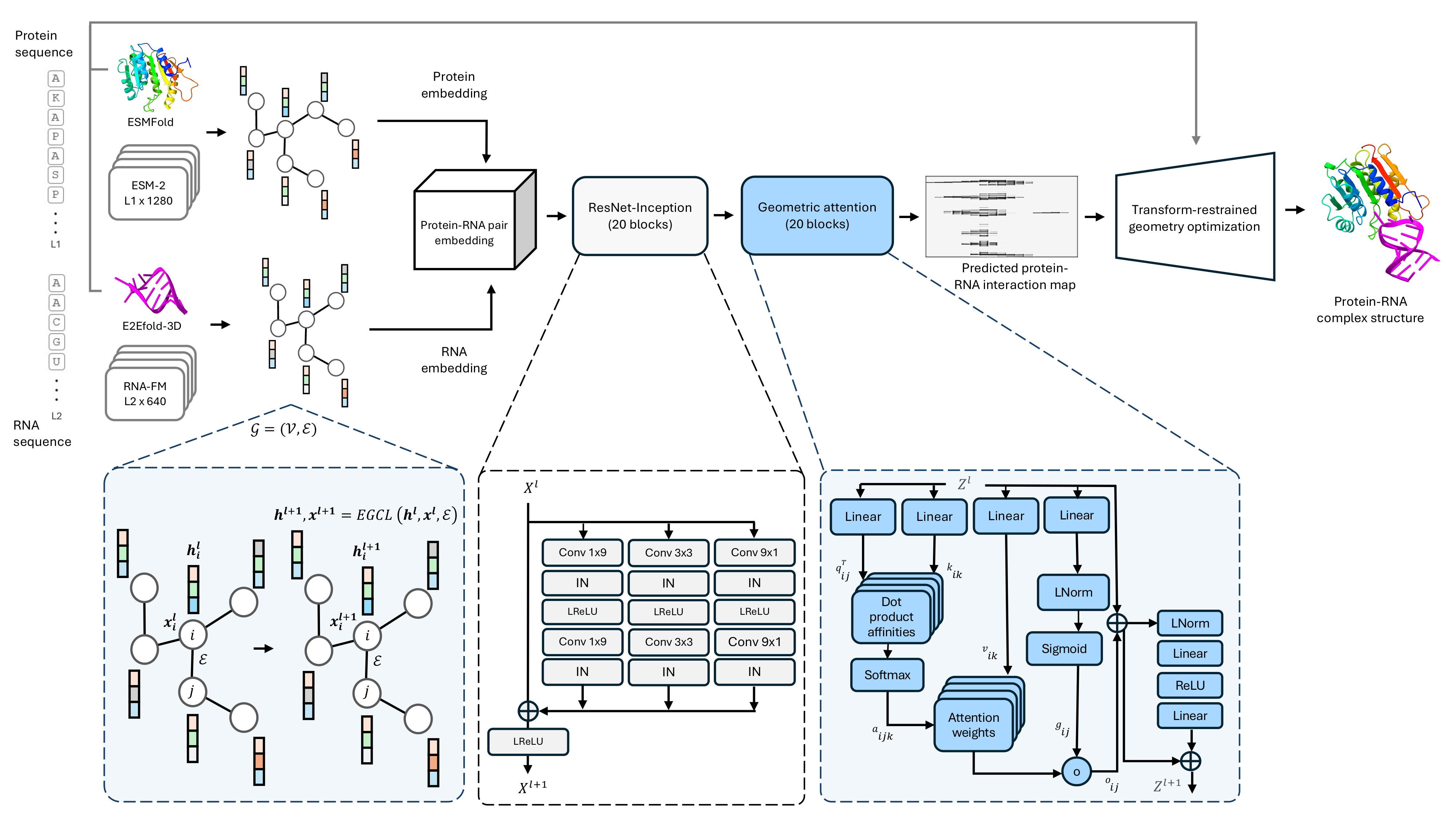
シングルシーケンスタンパク質-RNA複合体構造予測法のコードベースProRNA3D-Single。
1.)conda Virtual Environmentは、prona3d-singleの依存関係をインストールすることをお勧めします。次のコマンドは、「Prona3d-single」という名前の仮想環境を作成します
conda env create -f ProRNA3D-single_environment.yml
2.)次に、仮想環境をアクティブにします
conda activate ProRNA3D-single
3.)折りたたみスクリプトには、これらの施設に従ってインストールできるPyrosettaが必要です。
4.)ここから訓練されたモデルをダウンロードし、 ProRNA3D_model/内側に配置します
curl --output ProRNA3D_model/model.pt "https://zenodo.org/records/11477127/files/model.pt?download=1"
それでおしまい! ProRNA3D-Singleを使用する準備ができています。
1.)タンパク質とRNAモノマー(PDB)をinputs/
2.)ESM2埋め込みをinputs/内に配置します( inputs/7ZLQB.rep_1280.npyを参照)、RNA-FM埋め込みをinputs/内部に配置します(ここでinputs/7ZLQC_RNA.npy例を参照)。
3.) prot_dist/ ( prot_dist/7ZLQB_prot.dist例を参照)内部にタンパク質(Cα- cα )距離マップを配置し、 rna_dist/内にRNA(C ' 4 -C' 4 )距離マップを配置します( rna_dist/7ZLQC_RNA.c4p.distを参照)。
4.)inputs内部のファイルinputs.listにターゲットのリストをinputs/配置します。
5.)実行します
python run_predictions.py
このスクリプトは、推論を実行し、タンパク質間RNA相互作用を生成しますout_inter_rr/ 。次に、予測をpredictions/
Datasets/内で利用できます。