بقلم رحمة الله روش ، سوميت تارافدر ، وديبسوابنا بهاتشاريا
[Biorxiv] [PDF]
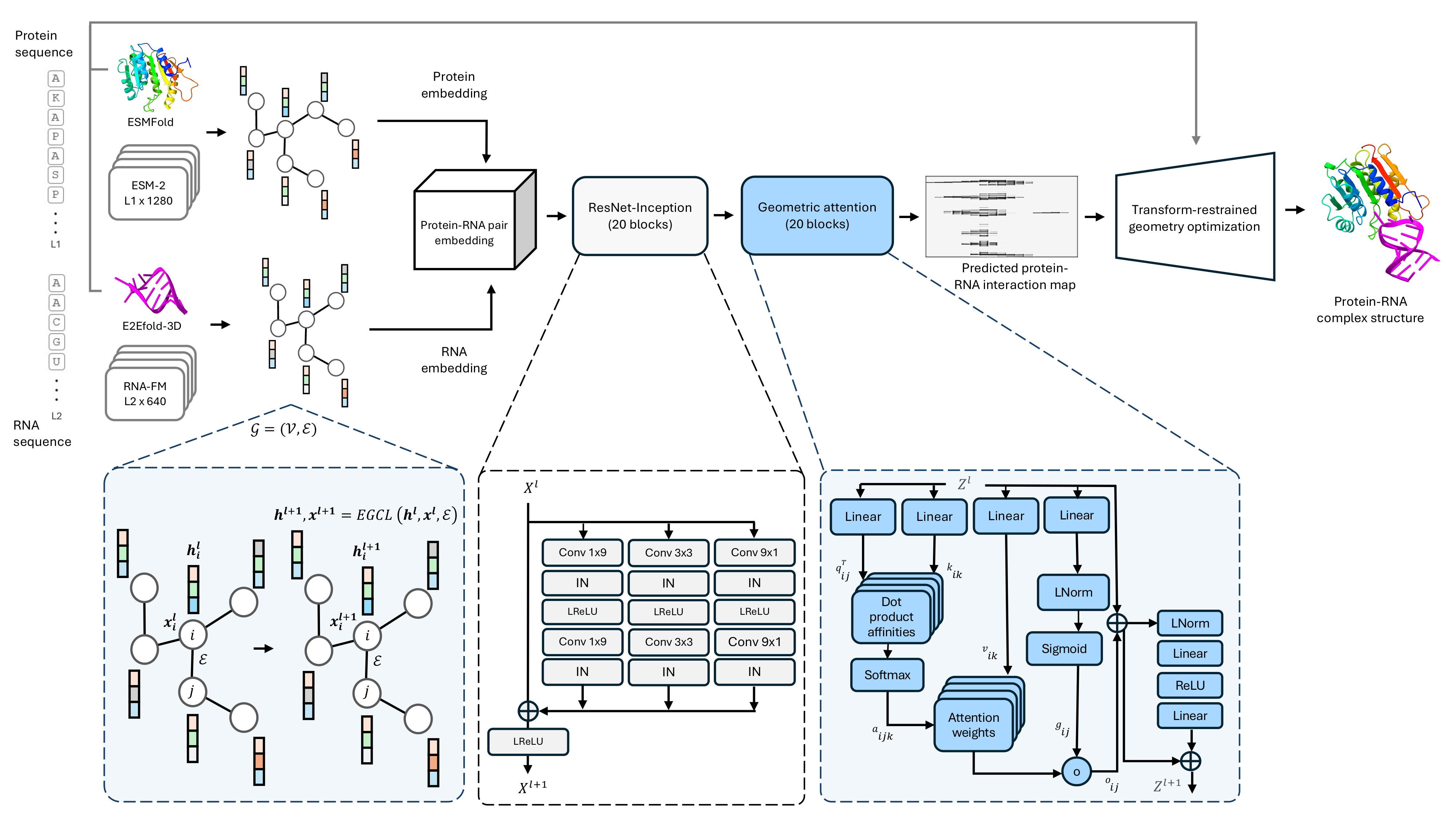
Codebase لطريقة التنبؤ البنية المعقدة للبروتين RNA التسلسل أحادي التسلسل ، PRORNA3D-single.
1.) نوصي بالبيئة الافتراضية كوندا لتثبيت التبعيات لـ PRORNA3D-Single. سيقوم الأمر التالي بإنشاء بيئة افتراضية تسمى "PRORNA3D-Single"
conda env create -f ProRNA3D-single_environment.yml
2.) ثم تفعيل البيئة الافتراضية
conda activate ProRNA3D-single
3.) يتطلب البرنامج النصي القابل للطي pyrosetta ، والذي يمكن تثبيته بعد هذه المؤسسات.
4.) قم بتنزيل النموذج المدرب من هنا ووضعه داخل ProRNA3D_model/
curl --output ProRNA3D_model/model.pt "https://zenodo.org/records/11477127/files/model.pt?download=1"
هذا كل شيء! Prorna3d-single جاهز للاستخدام.
1.) ضع البروتين و Monomers RNA (PDBs) داخل inputs/
2.) ضع التضمينات ESM2 داخل inputs/ (انظر مثال inputs/7ZLQB.rep_1280.npy ) ، ووضع التضمينات RNA-FM داخل inputs/ (انظر مثال هنا inputs/7ZLQC_RNA.npy ).
3.) ضع خرائط البروتين (C α -C α ) داخل prot_dist/ (انظر مثال prot_dist/7ZLQB_prot.dist ) ، ووضع RNA (C ' 4 -C' 4 ) خرائط المسافة داخل rna_dist/ (انظر مثال rna_dist/7ZLQC_RNA.c4p.dist ).
4.) ضع قائمة الأهداف في ملفات الملف inputs.list القائمة داخل inputs/ .
5.) الجري
python run_predictions.py
سيقوم هذا البرنامج النصي بتشغيل الاستدلال وإنشاء تفاعلات بين البروتين RNA داخل out_inter_rr/ . ثم سيحول التنبؤات إلى هياكل معقدة بروتين ثلاثي الأبعاد مطوي داخل predictions/
Datasets/ .