oleh Rahmatullah Roche, Sumit Tarafder, dan Debswapna Bhattacharya
[biorxiv] [pdf]
Basis kode untuk metode prediksi struktur kompleks protein-RNA-sequence kami, prorna3D-single.
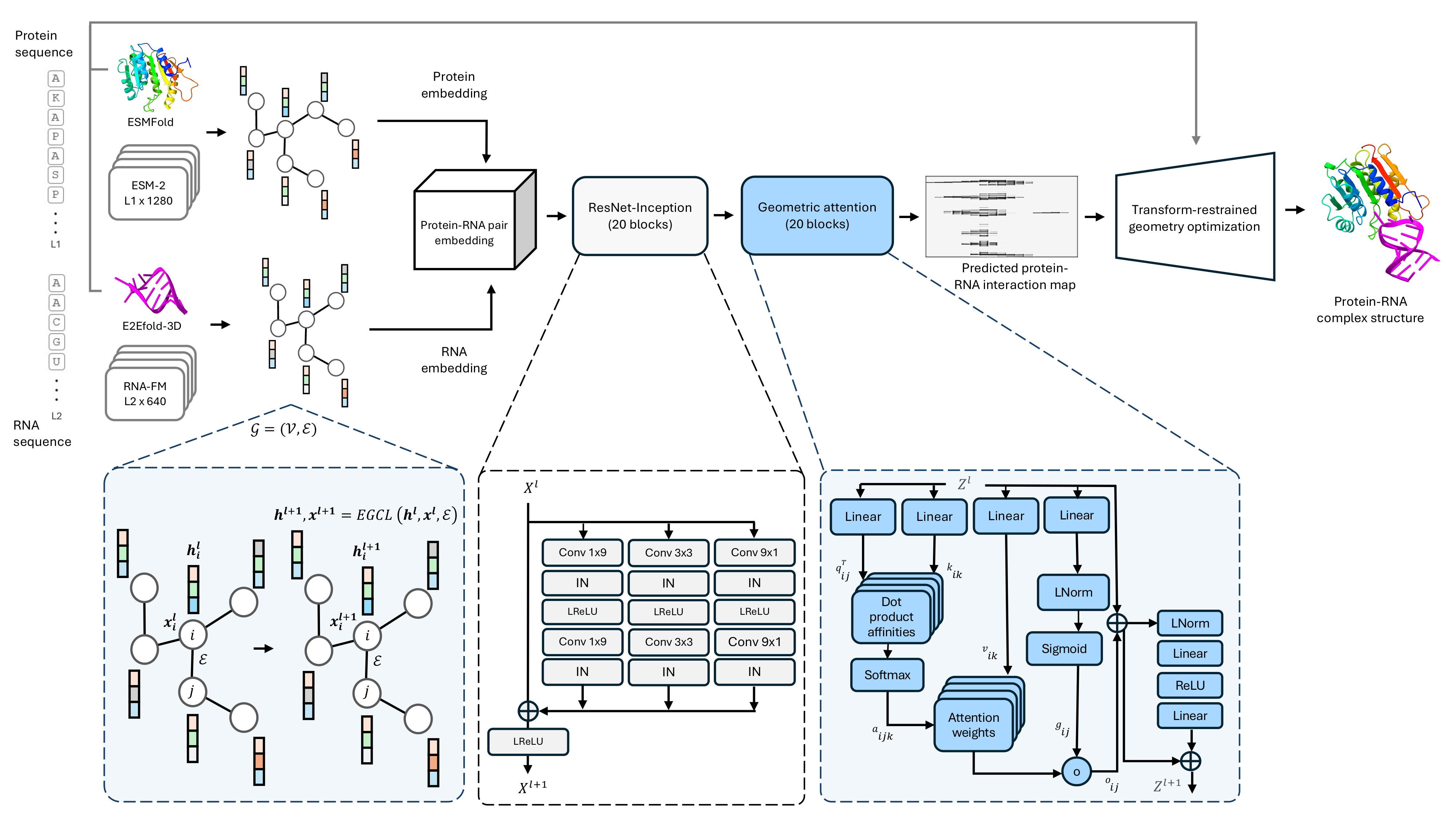
1.) Kami merekomendasikan lingkungan virtual Conda untuk menginstal dependensi untuk prorna3d-single. Perintah berikut akan menciptakan lingkungan virtual bernama 'prorna3d-single'
conda env create -f ProRNA3D-single_environment.yml
2.) Kemudian aktifkan lingkungan virtual
conda activate ProRNA3D-single
3.) Skrip lipat membutuhkan pyrosetta, yang dapat diinstal mengikuti instuksi ini.
4.) Unduh model terlatih dari sini dan tempatkan di dalam ProRNA3D_model/
curl --output ProRNA3D_model/model.pt "https://zenodo.org/records/11477127/files/model.pt?download=1"
Itu saja! Prorna3D-single siap digunakan.
1.) Tempatkan monomer protein dan RNA (PDB) di dalam inputs/
2.) Tempatkan embeddings ESM2 di dalam inputs/ (lihat contoh inputs/7ZLQB.rep_1280.npy ), dan tempatkan input RNA-FM di dalam inputs/ (lihat contoh di sini inputs/7ZLQC_RNA.npy ).
3.) Tempatkan peta jarak protein (c α -c α ) di dalam prot_dist/ (lihat contoh prot_dist/7ZLQB_prot.dist ), dan tempatkan peta jarak RNA (C ' 4 -C' 4 ) di dalam rna_dist/ (lihat contoh rna_dist/7ZLQC_RNA.c4p.dist ).
4.) Masukkan daftar target dalam inputs.list file. Daftar di dalam inputs/ .
5.) Jalankan
python run_predictions.py
Skrip ini akan menjalankan inferensi dan menghasilkan interaksi antar-protein-RNA di dalam out_inter_rr/ . Maka itu akan mengubah prediksi menjadi struktur kompleks protein-RNA 3D terlipat di dalam predictions/
Datasets/ .