Von Rahmatullah Roche, Sumit Tarafder und Debswapna Bhattacharya
[Biorxiv] [PDF]
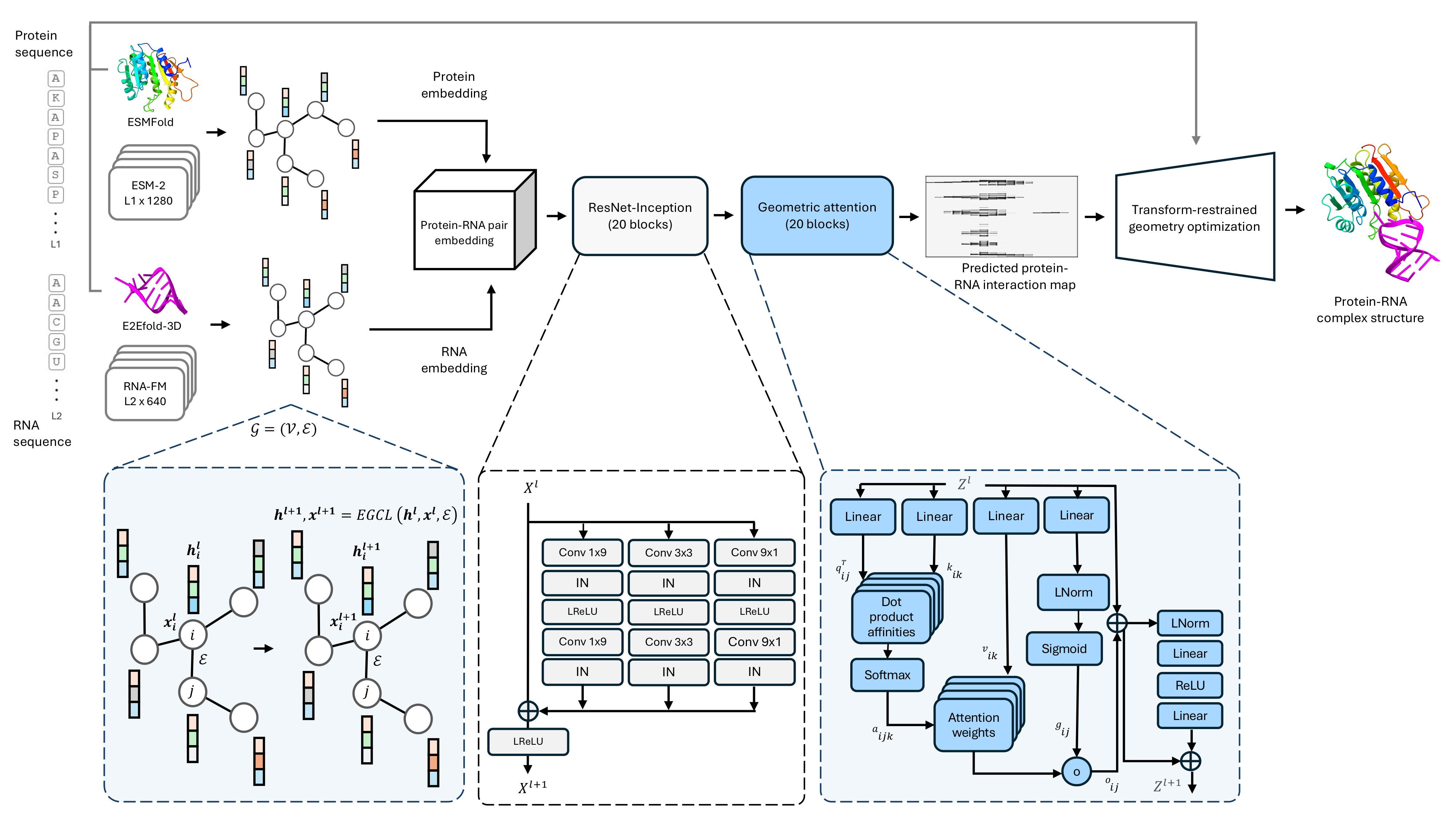
Codebasis für unsere Einzelsequenzprotein-RNA-Komplex-Struktur-Vorhersagemethode PRORNA3D-Single.
1.) Wir empfehlen Conda Virtual Environment, Abhängigkeiten für PRORNA3D-Single zu installieren. Der folgende Befehl erstellt eine virtuelle Umgebung mit dem Namen "PRORNA3D-Single"
conda env create -f ProRNA3D-single_environment.yml
2.) Dann die virtuelle Umgebung aktivieren
conda activate ProRNA3D-single
3.) Das Faltungsskript erfordert Pyrosetta, das nach diesen Instuktionen installiert werden kann.
4.) Laden Sie das trainierte Modell von hier herunter und platzieren Sie ProRNA3D_model/
curl --output ProRNA3D_model/model.pt "https://zenodo.org/records/11477127/files/model.pt?download=1"
Das war's! PRORNA3D-SINGLE ist zur Verwendung bereit.
1.) Legen Sie die Protein- und RNA -Monomere (PDBS) in inputs/
2.) Platzieren Sie die ESM2-Einbettungen in inputs/ (siehe Beispieleingänge inputs/7ZLQB.rep_1280.npy ) und platzieren Sie die RNA-FM-Einbettungen innerhalb inputs/ (siehe Beispiel hier inputs/7ZLQC_RNA.npy ).
3.) Platzieren Sie das Protein (c α -C α ) Distanzkarten in prot_dist/ (siehe Beispiel prot_dist/7ZLQB_prot.dist ) und platzieren Sie die RNA (C ' 4 -c' 4 ) Distanzkarten innerhalb von rna_dist/ (siehe Beispiel rna_dist/7ZLQC_RNA.c4p.dist ).
4.) Geben Sie die Liste der Ziele in die Dateieingänge inputs.list List in inputs/ .
5.) Laufen
python run_predictions.py
In diesem Skript werden Inferenz ausgeführt und interprotein-rNA-Interaktionen in out_inter_rr/ generiert. Dann verwandelt es die Vorhersagen in gefaltete 3D-Protein-RNA-Komplexstrukturen innerhalb predictions/ in die Falt-3D-Protein-RNA-Komplexstrukturen
Datasets/ verfügbar.