par Rahmatullah Roche, Sumit Tarafder et Debswapna Bhattacharya
[biorxiv] [PDF]
Base de code pour notre méthode de prédiction de la structure complexe de l'ARN de protéine à séquence à une seule séquence, Prorna3d-Single.
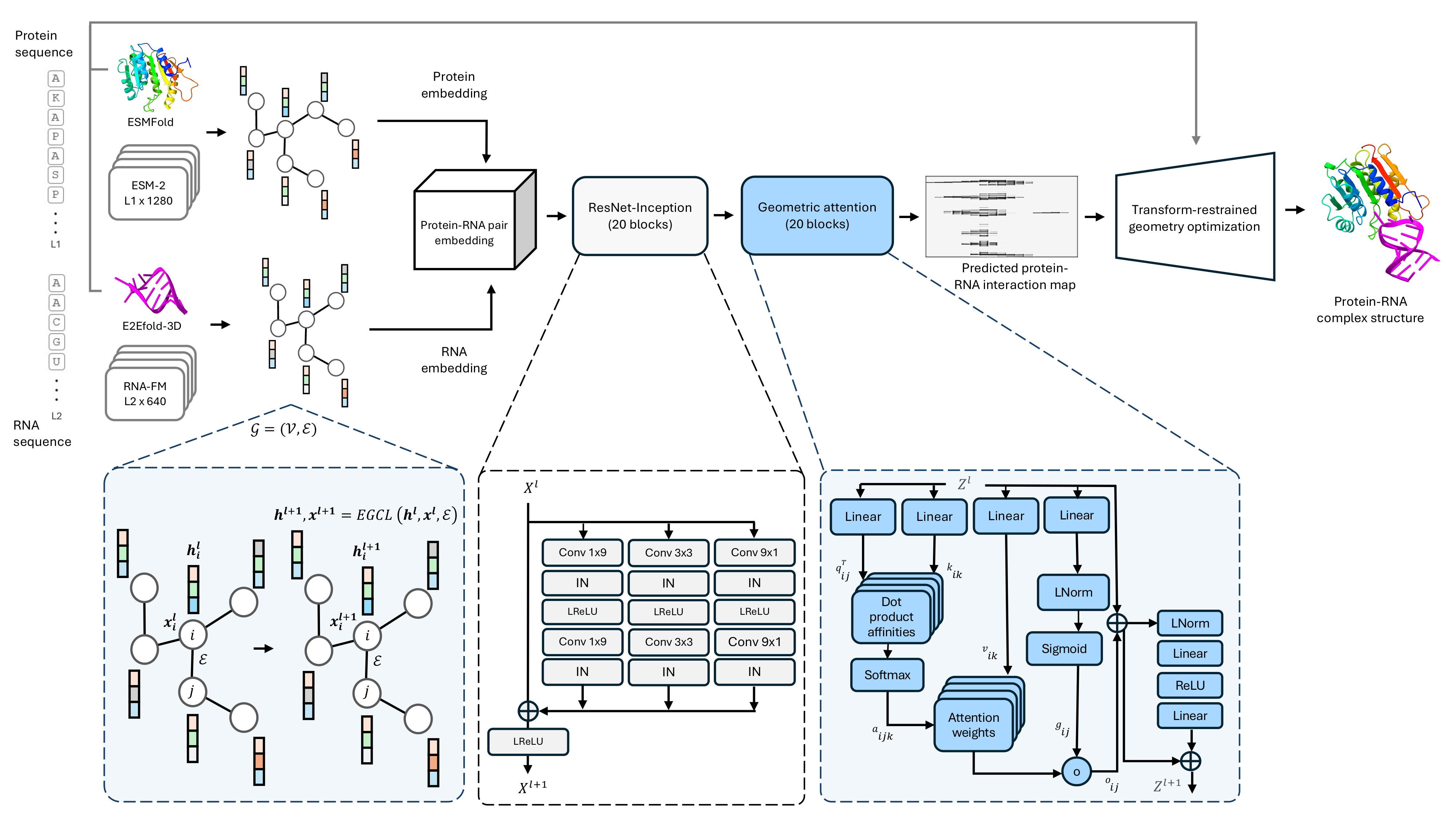
1.) Nous recommandons Conda Virtual Environment pour installer des dépendances pour Prorna3d-Single. La commande suivante créera un environnement virtuel nommé «Prorna3d-single»
conda env create -f ProRNA3D-single_environment.yml
2.) Ensuite, activez l'environnement virtuel
conda activate ProRNA3D-single
3.) Le script de pliage nécessite Pyrosetta, qui peut être installé après ces instuces.
4.) Téléchargez le modèle formé à partir d'ici et placez à l'intérieur ProRNA3D_model/
curl --output ProRNA3D_model/model.pt "https://zenodo.org/records/11477127/files/model.pt?download=1"
C'est ça! Prorna3d-single est prêt à être utilisé.
1.) Placer les monomères protéiques et ARN (PDBS) à l'intérieur inputs/
2.) Placez les intégres ESM2 à l'intérieur inputs/ (voir Exemple inputs/7ZLQB.rep_1280.npy ) et placez les incorporations RNA-FM à l'intérieur inputs/ (voir l'exemple ICI inputs/7ZLQC_RNA.npy ).
3.) Placer les cartes de distance de la protéine (C α -C α ) à l'intérieur prot_dist/ (voir exemple prot_dist/7ZLQB_prot.dist ), et placez les cartes de distance RNA (C ' 4 -C' 4 ) à l'intérieur rna_dist/ (voir exemple rna_dist/7ZLQC_RNA.c4p.dist ).
4.) Mettez la liste des cibles dans les inputs.list de fichiers. Liste à l'intérieur inputs/ .
5.) Courir
python run_predictions.py
Ce script exécutera l'inférence et générera des interactions inter-protéine-ARN à l'intérieur out_inter_rr/ . Ensuite, il transformera les prédictions en structures complexes de protéine 3D pliées à l'intérieur predictions/
Datasets/ .