Рахматулла Рош, Сумит Тарафдер и Дебсвапна Бхаттачарья
[biorxiv] [pdf]
Codebase для нашего метода прогнозирования структуры комплексной структуры в однопоследовании, PRORNA3D-Single.
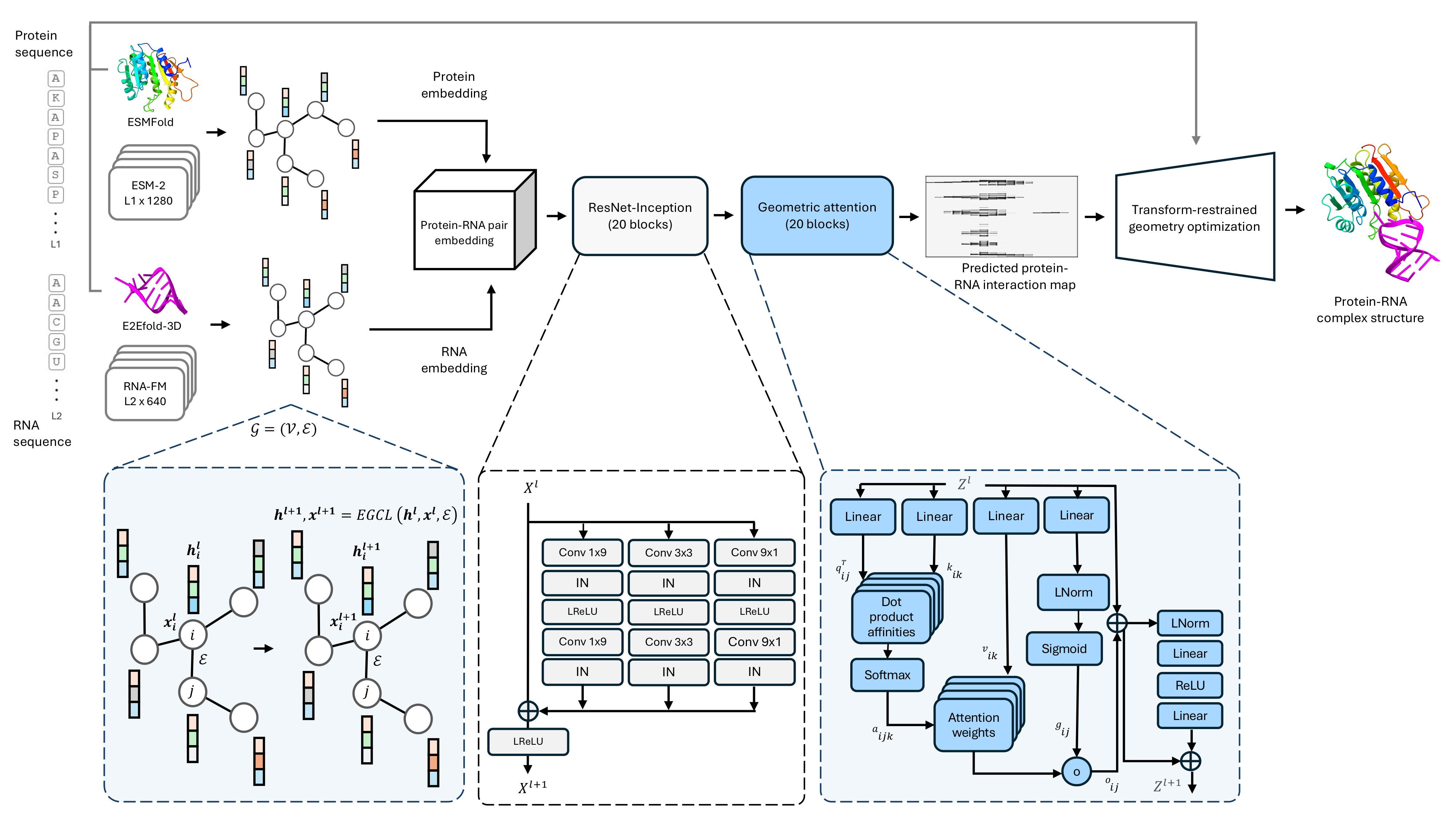
1.) Мы рекомендуем виртуальную среду Conda для установки зависимостей для Prorna3d-Single. Следующая команда создаст виртуальную среду с именем «prorna3d-single»
conda env create -f ProRNA3D-single_environment.yml
2.) Затем активируйте виртуальную среду
conda activate ProRNA3D-single
3.) Складной сценарий требует пирозетты, который может быть установлен после этих инстакций.
4.) Загрузите обученную модель отсюда и поместите в ProRNA3D_model/
curl --output ProRNA3D_model/model.pt "https://zenodo.org/records/11477127/files/model.pt?download=1"
Вот и все! Prorna3d-Single готов к использованию.
1.) Поместите мономеры белка и РНК (PDB) внутри inputs/
2.) Поместите вставки ESM2 внутри inputs/ (см. Пример inputs/7ZLQB.rep_1280.npy ) и поместите вставки RNA-FM внутри inputs/ (см. Пример здесь inputs/7ZLQC_RNA.npy ).
3.) Поместите карты расстояния белка (C α -C α ) внутри prot_dist/ (см. Пример prot_dist/7ZLQB_prot.dist ) и поместите РНК (C ' 4 -C' 4 ) Карты внутри rna_dist/ (см. Пример rna_dist/7ZLQC_RNA.c4p.dist ).
4.) Поместите список целей в файловые inputs.list Список внутренних inputs/ .
5.) запустить
python run_predictions.py
Этот скрипт будет выполнять вывод и генерировать взаимодействие между белковой РНК Inside out_inter_rr/ . Затем он преобразует прогнозы в сложенные трехмерные структуры белка-РНК внутри predictions/
Datasets/ .