Por Rahmatullah Roche, Sumit Tarafder e Debswapna Bhattacharya
[biorxiv] [pdf]
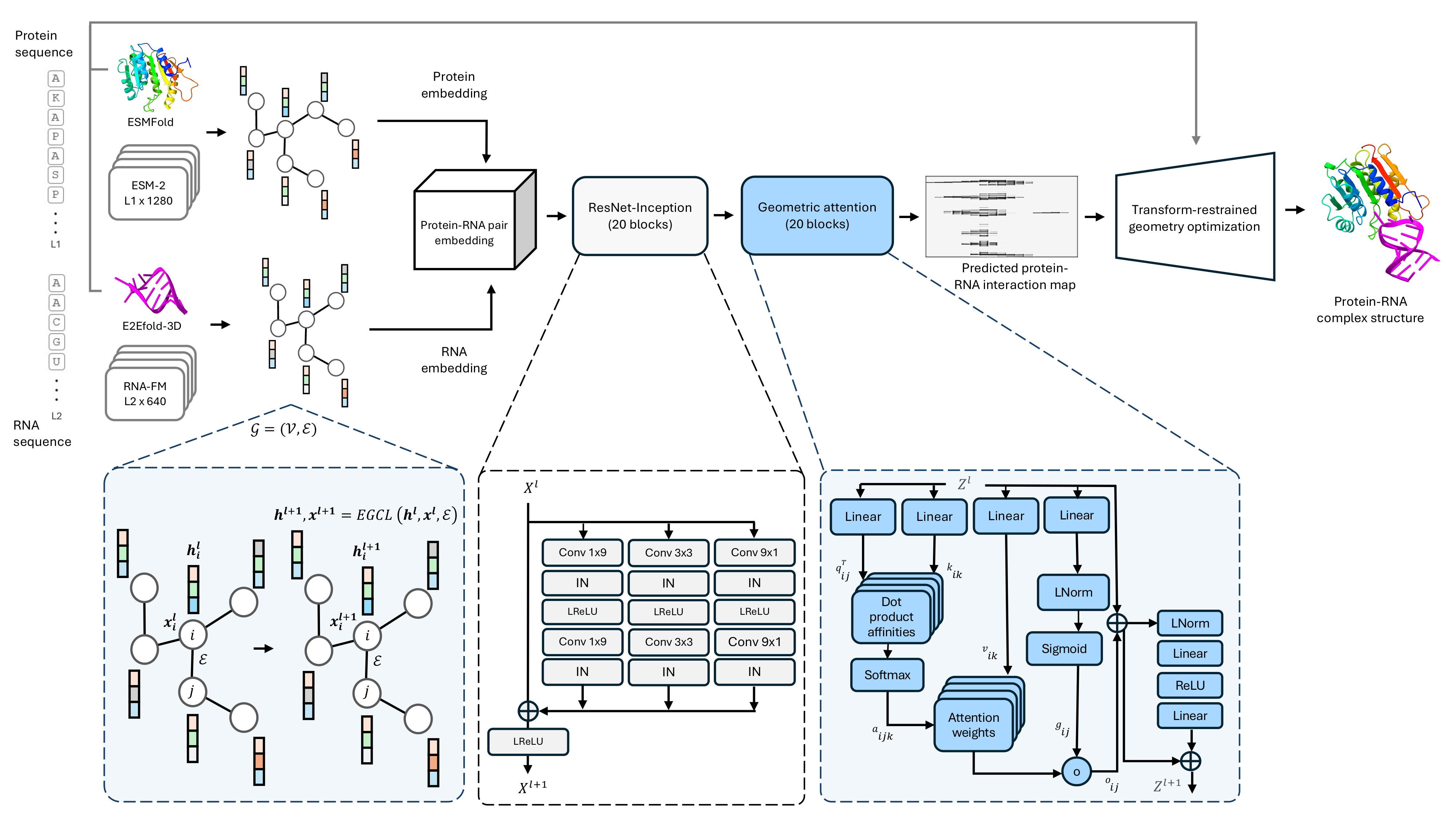
Base de código para o nosso método de previsão de estrutura de estrutura de proteína-RNA de sequência única, ProRNA3D-single.
1.) Recomendamos o ambiente virtual do CONDA para instalar dependências para o prorna3d-single. O comando a seguir criará um ambiente virtual chamado 'prorna3d-single'
conda env create -f ProRNA3D-single_environment.yml
2.) Em seguida, ative o ambiente virtual
conda activate ProRNA3D-single
3.) O script dobrável requer pirosetta, que pode ser instalado seguindo estas instituições.
4.) Faça o download do modelo treinado daqui e coloque dentro ProRNA3D_model/
curl --output ProRNA3D_model/model.pt "https://zenodo.org/records/11477127/files/model.pt?download=1"
É isso! Prorna3d-single está pronto para ser usado.
1.) Coloque os monômeros de proteína e RNA (PDBs) dentro de inputs/
2.) Coloque as incorporações ESM2 dentro inputs/ (consulte Exemplo inputs/7ZLQB.rep_1280.npy ) e coloque as incorporações RNA-fm dentro inputs/ (consulte o exemplo aqui inputs/7ZLQC_RNA.npy ).
3.) Coloque os mapas de distância da proteína (C α -C α ) dentro prot_dist/ (consulte o exemplo prot_dist/7ZLQB_prot.dist ) e coloque os mapas de distância RNA (c ' 4 -c' 4 ) dentro rna_dist/ (consulte o exemplo rna_dist/7ZLQC_RNA.c4p.dist ).
4.) Coloque a lista de metas na inputs.list do arquivo. Lista interna inputs/ .
5.) Execute
python run_predictions.py
Este script executará a inferência e gerará interações entre proteínas-RNA dentro out_inter_rr/ . Em seguida, ele transformará as previsões em estruturas de complexo proteico 3D dobradas dentro de predictions/
Datasets/ .