Unificar representações moleculares e textuais via modelagem de idiomas com várias tarefas
Dimitrios Christofidellis*, Giorgio Giannone*, Jannis nascido, Ole Winther, Teodoro Laino, Matteo Manica
Conferência Internacional sobre Machine Learning (ICML), 2023
[Paper] [App Gradio] [Código]
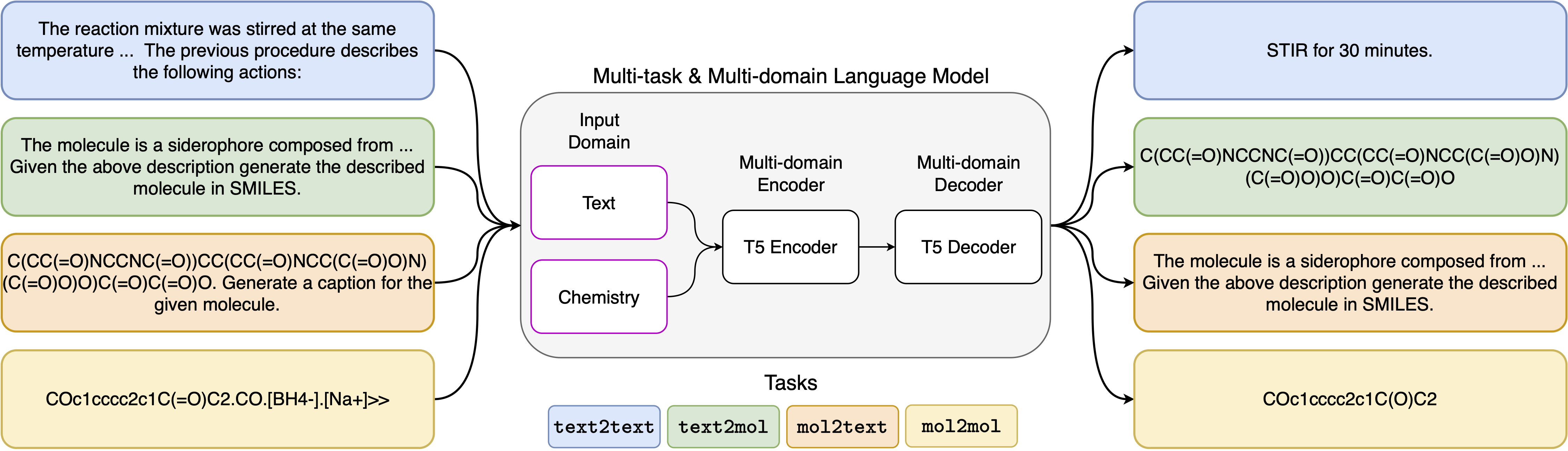
Os recentes avanços nos modelos de linguagem neural também foram aplicados com sucesso ao campo da química, oferecendo soluções generativas para problemas clássicos no projeto molecular e no planejamento da síntese. Esses novos métodos têm o potencial de alimentar uma nova era de automação orientada a dados na descoberta científica. No entanto, modelos especializados ainda são normalmente necessários para cada tarefa, levando à necessidade de ajuste fino específico e negligenciando inter-relações de tarefas. O principal obstáculo nesse campo é a falta de uma representação unificada entre linguagem natural e representações químicas, complicando e limitando a interação humana-máquina. Aqui, propomos o primeiro modelo de idioma de vários domínios e várias tarefas que pode resolver uma ampla gama de tarefas nos domínios de linguagem química e natural. Nosso modelo pode lidar com linguagem química e natural simultaneamente, sem exigir pré-treinamento caro em domínios únicos ou modelos específicos de tarefas. Curiosamente, o compartilhamento de pesos nos domínios melhora notavelmente nosso modelo quando comparado a linhas de base de última geração em tarefas de domínio único e domínio cruzado. Em particular, o compartilhamento de informações entre domínios e tarefas gera grandes melhorias nas tarefas de domínio cruzado, cuja magnitude aumenta com a escala, conforme medido por mais de uma dúzia de métricas relevantes. Nosso trabalho sugere que esses modelos podem acelerar de maneira robusta e eficiente a descoberta em ciências físicas, substituindo o ajuste fino específico do problema e aumentando as interações humanos-modelos .
Instalar requisitos:
pip install -r requirements.txtCrie um kernel dedicado:
python -m ipykernel install --name text_chem_t5_demoBom ir
O processo de treinamento é realizado usando o instrutor de modelagem de idiomas com base em abraçadores de transformadores de rosto (Wolf et al., 2020) e Pytorch Lightning (Falcon e a equipe de Pytorch Lightning, 2019) da biblioteca GT4SD (Manica et al., 2022). Para reproduzir o treinamento, você precisa primeiro instalar a biblioteca GT4SD. Para obter mais informações sobre o processo de instalação da biblioteca GT4SD, você pode visitar sua página. Depois que o GT4SD estiver instalado, você pode usar o seguinte comando para iniciar nosso treinamento. Observe que as divisões de conjunto de dados fornecidas no diretório dataset-sample contêm apenas um pequeno subconjunto de nossas divisões reais do conjunto de dados. Para regenerar nosso conjunto de dados de treinamento completo, encaminhamos o leitor interessado para a respectiva seção de nosso artigo e as referências que são fornecidas lá.
gt4sd-trainer --training_pipeline_name language-modeling-trainer
--model_name_or_path t5-base
--lr 6e-4
--lr_decay 0.99
--batch_size 8
--train_file dataset-sample/train.jsonl
--validation_file dataset-sample/valid.jsonl
--default_root_dir text_chem_t5_base
--type cgm
--val_check_interval 20000
--max_epochs 1
--limit_val_batches 500
--accumulate_grad_batches 4
--log_every_n_steps 5000
--monitor val_loss
--save_top_k 1
--mode min
--every_n_train_steps 20000
--accelerator 'ddp'
Os modelos de prompt que usamos para as 5 tarefas diferentes podem ser encontrados na tabela a seguir, onde o <input> representa a entrada real para cada tarefa.
| Tarefa | Modelo |
|---|---|
| Previsão para a frente | Preveja o produto da seguinte reação: <input> |
| Retrossíntese | Preveja a reação que produz o seguinte produto: <input> |
| Parágrafo a ações | Quais ações são descritas no seguinte parágrafo: <input> |
| Descrição para sorrir | Escreva em sorrisos a molécula descrita: <input> |
| Sorrisos para a captura | Legenda os seguintes sorrisos: <input> |
As quatro variantes do nosso modelo estão disponíveis no hub Huggignface nos links a seguir:
No notebook fornecido (Demo.ipynb), apresentamos exemplos de como o modelo pode ser usado para as 5 tarefas diferentes.
@inproceedings { christofidellis2023unifying ,
title = { Unifying Molecular and Textual Representations via Multi-task Language Modelling } ,
author = { Christofidellis, Dimitrios and Giannone, Giorgio and Born, Jannis and Winther, Ole and Laino, Teodoro and Manica, Matteo } ,
booktitle = { Proceedings of the 40th International Conference on Machine Learning } ,
pages = { 6140--6157 } ,
year = { 2023 } ,
volume = { 202 } ,
series = { Proceedings of Machine Learning Research } ,
publisher = { PMLR } ,
pdf = { https://proceedings.mlr.press/v202/christofidellis23a/christofidellis23a.pdf } ,
url = { https://proceedings.mlr.press/v202/christofidellis23a.html } ,
}