Représentations moléculaires et textuelles unificatrices via la modélisation de la langue multi-tâches
Dimitrios Christofidellis *, Giorgio Giannone *, Jannis Born, Ole Winter, Teodoro Laino, Matteo Manica
Conférence internationale sur l'apprentissage automatique (ICML), 2023
[Paper] [Application Gradio] [Code]
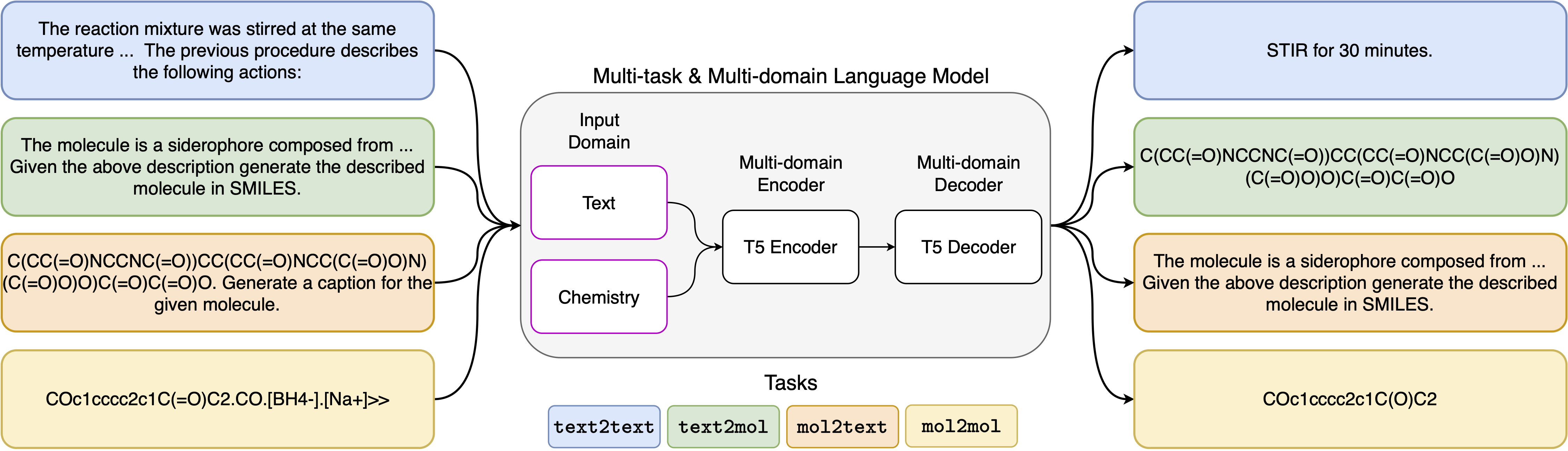
Les progrès récents des modèles de langage neuronal ont également été appliqués avec succès dans le domaine de la chimie, offrant des solutions génératives pour les problèmes classiques dans la conception moléculaire et la planification de la synthèse. Ces nouvelles méthodes peuvent alimenter une nouvelle ère d'automatisation basée sur les données dans la découverte scientifique. Cependant, des modèles spécialisés sont encore généralement requis pour chaque tâche, conduisant à la nécessité de régler les interrelations de tâches et de négligence spécifiques au problème. Le principal obstacle dans ce domaine est l'absence d'une représentation unifiée entre le langage naturel et les représentations chimiques, compliquant et limitant l'interaction humaine-machine. Ici, nous proposons le premier modèle de langue multi-domaines multi-tâches qui peut résoudre un large éventail de tâches dans les domaines chimiques et en langage naturel. Notre modèle peut gérer simultanément le langage chimique et naturel, sans nécessiter une pré-formation coûteuse sur des domaines uniques ou des modèles spécifiques à des tâches. Fait intéressant, le partage de poids dans les domaines améliore remarquablement notre modèle lorsqu'il est compatible contre les lignes de base de pointe sur des tâches à domaine unique et inter-domaines. En particulier, le partage d'informations entre les domaines et les tâches donne lieu à de grandes améliorations des tâches intermédiaires, dont l'ampleur augmente avec l'échelle, comme mesuré par plus d'une douzaine de mesures pertinentes. Nos travaux suggèrent que de tels modèles peuvent accélérer de manière robuste et efficiente la découverte en sciences physiques en remplaçant les interactions fins et améliorant des interactions de modes humains spécifiques au problème .
Installation des exigences:
pip install -r requirements.txtCréez un noyau dédié:
python -m ipykernel install --name text_chem_t5_demoRavi d'y aller
Le processus de formation est réalisé à l'aide du formateur de modélisation du langage en fonction des transformateurs de visage étreintes (Wolf et al., 2020) et de Pytorch Lightning (Falcon et The Pytorch Lightning Team, 2019) de la bibliothèque GT4SD (Manica et al., 2022). Pour reproduire la formation, vous devez d'abord installer la bibliothèque GT4SD. Pour plus d'informations concernant le processus d'installation de la bibliothèque GT4SD, vous pouvez visiter sa page. Une fois GT4SD installé, vous pouvez utiliser la commande suivante pour lancer notre formation. Notez que l'ensemble de données fourni dans le répertoire dataset-sample ne contiennent qu'un petit sous-ensemble de nos divisions réelles de données de données. Pour régénérer notre ensemble de données de formation complet, nous renvoyons le lecteur intéressé à la section respective de notre article et aux références qui y sont fournies.
gt4sd-trainer --training_pipeline_name language-modeling-trainer
--model_name_or_path t5-base
--lr 6e-4
--lr_decay 0.99
--batch_size 8
--train_file dataset-sample/train.jsonl
--validation_file dataset-sample/valid.jsonl
--default_root_dir text_chem_t5_base
--type cgm
--val_check_interval 20000
--max_epochs 1
--limit_val_batches 500
--accumulate_grad_batches 4
--log_every_n_steps 5000
--monitor val_loss
--save_top_k 1
--mode min
--every_n_train_steps 20000
--accelerator 'ddp'
Les modèles d'invite que nous avons utilisés pour les 5 tâches différentes peuvent être trouvés dans le tableau suivant, où <port> représente l'entrée réelle pour chaque tâche.
| Tâche | Modèle |
|---|---|
| Prédiction vers l'avant | Prédire le produit de la réaction suivante: <fort> |
| Rétrosynthèse | Prédire la réaction qui produit le produit suivant: <fort> |
| Paragraphe aux actions | Quelles actions sont décrites dans le paragraphe suivant: <fort> |
| Description à SMILS | Écrivez en sourit la molécule décrite: <fort> |
| Sourires à caption | Légende les sourires suivants: <fort> |
Les quatre variantes de notre modèle sont disponibles via le Huggignface Hub dans les liens suivants:
Dans le cahier fourni (Demo.ipynb), nous présentons des exemples de la façon dont le modèle peut être utilisé pour les 5 tâches différentes.
@inproceedings { christofidellis2023unifying ,
title = { Unifying Molecular and Textual Representations via Multi-task Language Modelling } ,
author = { Christofidellis, Dimitrios and Giannone, Giorgio and Born, Jannis and Winther, Ole and Laino, Teodoro and Manica, Matteo } ,
booktitle = { Proceedings of the 40th International Conference on Machine Learning } ,
pages = { 6140--6157 } ,
year = { 2023 } ,
volume = { 202 } ,
series = { Proceedings of Machine Learning Research } ,
publisher = { PMLR } ,
pdf = { https://proceedings.mlr.press/v202/christofidellis23a/christofidellis23a.pdf } ,
url = { https://proceedings.mlr.press/v202/christofidellis23a.html } ,
}