Unificar representaciones moleculares y textuales a través del modelado de lenguaje de varias tareas
Dimitrios Christofidellis*, Giorgio Giannone*, Jannis nacido, Ole Winther, Teodoro Laino, Matteo Manica
Conferencia internacional sobre aprendizaje automático (ICML), 2023
[Paper] [Aplicación Gradio] [Código]
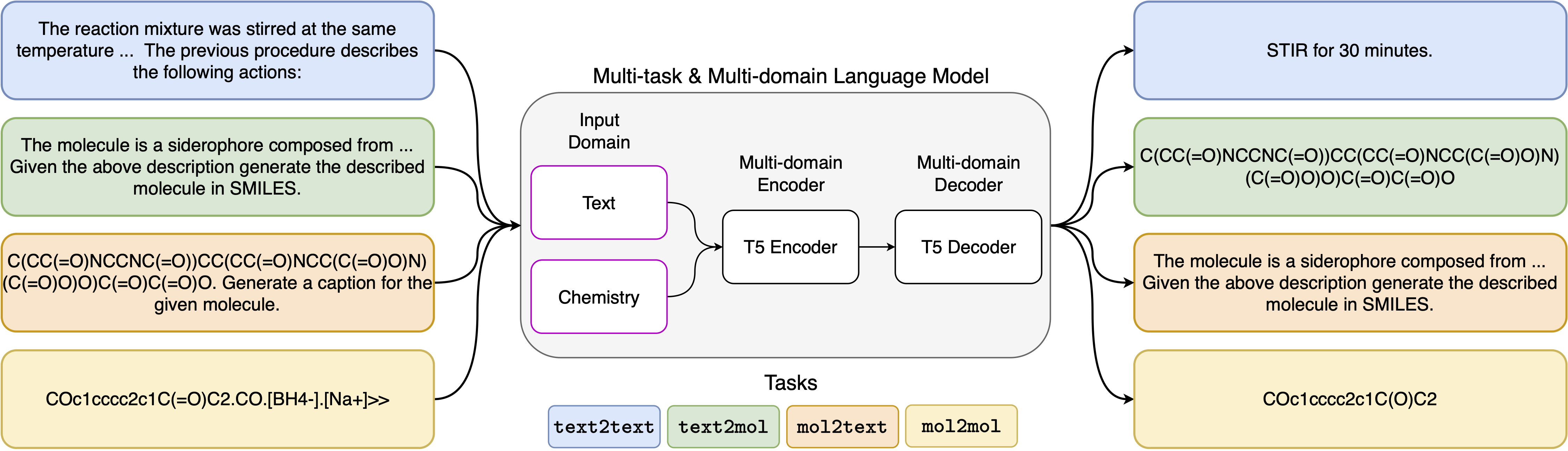
Los avances recientes en los modelos de lenguaje neuronal también se han aplicado con éxito al campo de la química, ofreciendo soluciones generativas para problemas clásicos en diseño molecular y planificación de síntesis. Estos nuevos métodos tienen el potencial de alimentar una nueva era de automatización basada en datos en el descubrimiento científico. Sin embargo, los modelos especializados todavía se requieren para cada tarea, lo que lleva a la necesidad de ajustar y descuidar las interrelaciones de la tarea de ajuste y descuido del problema. El principal obstáculo en este campo es la falta de una representación unificada entre el lenguaje natural y las representaciones químicas, complicando y limitando la interacción humana-máquina. Aquí, proponemos el primer modelo de lenguaje de varias tareas múltiples que puede resolver una amplia gama de tareas en los dominios del lenguaje químico y natural. Nuestro modelo puede manejar el lenguaje químico y natural simultáneamente, sin requerir un pre-entrenamiento costoso en dominios individuales o modelos específicos de tareas. Curiosamente, compartir pesos en todos los dominios mejora notablemente nuestro modelo cuando se comparó con líneas de base de última generación en tareas de dominio único y dominio cruzado. En particular, compartir información entre dominios y tareas da lugar a grandes mejoras en las tareas de dominio cruzado, cuya magnitud aumenta con la escala, medida por más de una docena de métricas relevantes. Nuestro trabajo sugiere que dichos modelos pueden acelerar de manera robusta y eficiente el descubrimiento en ciencias físicas mediante el reemplazo del ajuste fino específico de los problemas y la mejora de las interacciones del modelo humano .
Requisitos de instalación:
pip install -r requirements.txtCrea un kernel dedicado:
python -m ipykernel install --name text_chem_t5_demoBien para ir
El proceso de capacitación se lleva a cabo utilizando el entrenador de modelado de idiomas basado en los transformadores faciales para abrazos (Wolf et al., 2020) y Pytorch Lightning (Falcon y el equipo de Pytorch Lightning, 2019) de la biblioteca GT4SD (Manica et al., 2022). Para reproducir el entrenamiento, primero debe instalar la biblioteca GT4SD. Para obtener más información sobre el proceso de instalación de la biblioteca GT4SD, puede visitar su página. Una vez que se instala GT4SD, puede usar el siguiente comando para iniciar nuestra capacitación. Tenga en cuenta que las divisiones de conjunto de datos proporcionadas en el directorio dataset-sample contienen solo un pequeño subconjunto de nuestras divisiones reales del conjunto de datos. Para regenerar nuestro conjunto de datos de capacitación completa, remitimos al lector interesado a la sección respectiva de nuestro documento y las referencias que se proporcionan allí.
gt4sd-trainer --training_pipeline_name language-modeling-trainer
--model_name_or_path t5-base
--lr 6e-4
--lr_decay 0.99
--batch_size 8
--train_file dataset-sample/train.jsonl
--validation_file dataset-sample/valid.jsonl
--default_root_dir text_chem_t5_base
--type cgm
--val_check_interval 20000
--max_epochs 1
--limit_val_batches 500
--accumulate_grad_batches 4
--log_every_n_steps 5000
--monitor val_loss
--save_top_k 1
--mode min
--every_n_train_steps 20000
--accelerator 'ddp'
Las plantillas de solicitud que hemos utilizado para las 5 tareas diferentes se pueden encontrar en la siguiente tabla, donde <put> representa la entrada real para cada tarea.
| Tarea | Plantilla |
|---|---|
| Predicción hacia adelante | Predecir el producto de la siguiente reacción: <put> |
| Retrosínesis | Predecir la reacción que produce el siguiente producto: <put> |
| Párrafo a las actividades | Qué acciones se describen en el siguiente párrafo: <put> |
| Descripción a las suaves | Escribe en Smiles la molécula descrita: <put> |
| Sonrisas a la toma | Leyenda las siguientes sonrisas: <put> |
Las cuatro variantes de nuestro modelo están disponibles a través del Huggignface Hub en los siguientes enlaces:
En el cuaderno proporcionado (demo.ipynb), presentamos ejemplos de cómo el modelo se puede usar para las 5 tareas diferentes.
@inproceedings { christofidellis2023unifying ,
title = { Unifying Molecular and Textual Representations via Multi-task Language Modelling } ,
author = { Christofidellis, Dimitrios and Giannone, Giorgio and Born, Jannis and Winther, Ole and Laino, Teodoro and Manica, Matteo } ,
booktitle = { Proceedings of the 40th International Conference on Machine Learning } ,
pages = { 6140--6157 } ,
year = { 2023 } ,
volume = { 202 } ,
series = { Proceedings of Machine Learning Research } ,
publisher = { PMLR } ,
pdf = { https://proceedings.mlr.press/v202/christofidellis23a/christofidellis23a.pdf } ,
url = { https://proceedings.mlr.press/v202/christofidellis23a.html } ,
}