Pyoptim: pacote Python para operações de otimização e matriz
Este projeto, um componente do curso de análise e otimização numérica em Ensias, Mohammed V University, surgiu da sugestão do professor M. Naoum de criar um pacote Python que encapsulou algoritmos praticados durante as sessões de laboratório. Ele se concentra em cerca de dez algoritmos de otimização sem restrições, oferecendo visualizações 2D e 3D, permitindo uma comparação de desempenho na eficiência e precisão computacional. Além disso, o projeto abrange operações de matriz, como inversão, decomposição e resolução de sistemas lineares, todos integrados no pacote que contém os algoritmos derivados do laboratório.
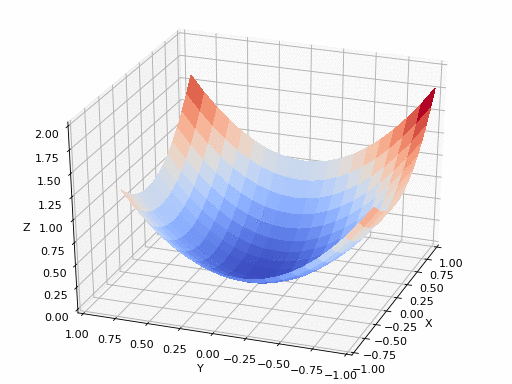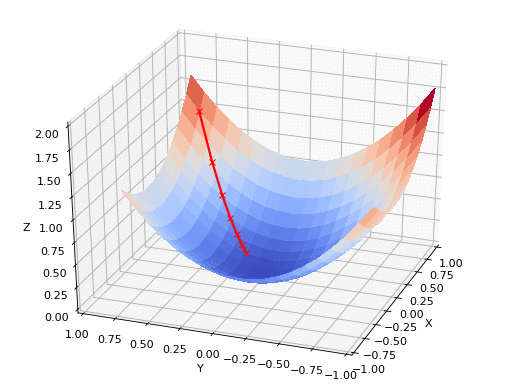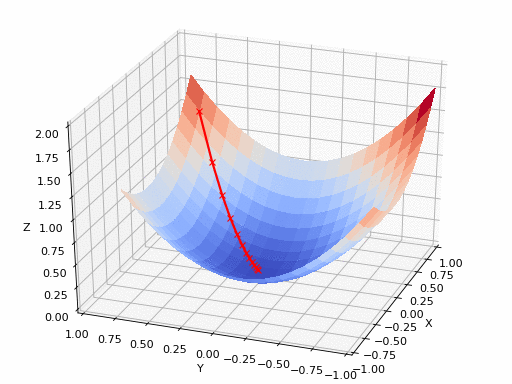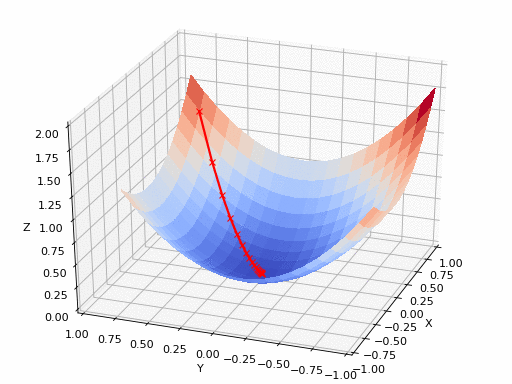
Fonte do GIF: https://www.nsf.gov/news/mmg/mmg_disp.jsp?med_id=78950&from= ](https://aria42.com/blog/2014/12/undestanding-lbfgs
Cada parte deste projeto é o código de exemplo, que mostra como fazer o seguinte:
Implementação de algoritmos de otimização de uma variável, abrangendo fixo_step, acelerado_step, exaustivo_search, dichotomous_search, interval_halving, fibonacci, Golden_section, armijo_backward e Armijo_forward.
Incorporação de vários algoritmos de otimização unidiriável, incluindo descida de gradiente, conjugado de gradiente, newton, quase_newton_dfp, descida de gradiente estocástico.
Visualização de cada etapa de iteração para todos os algoritmos nos formatos 2D, contorno e 3D.
Conduzindo uma análise comparativa com foco em suas métricas de tempo de execução e precisão.
Implementação de operações de matriz, como inversão (por exemplo, gauss-jordan), decomposição (por exemplo, lu, choleski) e soluções para sistemas lineares (por exemplo, Gauss-Jordan, Lu, Choleski).
Para instalar este pacote Python:
pip install pyoptimExecute este caderno de demonstração
import pyoptim . my_scipy . onevar_optimize . minimize as soom
import pyoptim . my_plot . onevar . _2D as po2 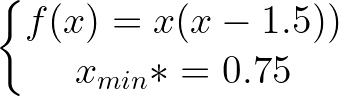
def f ( x ):
return x * ( x - 1.5 ) # analytically, argmin(f) = 0.75 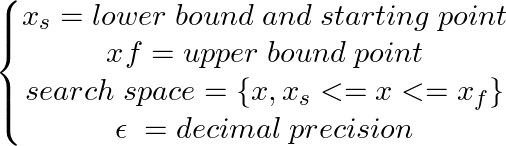
xs = - 10
xf = 10
epsilon = 1.e-2 print ( 'x* =' , soom . fixed_step ( f , xs , epsilon ))
po2 . fixed_step ( f , xs , epsilon ) x* = 0.75 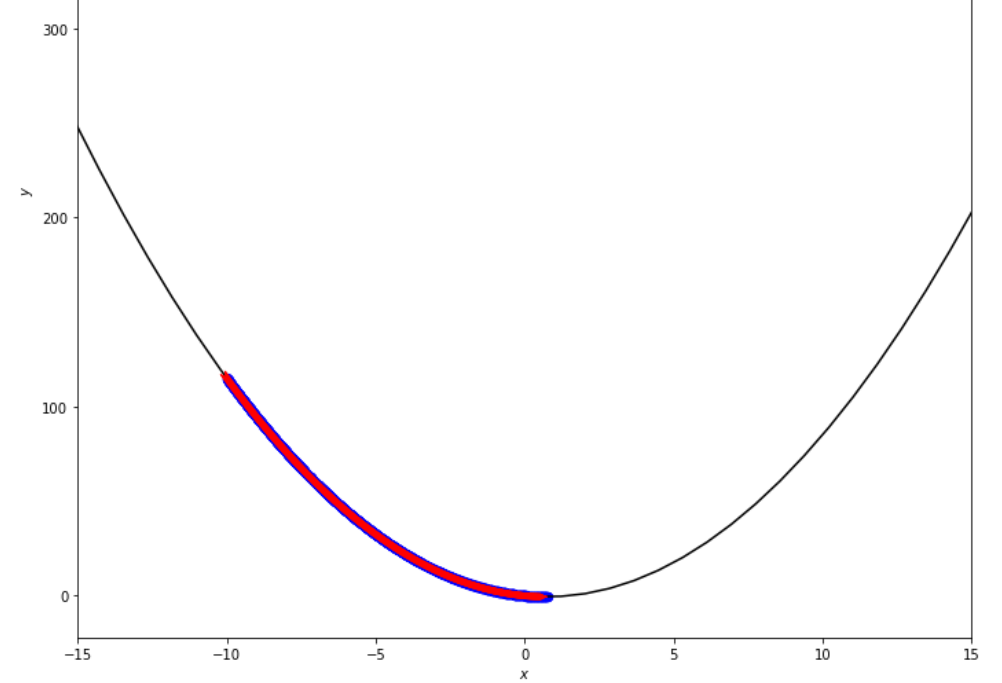
A sequência de medidas tomadas pelo algoritmo de etapa fixa antes de atingir o mínimo
print ( 'x* =' , soom . accelerated_step ( f , xs , epsilon ))
po2 . accelerated_step ( f , xs , epsilon ) x* = 0.86 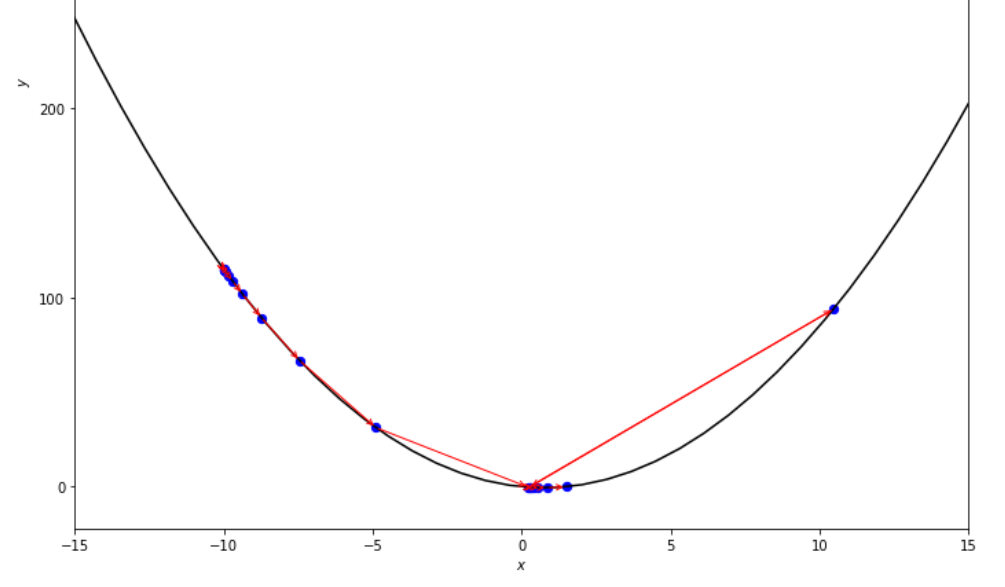
A sequência de medidas tomadas pelo algoritmo de etapa acelerado antes de atingir o mínimo
print ( 'x* =' , soom . exhaustive_search ( f , xs , xf , epsilon ))
po2 . exhaustive_search ( f , xs , xf , epsilon ) x* = 0.75
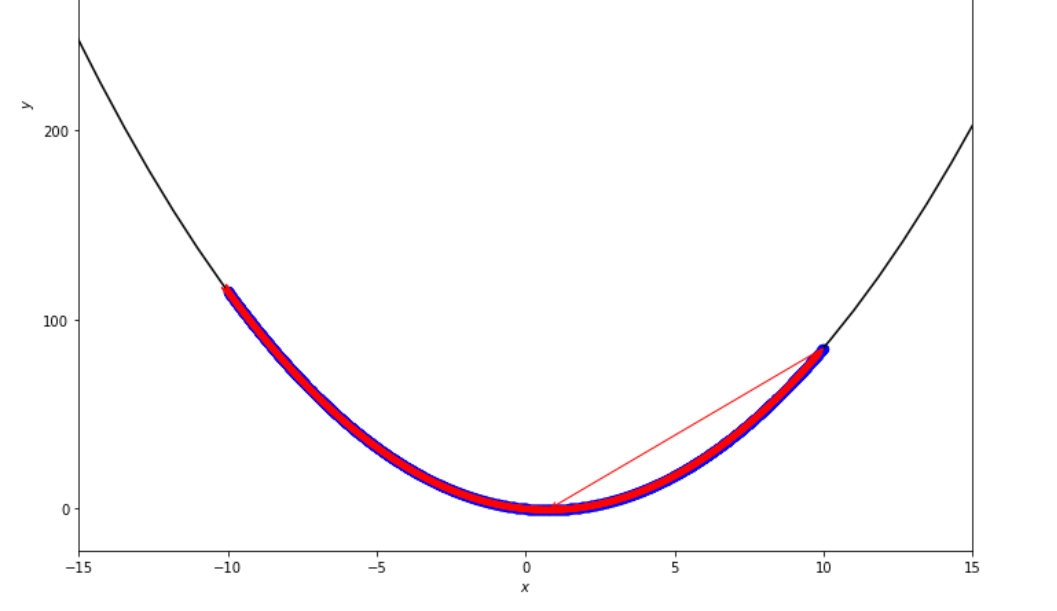
A sequência de medidas tomadas pelo algoritmo de pesquisa exaustiva antes de atingir o mínimo
mini_delta = 1.e-3
print ( 'x* =' , soom . dichotomous_search ( f , xs , xf , epsilon , mini_delta ))
po2 . dichotomous_search ( f , xs , xf , epsilon , mini_delta ) x* = 0.7494742431640624
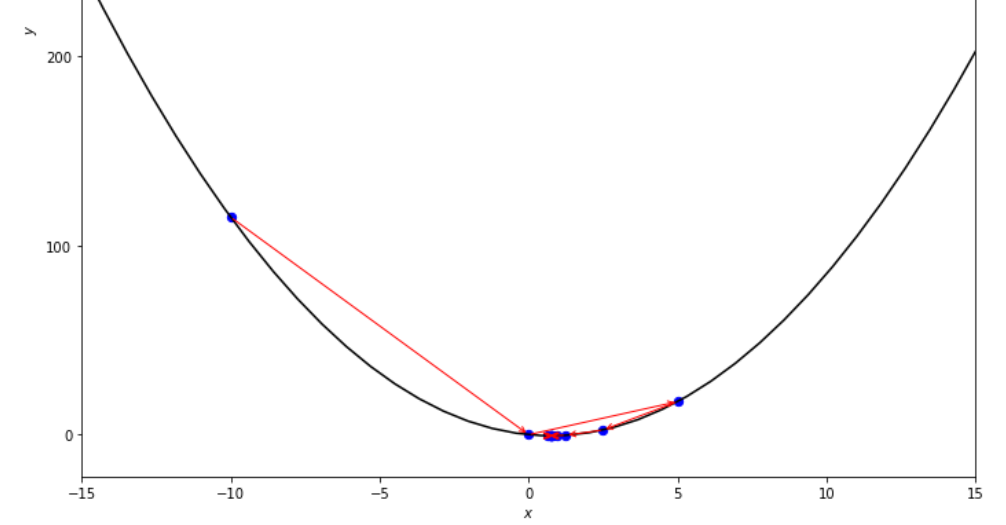
A sequência de medidas tomadas pelo algoritmo de pesquisa dicotômica antes de atingir o mínimo
print ( 'x* =' , soom . interval_halving ( f , xs , xf , epsilon ))
po2 . interval_halving ( f , xs , xf , epsilon ) x* = 0.75
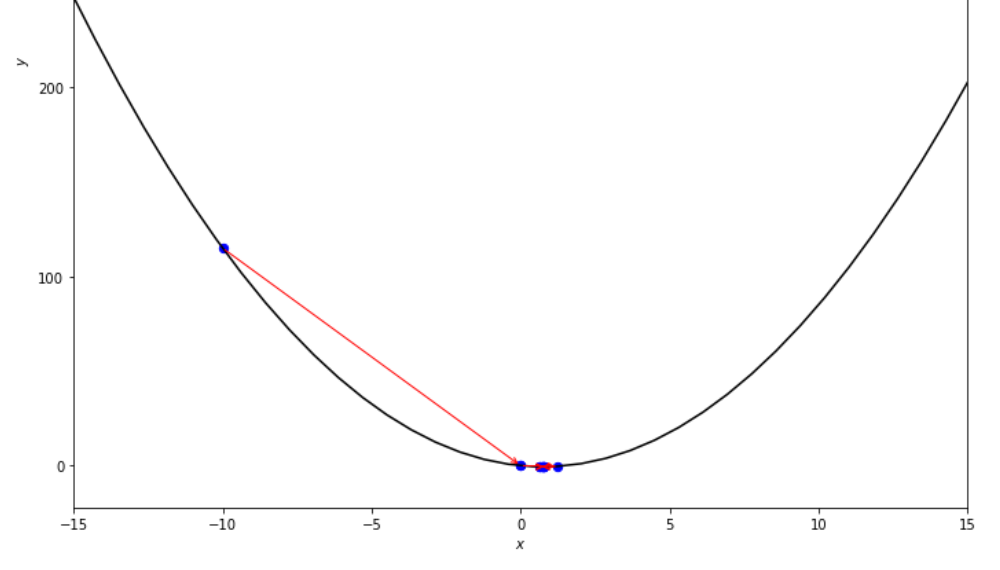
A sequência de etapas tomadas pelo algoritmo intervalo pela metade antes de atingir o mínimo.
n = 15
print ( 'x* =' , soom . fibonacci ( f , xs , xf , n ))
po2 . fibonacci ( f , xs , xf , n ) x* = 0.76 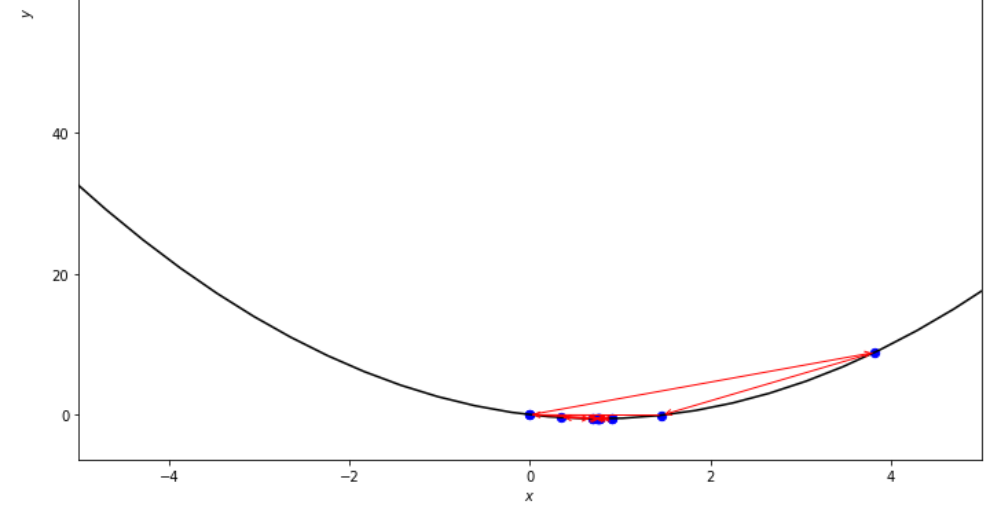
A sequência de etapas tomadas pelo algoritmo Fibonacci antes de atingir o mínimo.
print ( 'x* =' , soom . golden_section ( f , xs , xf , epsilon ))
po2 . golden_section ( f , xs , xf , epsilon ) x* = 0.75 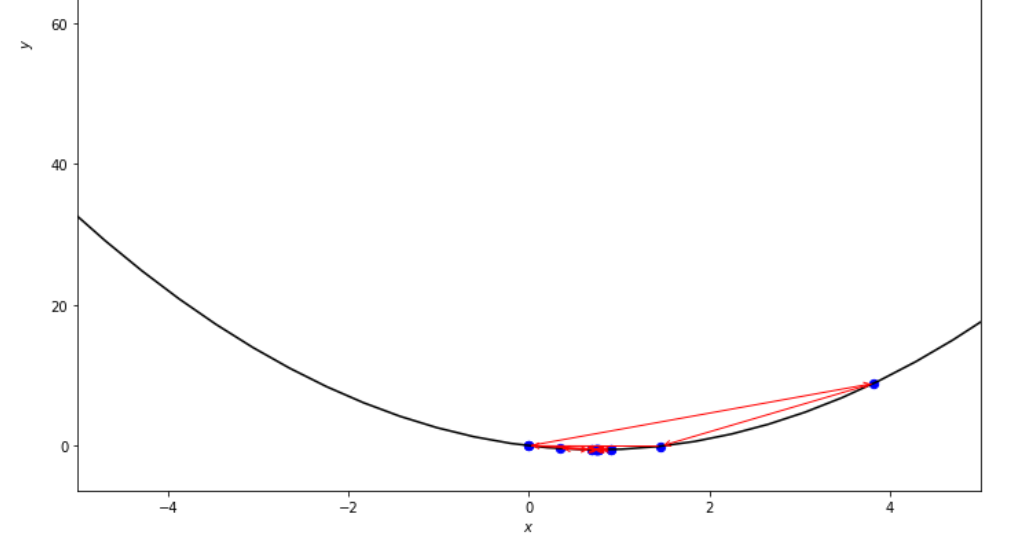
A sequência de medidas tomadas pelo algoritmo da seção de ouro antes de atingir o mínimo
ŋ = 2
xs = 100
print ( 'x* =' , soom . armijo_backward ( f , xs , ŋ , epsilon ))
po2 . armijo_backward ( f , xs , ŋ , epsilon ) x* = 0.78 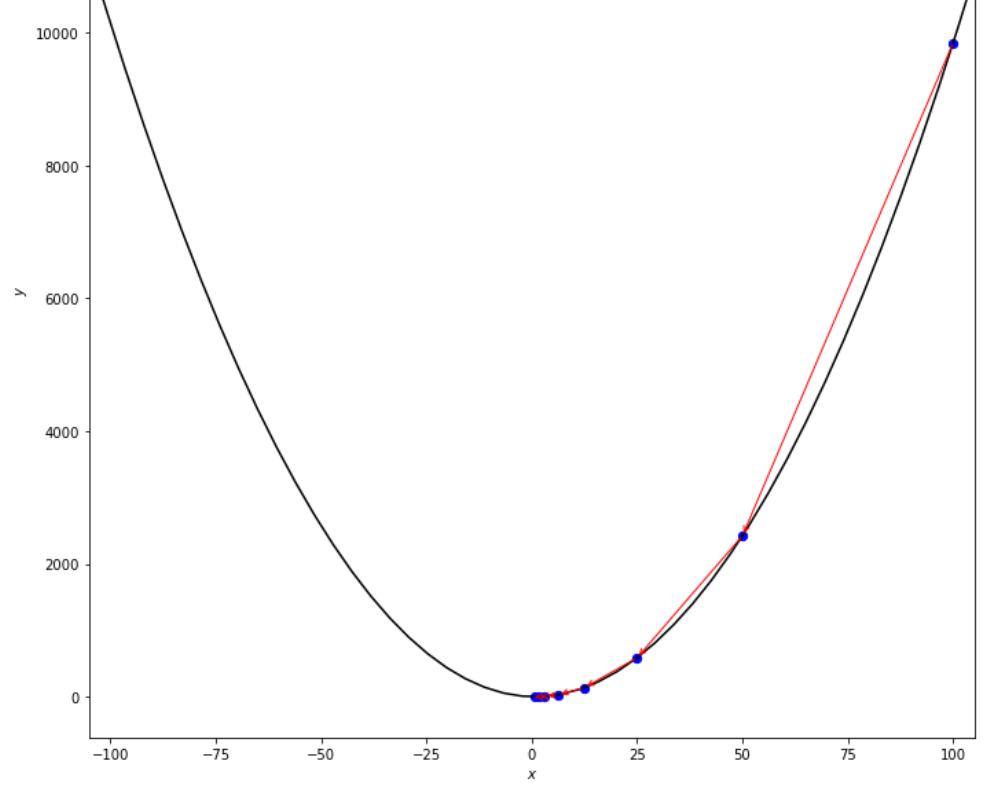
A sequência de medidas tomadas pelo algoritmo para trás do Armijo antes de atingir o mínimo
xs = 0.1
epsilon = 0.5
ŋ = 2
print ( 'x* =' , soom . armijo_forward ( f , xs , ŋ , epsilon ))
po2 . armijo_forward ( f , xs , ŋ , epsilon ) x* = 0.8 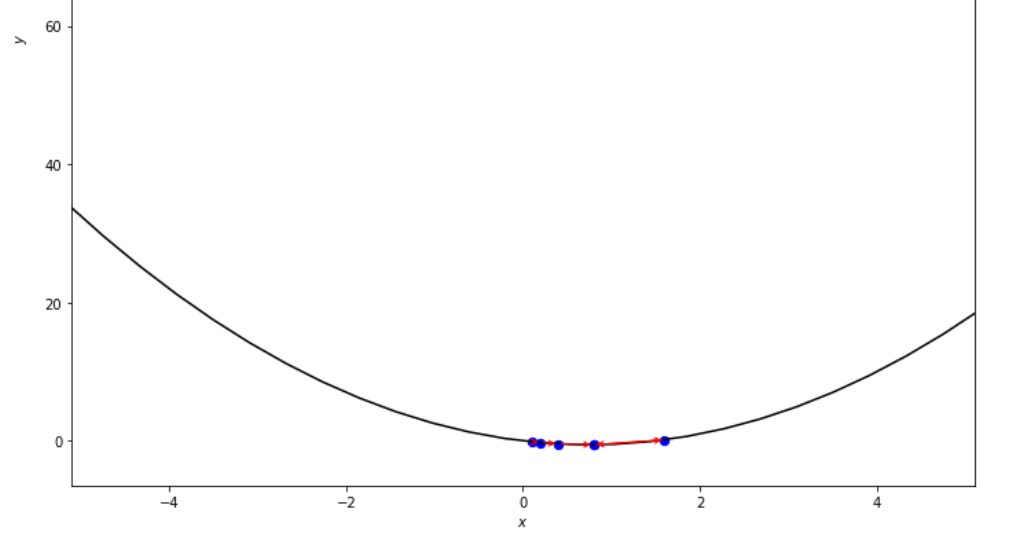
A sequência de medidas tomadas pelo algoritmo de armijo para a frente antes de atingir o mínimo
po2 . compare_all_time ( f , 0 , 2 , 1.e-2 , 1.e-3 , 10 , 2 , 0.1 , 100 ) Tempo de execução 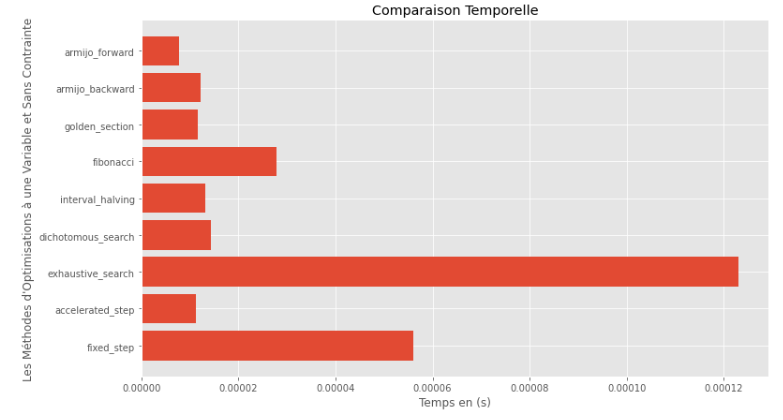
po2 . compare_all_precision ( f , 0 , 2 , 1.e-2 , 1.e-3 , 10 , 2 , 0.1 , 100 ) Lacuna entre o mínimo verdadeiro e computado 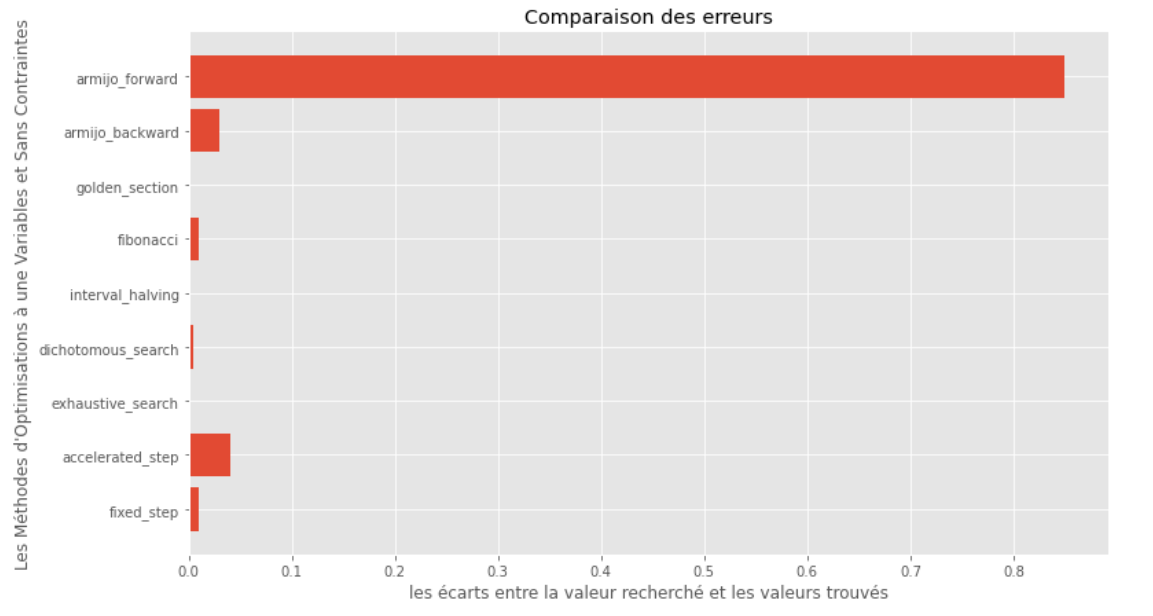
A partir dos gráficos de tempo de execução e precisão, pode-se deduzir que, entre os algoritmos avaliados para essa função convexa de variável única, o método da seção dourada surge como a escolha ideal, oferecendo uma mistura de alta precisão e tempo de execução notavelmente baixo.
import pyoptim . my_plot . multivar . _3D as pm3
import pyoptim . my_plot . multivar . contour2D as pmc def h ( x ):
return x [ 0 ] - x [ 1 ] + 2 * ( x [ 0 ] ** 2 ) + 2 * x [ 1 ] * x [ 0 ] + x [ 1 ] ** 2
# analytically, argmin(f) = [-1, 1.5]solução analítica
X = [ 1000 , 897 ]
alpha = 1.e-2
tol = 1.e-2 pmc . gradient_descent ( h , X , tol , alpha )
pm3 . gradient_descent ( h , X , tol , alpha ) Y* = [-1.01 1.51] A sequência de medidas tomadas pelo algoritmo de descida de gradiente antes de atingir o mínimo 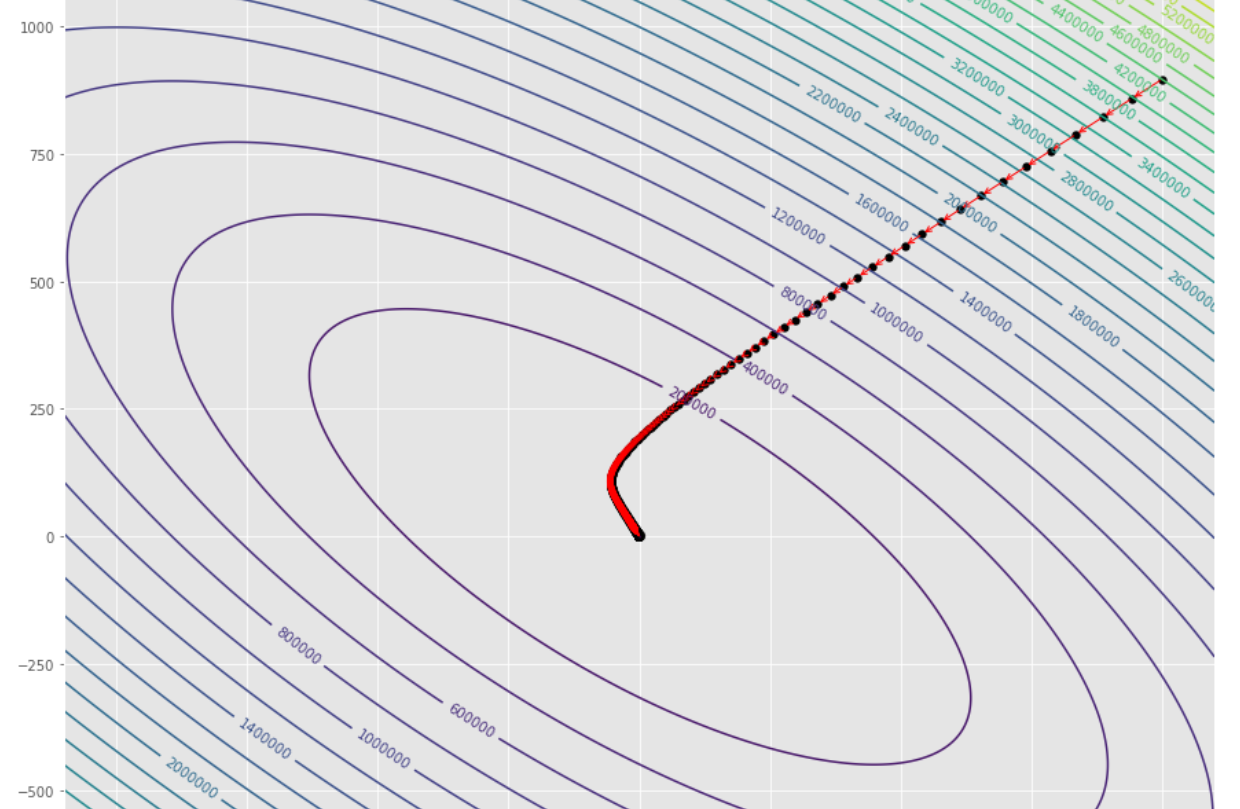
Lote de contorno 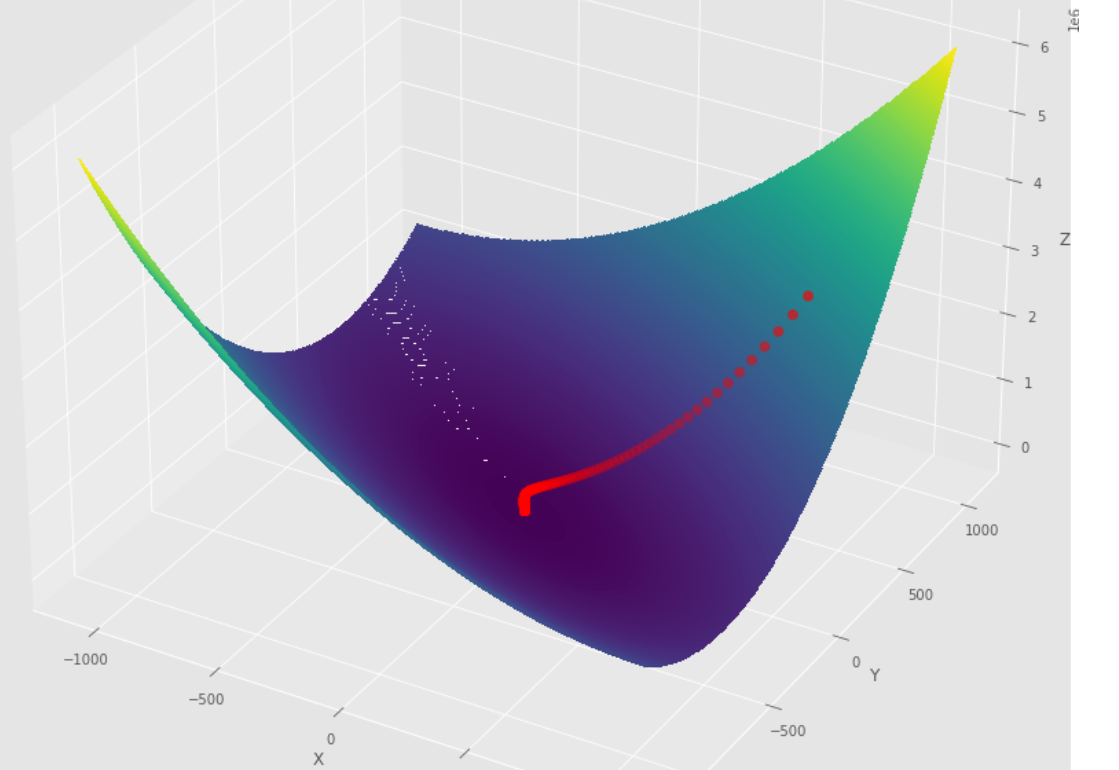
Lote 3D
pmc . gradient_conjugate ( h , X , tol )
pm3 . gradient_conjugate ( h , X , tol ) Y* = [-107.38 172.18] A sequência de medidas tomadas pelo algoritmo conjugado de gradiente antes de atingir o mínimo 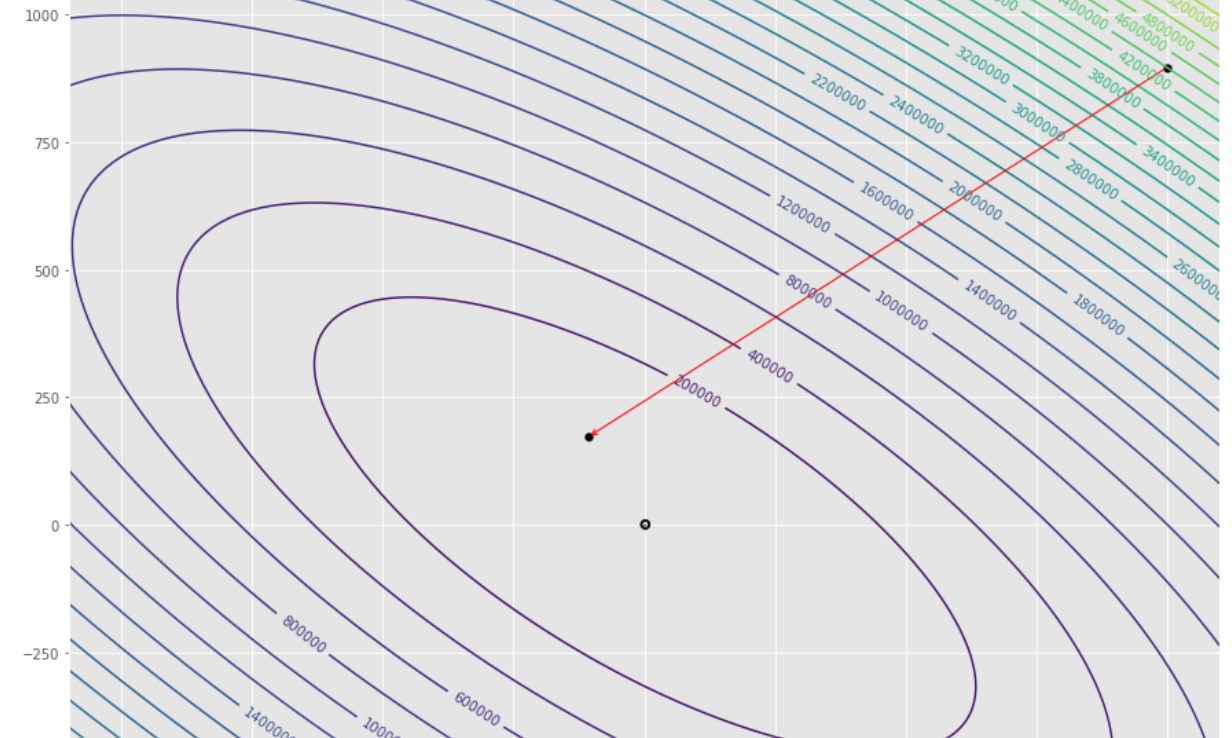
Lote de contorno 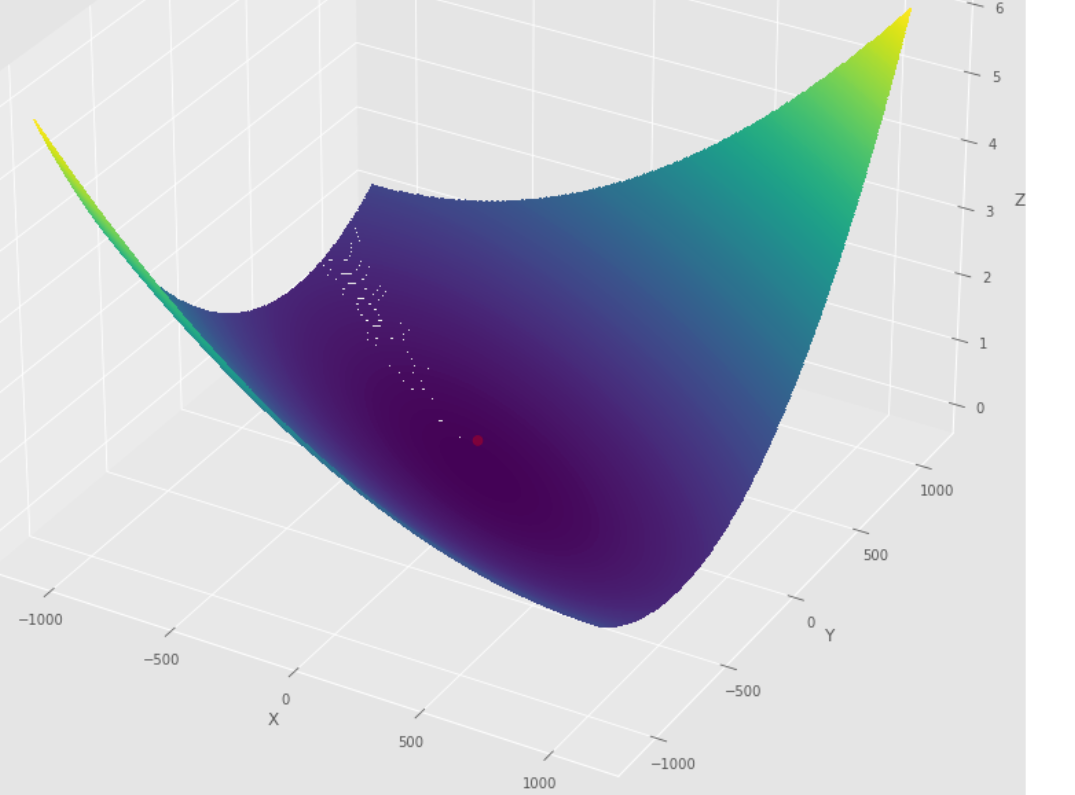
Lote 3D
pmc . newton ( h , X , tol )
pm3 . newton ( h , X , tol ) Y* = [-1. 1.5] A sequência de medidas tomadas pelo algoritmo Newton antes de atingir o mínimo 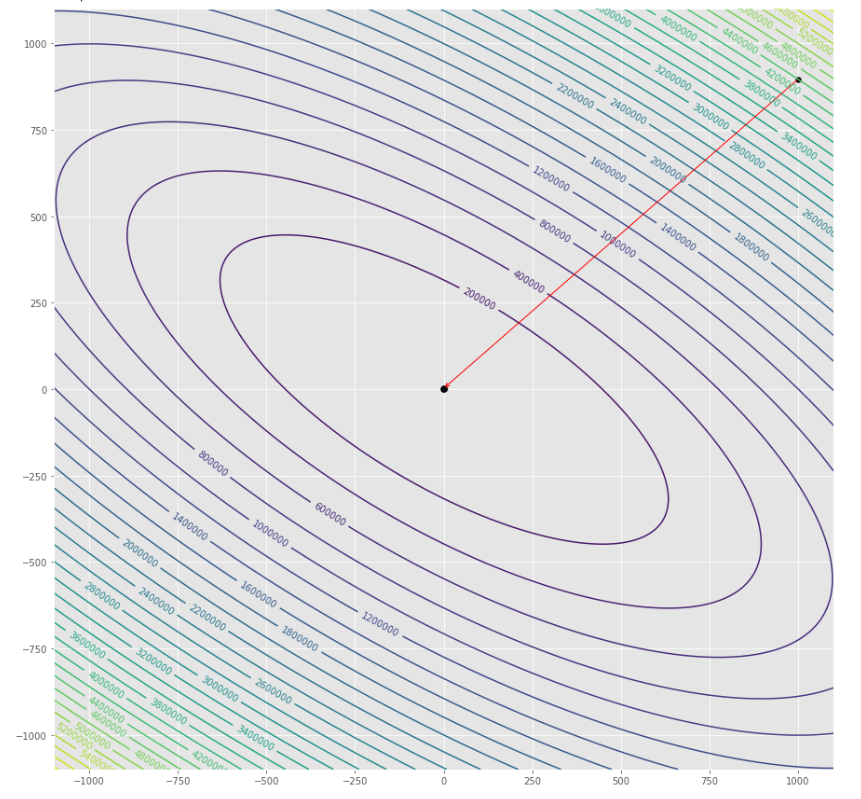
Lote de contorno 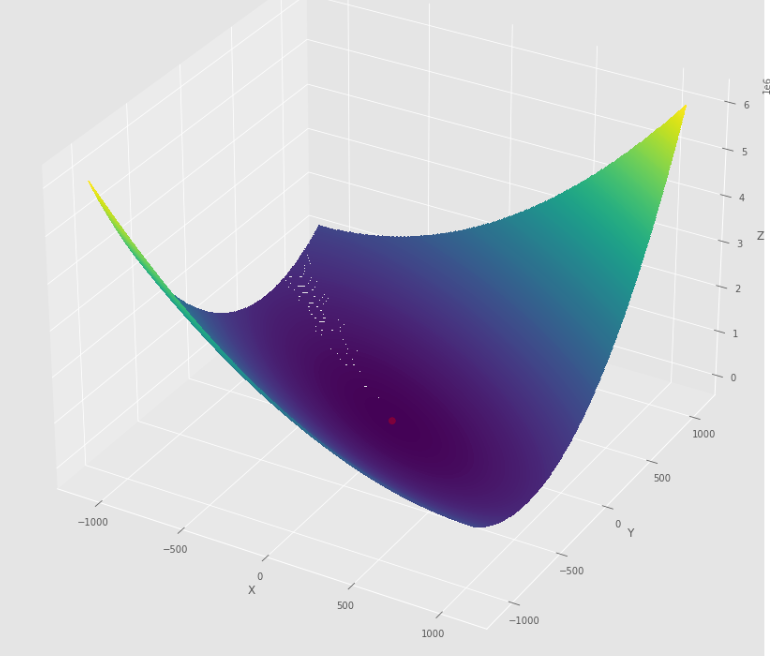
Lote 3D
pmc . quasi_newton_dfp ( h , X , tol )
pm3 . quasi_newton_dfp ( h , X , tol ) Y* = [-1. 1.5] A sequência de medidas tomadas pelo algoritmo quase newton dfp antes de atingir o mínimo 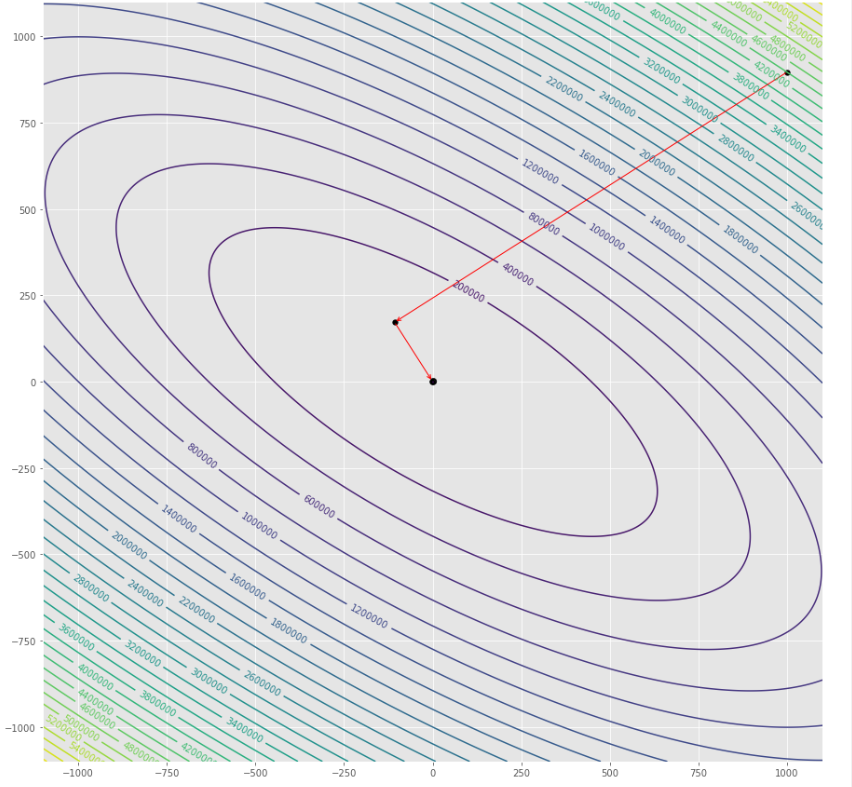
Lote de contorno 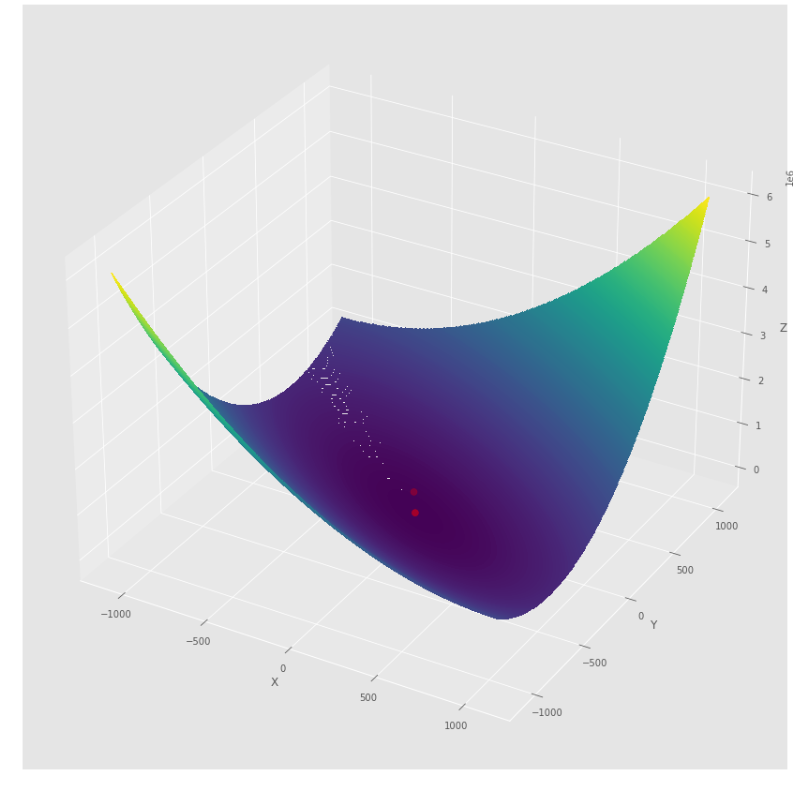
Lote 3D
pmc . sgd ( h , X , tol , alpha )
pm3 . sgd ( h , X , tol , alpha ) Y* = [-1.01 1.52] A sequência de medidas tomadas pelo algoritmo de descida de gradiente estocástico antes de atingir o mínimo 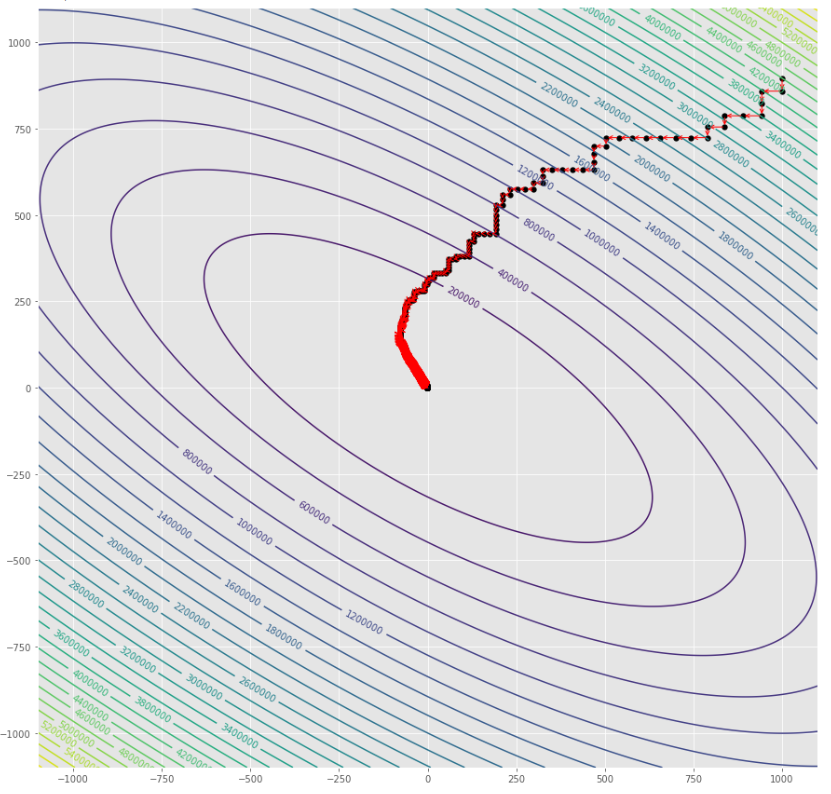
Lote de contorno 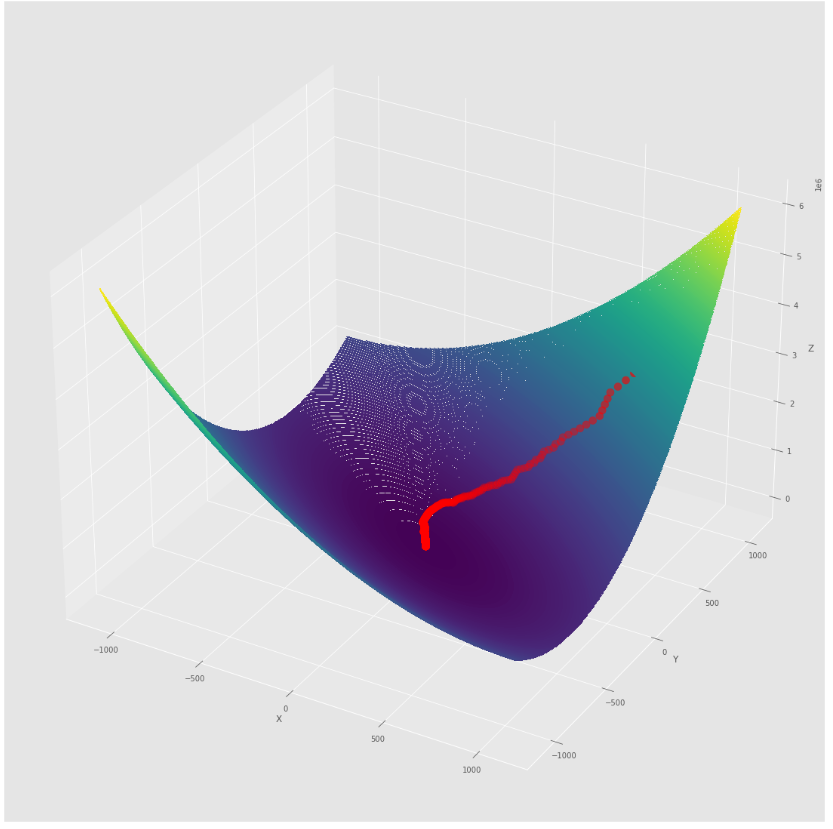
Lote 3D
alpha = 100 #it must be high because of BLS
c = 2
pmc . sgd_with_bls ( h , X , tol , alpha , c )
pm3 . sgd_with_bls ( h , X , tol , alpha , c ) Y* = [-1. 1.5] A sequência de medidas tomadas pela descida de gradiente estocástica com o algoritmo BLS antes de atingir o mínimo 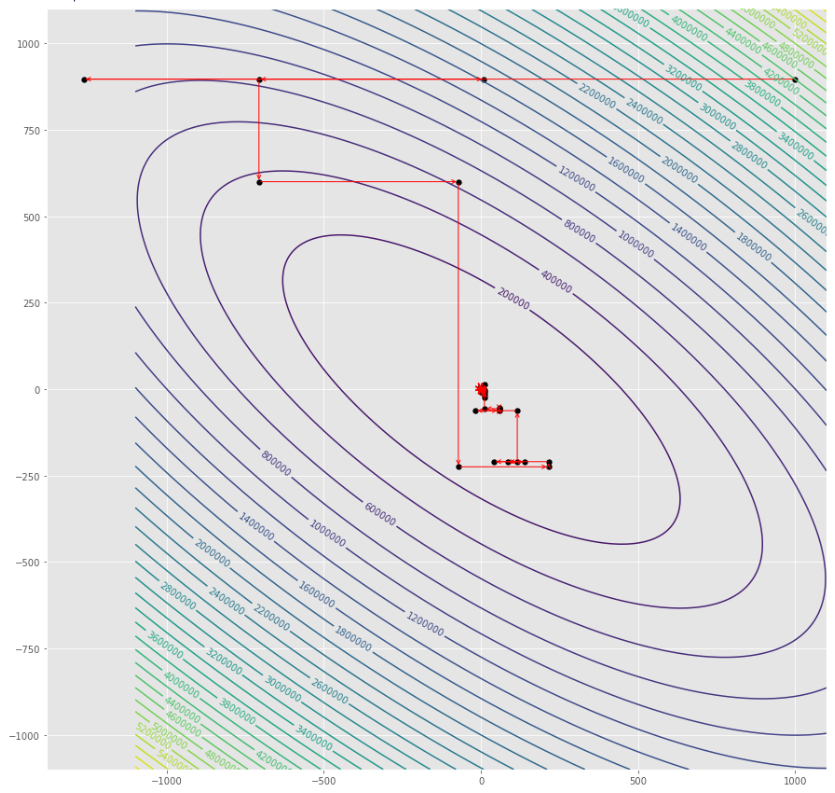
Lote de contorno 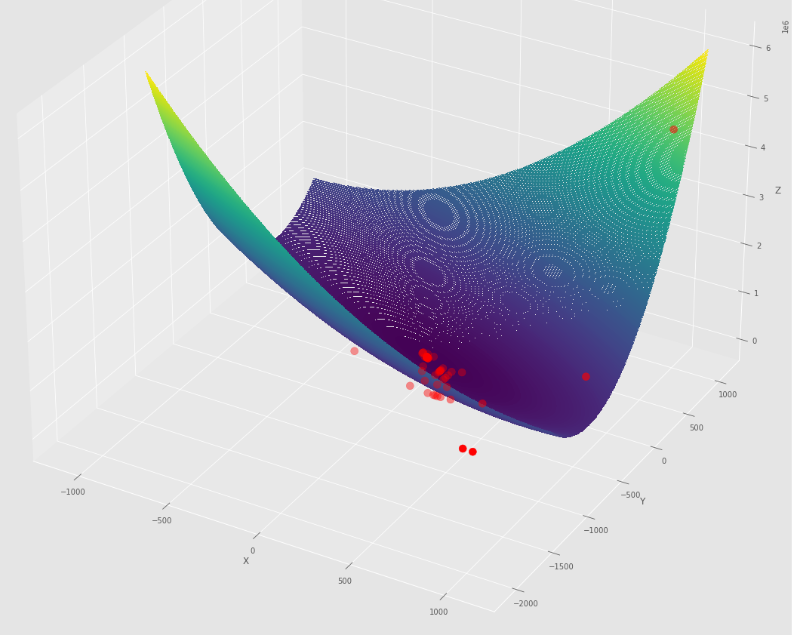
Lote 3D
pm3 . compare_all_time ( h , X , 1.e-2 , 1.e-1 , 100 , 2 ) Tempo de execução 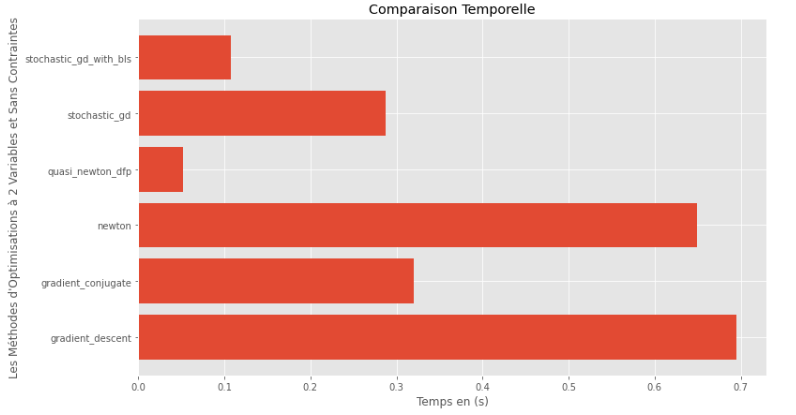
pm3 . compare_all_precision ( h , X , 1.e-2 , 1.e-1 , 100 , 2 ) Lacuna entre o mínimo verdadeiro e computado 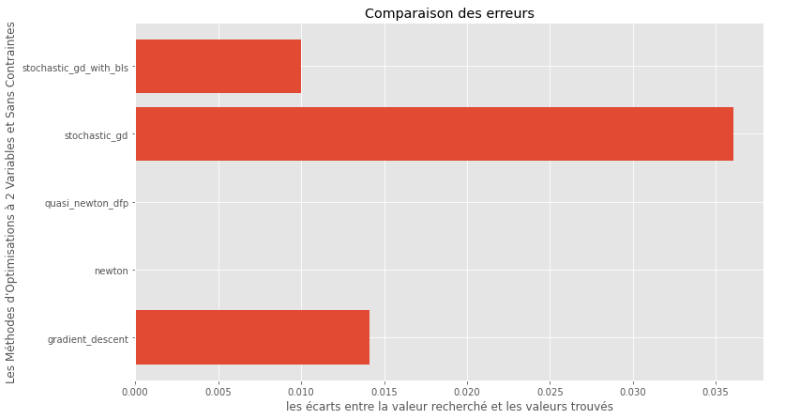
A partir dos gráficos de tempo de execução e precisão, pode -se deduzir que, entre os algoritmos avaliados para essa função multivariável convexa, o método quase Newton DFP surge como a escolha ideal, oferecendo uma mistura de alta precisão e tempo de execução baixa.
import pyoptim . my_numpy . inverse as npi 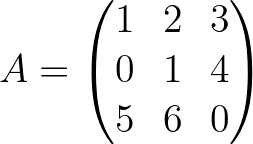
A = np . array ([[ 1. , 2. , 3. ],[ 0. , 1. , 4. ],[ 5. , 6. , 0. ]]) A_1 = npi . gaussjordan ( A . copy ())
I = A @ A_1
I = np . around ( I , 1 )
print ( 'A_1 = n n ' , A_1 )
print ( ' n A_1*A = n n ' , I ) A_1 =
[[-24. 18. 5.]
[ 20. -15. -4.]
[ -5. 4. 1.]]
A_1*A =
[[1. 0. 0.]
[0. 1. 0.]
[0. 0. 1.]] import pyoptim . my_numpy . decompose as npd A = np . array ([[ 1. , 2. , 3. ],[ 0. , 1. , 4. ],[ 5. , 6. , 0. ]]) #A is not positive definite
B = np . array ([[ 2. , - 1. , 0. ],[ - 1. , 2. , - 1. ],[ 0. , - 1. , 2. ]]) #B is positive definite
Y = np . array ([ 45 , - 78 , 95 ]) #randomly chosen column vector L , U , P = npd . LU ( A )
print ( "P = n " , P , " n n L = n " , L , " n n U = n " , U )
print ( " n " , A == P @ L @ U ) P =
[[0. 0. 1.]
[0. 1. 0.]
[1. 0. 0.]]
L =
[[1. 0. 0. ]
[0. 1. 0. ]
[0.2 0.8 1. ]]
U =
[[ 5. 6. 0. ]
[ 0. 1. 4. ]
[ 0. 0. -0.2]]
[[ True True True]
[ True True True]
[ True True True]] L = npd . choleski ( A ) # A is not positive definite
print ( L )
print ( "--------------------------------------------------" )
L = npd . choleski ( B ) # B is positive definite
print ( 'L = n ' , L , ' n ' )
C = np . around ( L @( L . T ), 1 )
print ( 'B = L@(L.T) n n ' , B == C ) A must be positive definite !
None
--------------------------------------------------
L =
[[ 1.41421356 0. 0. ]
[-0.70710678 1.22474487 0. ]
[ 0. -0.81649658 1.15470054]]
B = L@(L.T)
[[ True True True]
[ True True True]
[ True True True]] import pyoptim . my_numpy . solve as nps 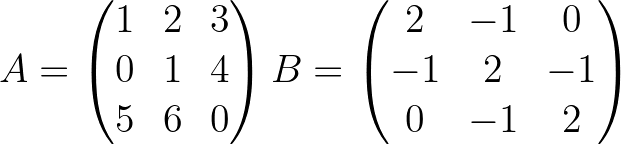
A = np . array ([[ 1. , 2. , 3. ], [ 0. , 1. , 4. ], [ 5. , 6. , 0. ]]) # A is not positive definite
B = np . array ([[ 2. , - 1. , 0. ], [ - 1. , 2. , - 1. ], [ 0. , - 1. , 2. ]]) # B is positive definite
Y = np . array ([ 45 , - 78 , 95 ]) # randomly chosen column vector X = nps . gaussjordan ( A , Y )
print ( "X = n " , X )
print ( " n A@X=Y n " , A @ X == Y , ' n ' )
print ( '---------------------------------------------------------------' )
X = nps . gaussjordan ( B , Y )
print ( "X = n " , X )
Y_ = np . around ( B @ X , 1 )
print ( " n B@X=Y n " , Y_ == Y , ' n ' ) X =
[[-2009.]
[ 1690.]
[ -442.]]
A@X=Y
[[ True]
[ True]
[ True]]
---------------------------------------------------------------
X =
[[18.5]
[-8. ]
[43.5]]
B@X=Y
[[ True]
[ True]
[ True]]
X = nps . LU ( A , Y )
print ( "X* = n " , X )
print ( " n AX*=Y n " , A @ X == Y )
print ( "-------------------------------------------------------------------------------" );
X = nps . LU ( B , Y )
print ( "X* = n " , X )
Y_ = np . around ( B @ X , 1 )
print ( " n BX*=Y n " , Y_ == Y ) X* =
[[-2009.]
[ 1690.]
[ -442.]]
AX*=Y
[[ True]
[ True]
[ True]]
-------------------------------------------------------------------------------
X* =
[[18.5]
[-8. ]
[43.5]]
BX*=Y
[[ True]
[ True]
[ True]] X = nps . choleski ( A , Y )
print ( "-------------------------------------------------------------------------------" )
X = nps . choleski ( B , Y )
print ( "X = n " , X )
Y_ = np . around ( B @ X , 1 )
print ( " n BX*=Y n " , Y_ == Y ) !! A must be positive definite !!
-------------------------------------------------------------------------------
X =
[[18.5]
[-8. ]
[43.5]]
BX*=Y
[[ True]
[ True]
[ True]]