Pyoptim : 최적화 및 매트릭스 작업을위한 Python 패키지
이 프로젝트는 ENSIAS의 수치 분석 및 최적화 과정의 구성 요소 인 Mohammed v University 교수 M. Naoum의 제안에서 실험실 세션 중에 실행되는 Python 패키지 캡슐화 알고리즘을 만들기위한 제안에서 발생했습니다. 약 10 개의 구속되지 않은 최적화 알고리즘에 중점을 두어 2D 및 3D 시각화를 제공하여 계산 효율성과 정확도의 성능 비교를 가능하게합니다. 또한이 프로젝트에는 실험실에서 유래 된 알고리즘이 포함 된 패키지 내에 통합 된 역전, 분해 및 선형 시스템 해결과 같은 행렬 작업이 포함됩니다.
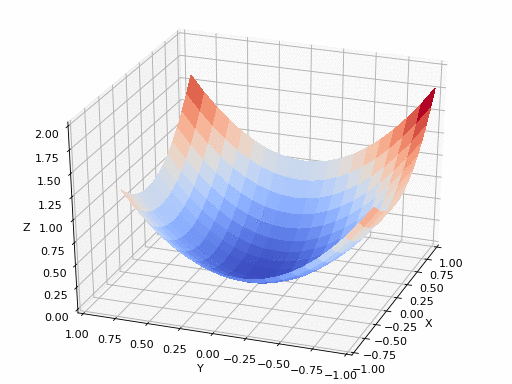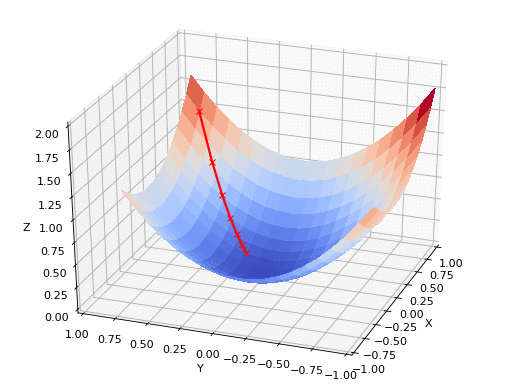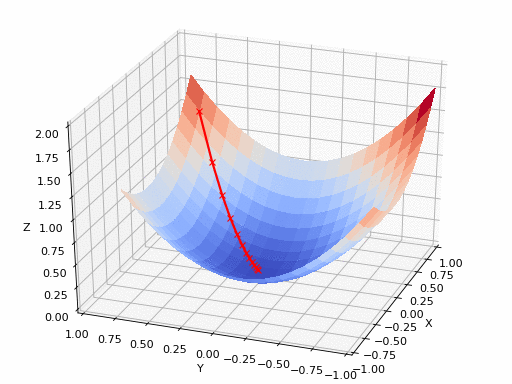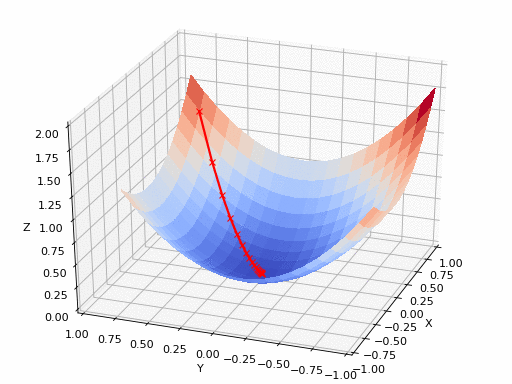
gif 출처 : https://www.nsf.gov/news/mmg/mmg_disp.jsp?med_id_id_id=78950&from= ](https://aria42.com/blog/2014/12/understanding-lbfgs
이 프로젝트의 모든 부분은 다음을 수행하는 방법을 보여주는 샘플 코드입니다.
고정 된 최적화 알고리즘의 구현, 고정 _step, accelerated_step, paridrive_search, dichotomous_search, interval_halving, fibonacci, golden_section, armijo_backward 및 armijo_forward를 포함합니다.
그라디언트 하강, 그라디언트 컨쥬 게이트, 뉴턴, Quasi_Newton_DFP, 확률 적 구배 하강을 포함한 다양한 1 가지 변수 최적화 알고리즘의 통합.
2D, 윤곽 및 3D 형식의 모든 알고리즘에 대한 각 반복 단계의 시각화.
런타임 및 정확도 지표에 중점을 둔 비교 분석을 수행합니다.
반전 (예 : Gauss-Jordan), 분해 (예 : Lu, Choleski) 및 선형 시스템 용 솔루션 (예 : Gauss-Jordan, Lu, Choleski)과 같은 매트릭스 작업의 구현.
이 파이썬 패키지를 설치하려면 :
pip install pyoptim이 데모 노트북을 실행하십시오
import pyoptim . my_scipy . onevar_optimize . minimize as soom
import pyoptim . my_plot . onevar . _2D as po2 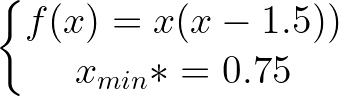
def f ( x ):
return x * ( x - 1.5 ) # analytically, argmin(f) = 0.75 
xs = - 10
xf = 10
epsilon = 1.e-2 print ( 'x* =' , soom . fixed_step ( f , xs , epsilon ))
po2 . fixed_step ( f , xs , epsilon ) x* = 0.75 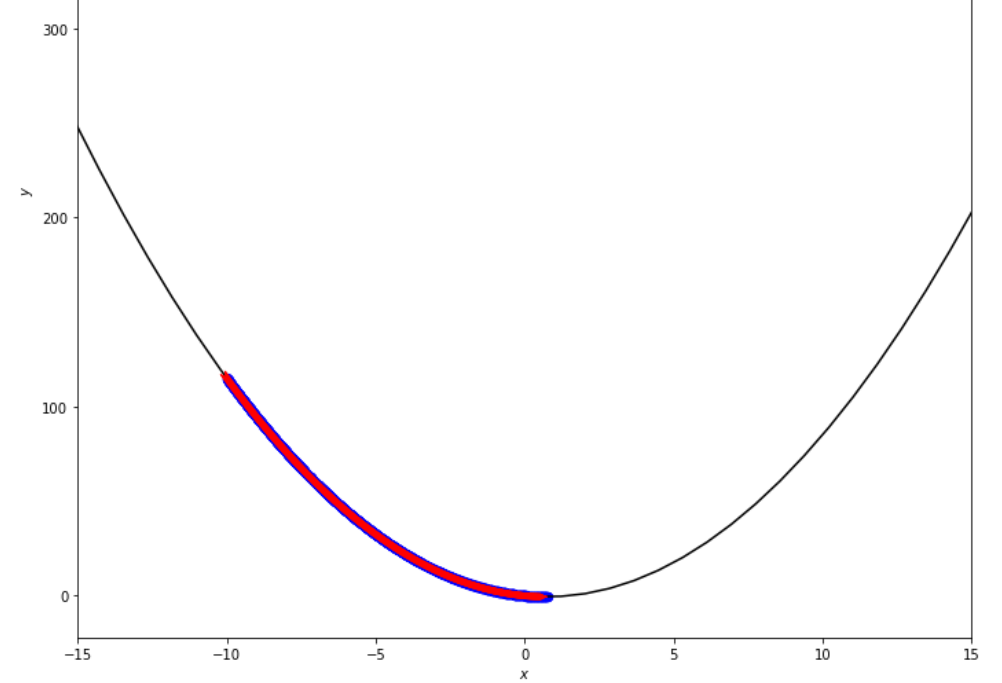
최소에 도달하기 전에 고정 된 단계 알고리즘에 의해 수행 된 단계 순서
print ( 'x* =' , soom . accelerated_step ( f , xs , epsilon ))
po2 . accelerated_step ( f , xs , epsilon ) x* = 0.86 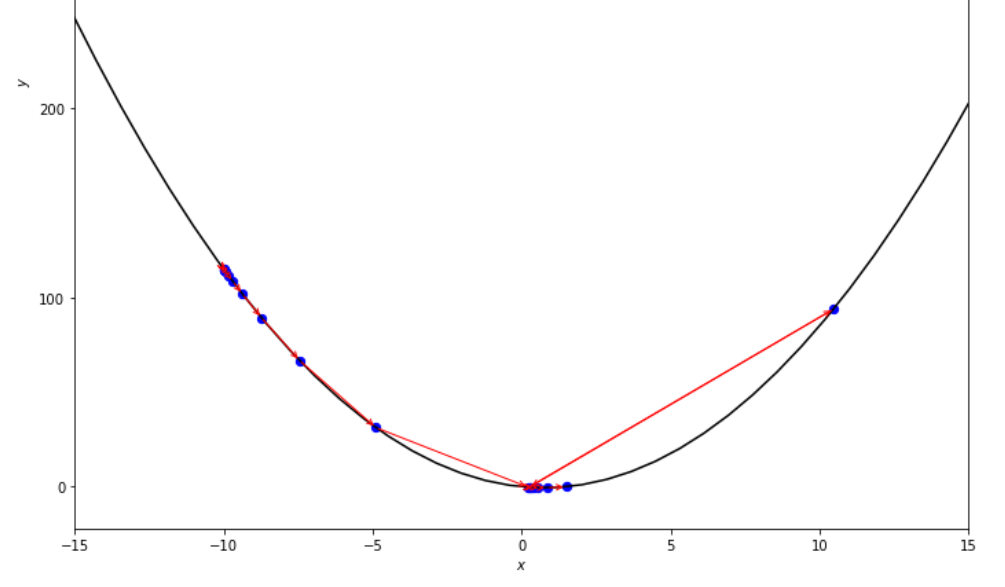
최소에 도달하기 전에 가속 된 단계 알고리즘에 의해 취한 단계의 순서
print ( 'x* =' , soom . exhaustive_search ( f , xs , xf , epsilon ))
po2 . exhaustive_search ( f , xs , xf , epsilon ) x* = 0.75
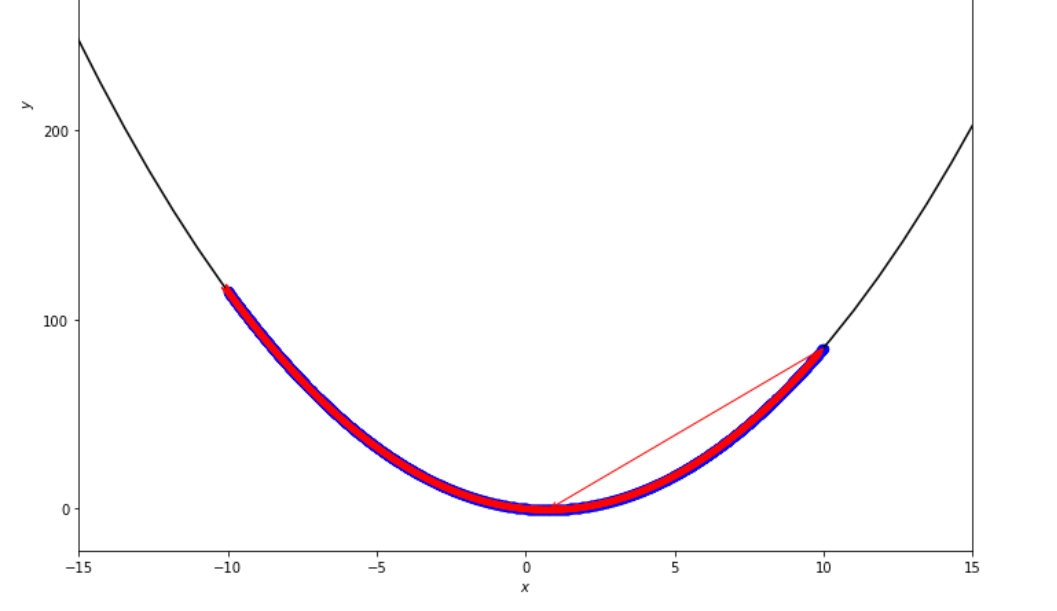
최소에 도달하기 전에 철저한 검색 알고리즘에 의해 수행 된 일련의 단계
mini_delta = 1.e-3
print ( 'x* =' , soom . dichotomous_search ( f , xs , xf , epsilon , mini_delta ))
po2 . dichotomous_search ( f , xs , xf , epsilon , mini_delta ) x* = 0.7494742431640624
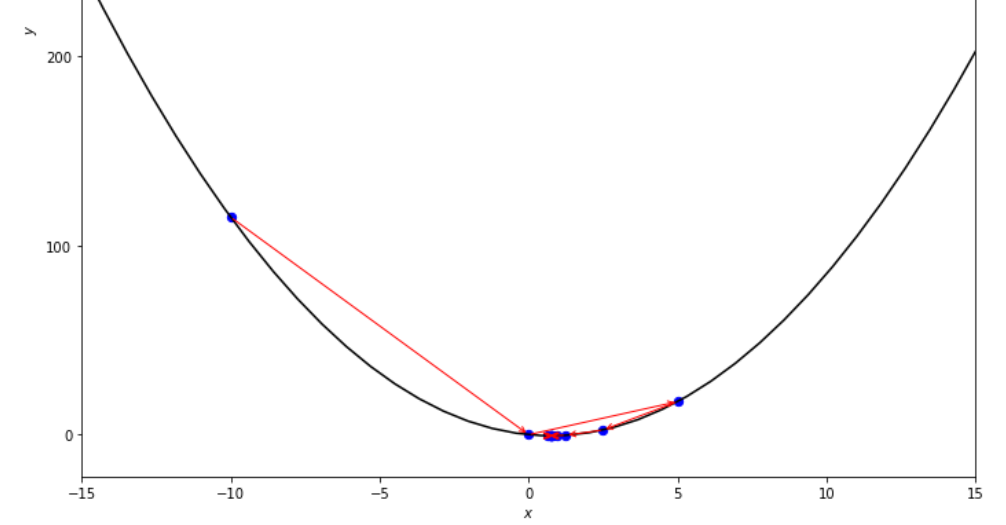
최소에 도달하기 전에 이분법 검색 알고리즘에 의해 수행 된 단계의 순서
print ( 'x* =' , soom . interval_halving ( f , xs , xf , epsilon ))
po2 . interval_halving ( f , xs , xf , epsilon ) x* = 0.75
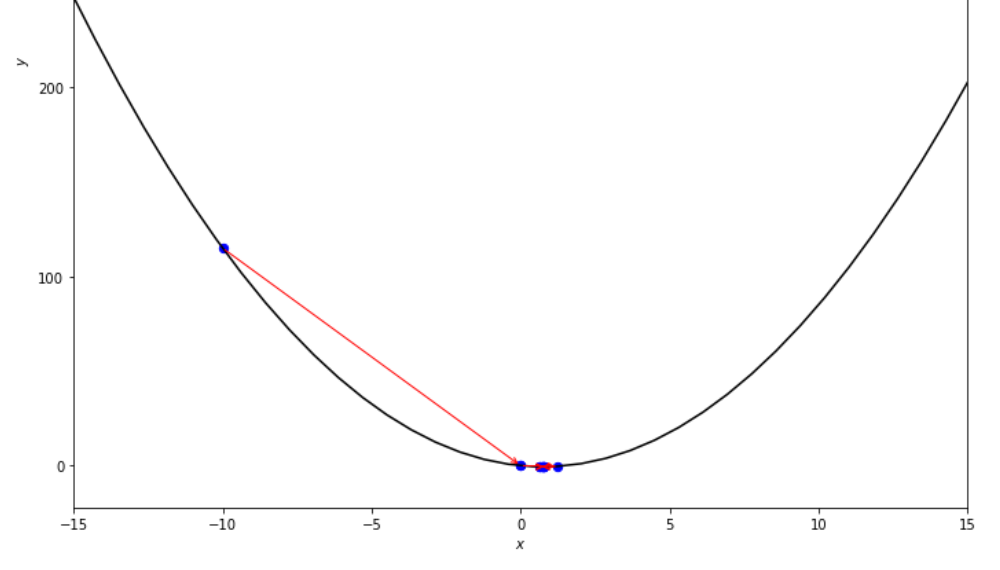
최소에 도달하기 전에 간격 절반 알고리즘에 의해 취한 단계 일련의 단계.
n = 15
print ( 'x* =' , soom . fibonacci ( f , xs , xf , n ))
po2 . fibonacci ( f , xs , xf , n ) x* = 0.76 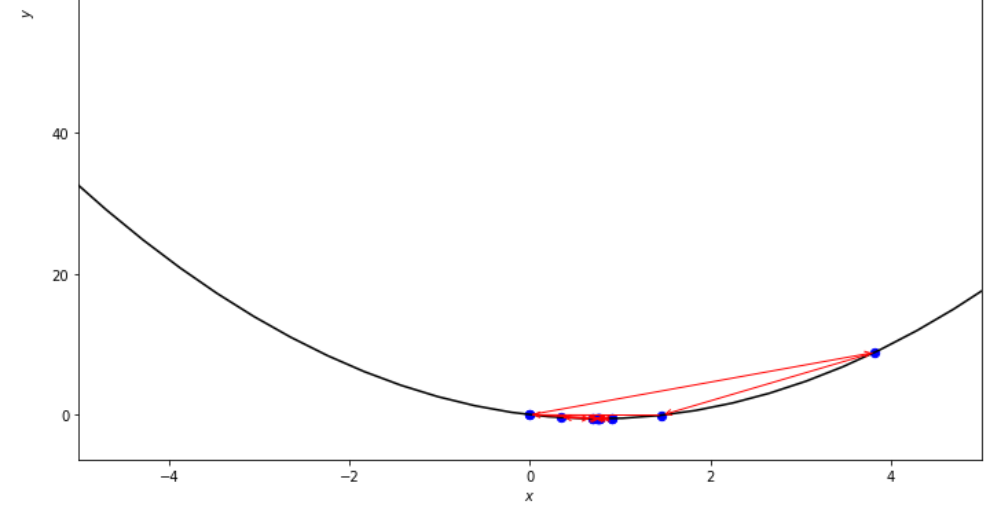
최소에 도달하기 전에 Fibonacci 알고리즘에 의해 취한 단계 일련의 단계.
print ( 'x* =' , soom . golden_section ( f , xs , xf , epsilon ))
po2 . golden_section ( f , xs , xf , epsilon ) x* = 0.75 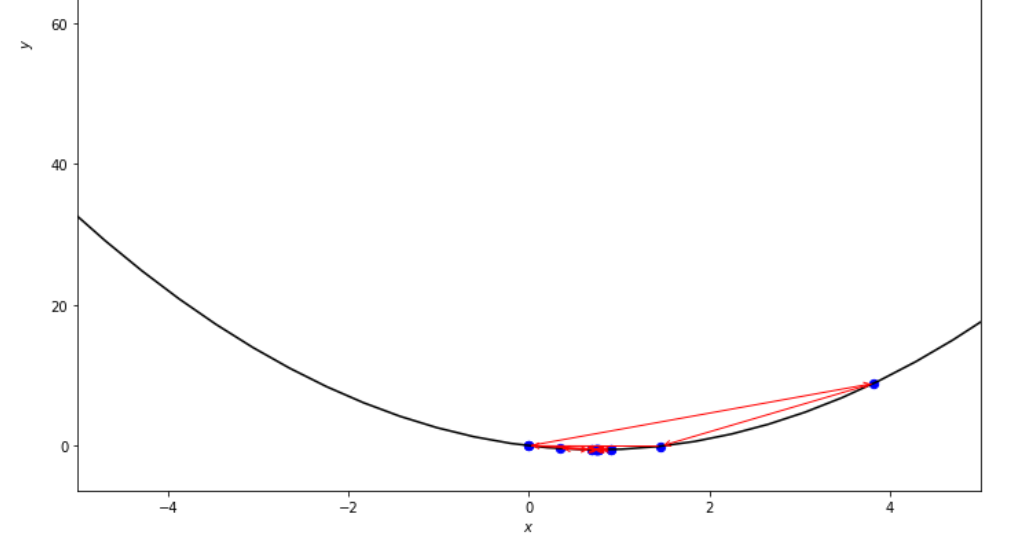
최소값에 도달하기 전에 황금 섹션 알고리즘에 의해 취한 일련의 단계
ŋ = 2
xs = 100
print ( 'x* =' , soom . armijo_backward ( f , xs , ŋ , epsilon ))
po2 . armijo_backward ( f , xs , ŋ , epsilon ) x* = 0.78 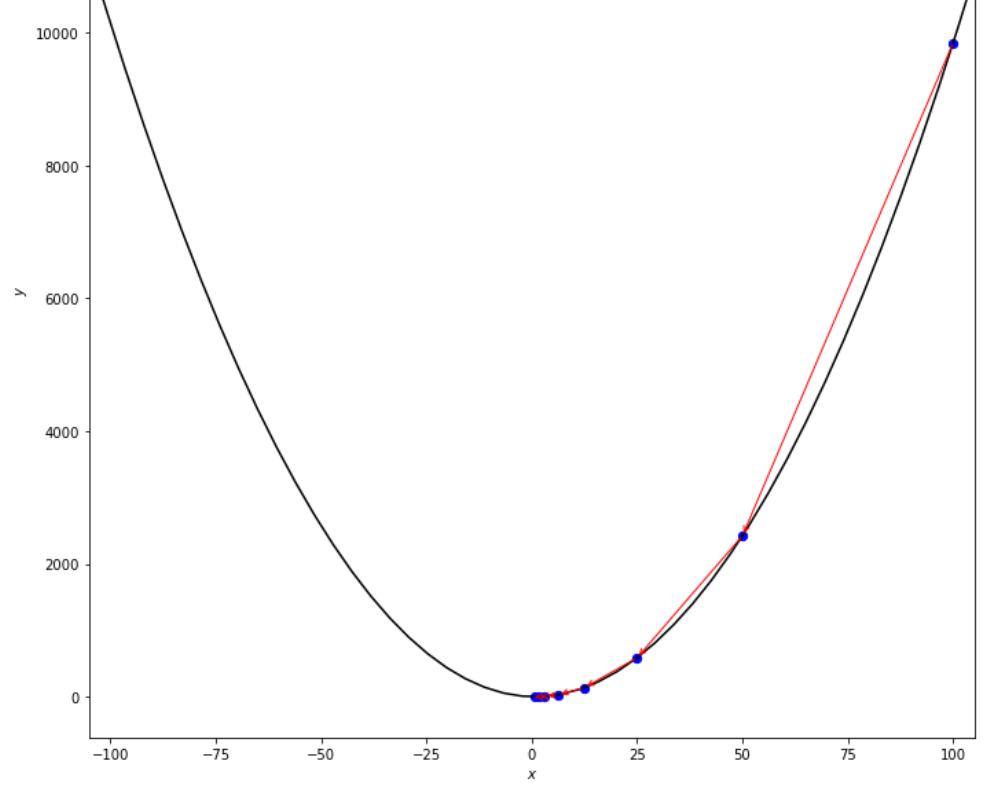
최소에 도달하기 전에 Armijo Backward 알고리즘이 수행 한 단계의 순서
xs = 0.1
epsilon = 0.5
ŋ = 2
print ( 'x* =' , soom . armijo_forward ( f , xs , ŋ , epsilon ))
po2 . armijo_forward ( f , xs , ŋ , epsilon ) x* = 0.8 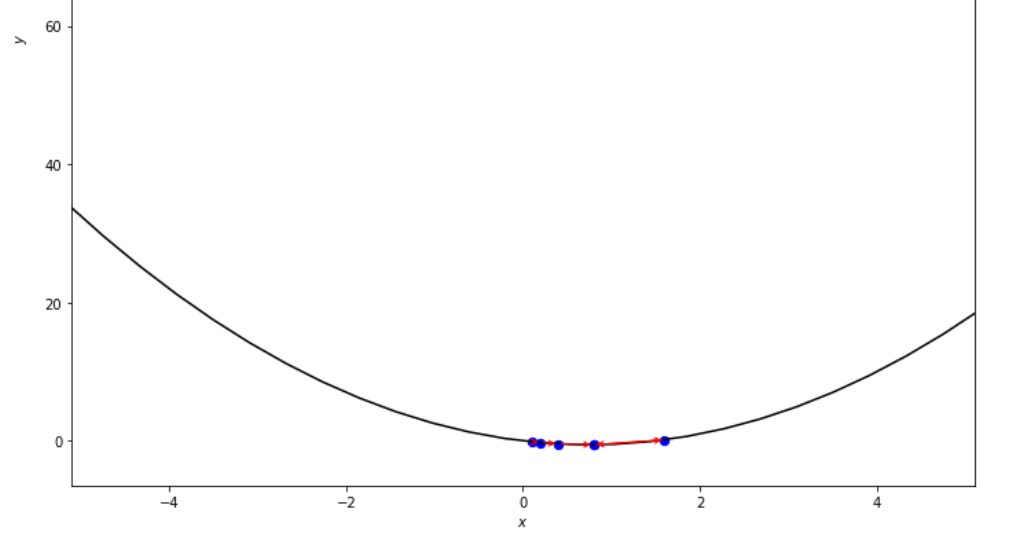
최소에 도달하기 전에 Armijo Forward 알고리즘이 수행 한 단계의 순서
po2 . compare_all_time ( f , 0 , 2 , 1.e-2 , 1.e-3 , 10 , 2 , 0.1 , 100 ) 실행 시간 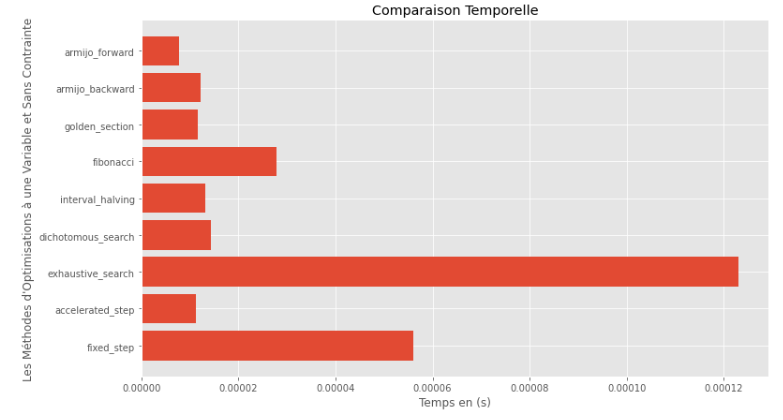
po2 . compare_all_precision ( f , 0 , 2 , 1.e-2 , 1.e-3 , 10 , 2 , 0.1 , 100 ) True와 Computed Minims 사이의 간격 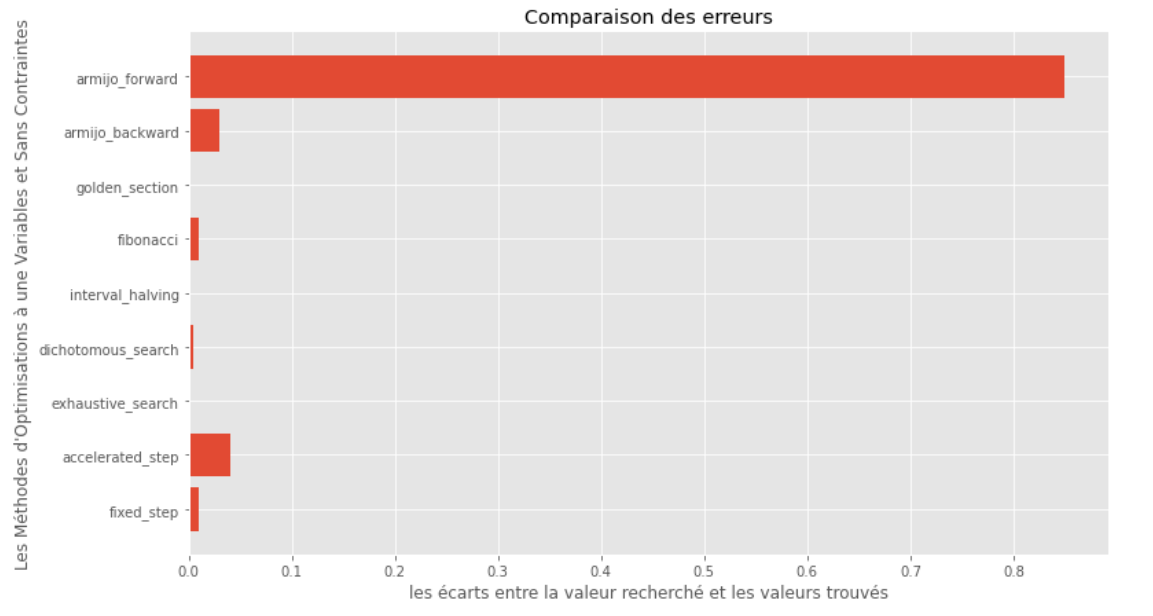
런타임 및 정확도 그래프 에서이 볼록 단일 변수 기능에 대해 평가 된 알고리즘 중에서 골든 섹션 방법이 최적의 선택으로 나타나서 높은 정확도와 특히 낮은 런타임을 제공한다는 것을 추론 할 수 있습니다.
import pyoptim . my_plot . multivar . _3D as pm3
import pyoptim . my_plot . multivar . contour2D as pmc def h ( x ):
return x [ 0 ] - x [ 1 ] + 2 * ( x [ 0 ] ** 2 ) + 2 * x [ 1 ] * x [ 0 ] + x [ 1 ] ** 2
# analytically, argmin(f) = [-1, 1.5]항문 솔루션
X = [ 1000 , 897 ]
alpha = 1.e-2
tol = 1.e-2 pmc . gradient_descent ( h , X , tol , alpha )
pm3 . gradient_descent ( h , X , tol , alpha ) Y* = [-1.01 1.51] 최소에 도달하기 전에 그라디언트 하강 알고리즘에 의해 수행 된 단계의 순서 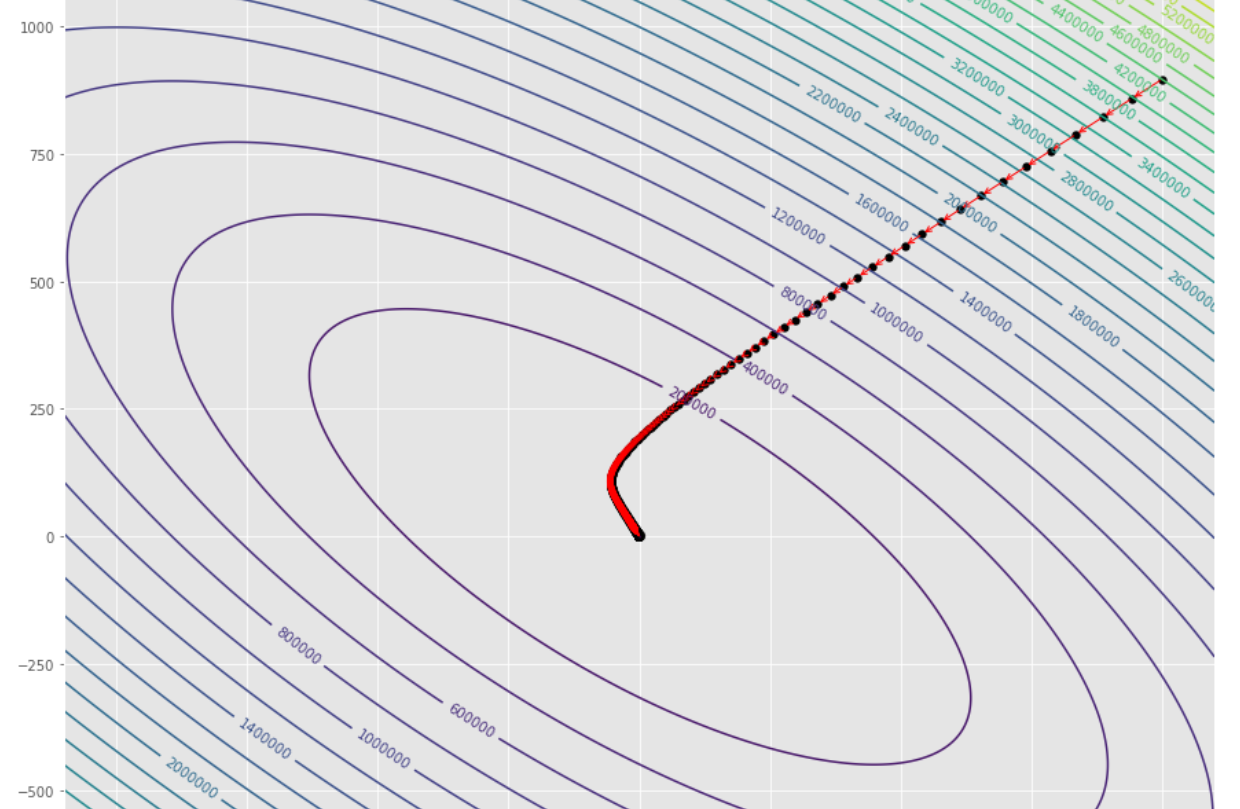
윤곽 줄거리 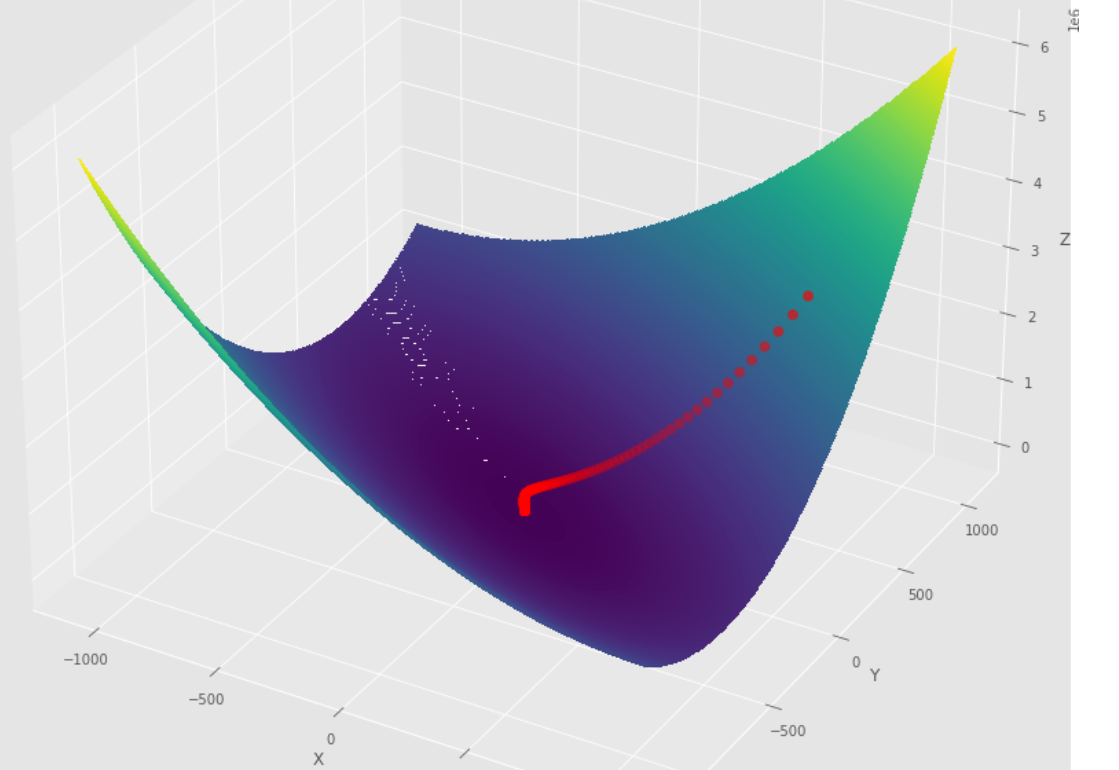
3D 플롯
pmc . gradient_conjugate ( h , X , tol )
pm3 . gradient_conjugate ( h , X , tol ) Y* = [-107.38 172.18] 최소에 도달하기 전에 그라디언트 컨쥬 게이트 알고리즘에 의해 수행 된 단계의 순서 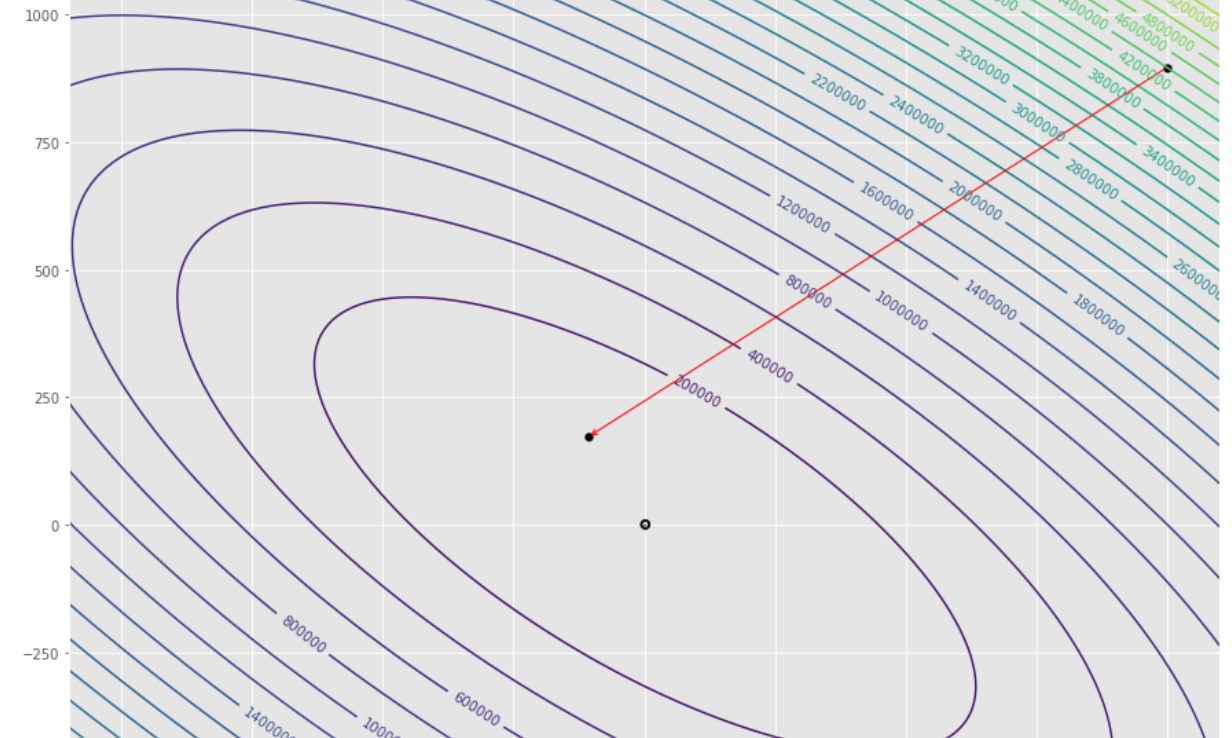
윤곽 줄거리 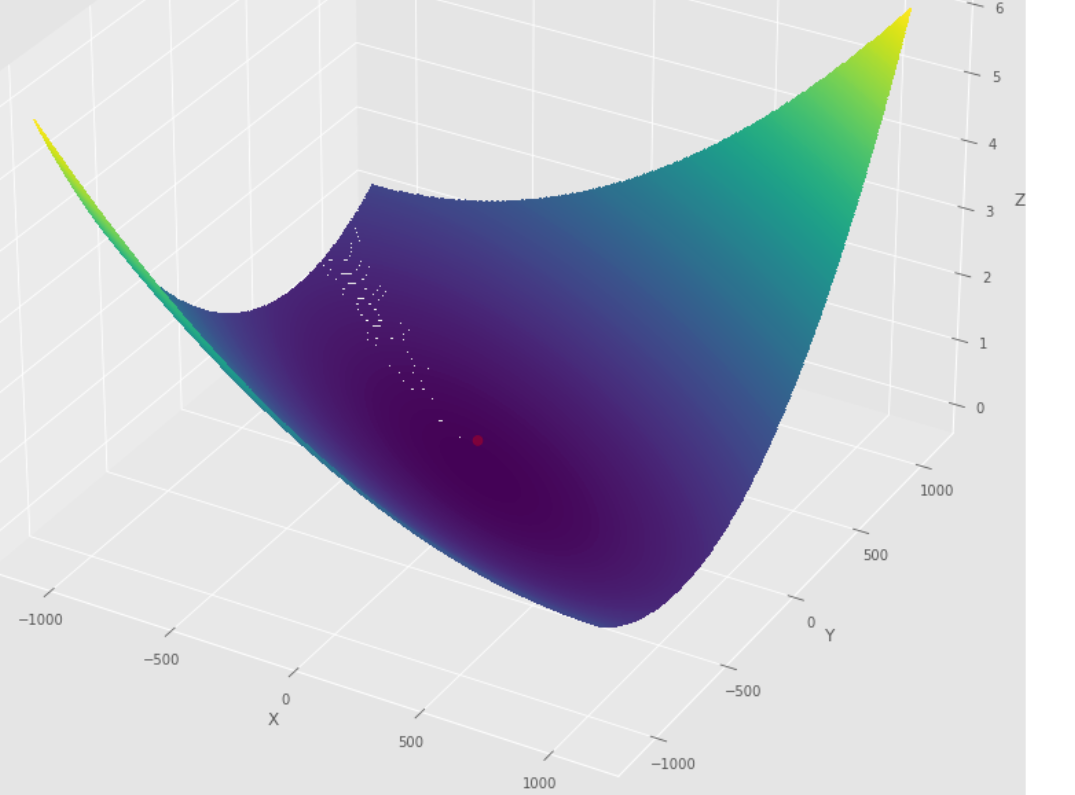
3D 플롯
pmc . newton ( h , X , tol )
pm3 . newton ( h , X , tol ) Y* = [-1. 1.5] 최소에 도달하기 전에 Newton 알고리즘에 의해 수행 된 일련의 단계 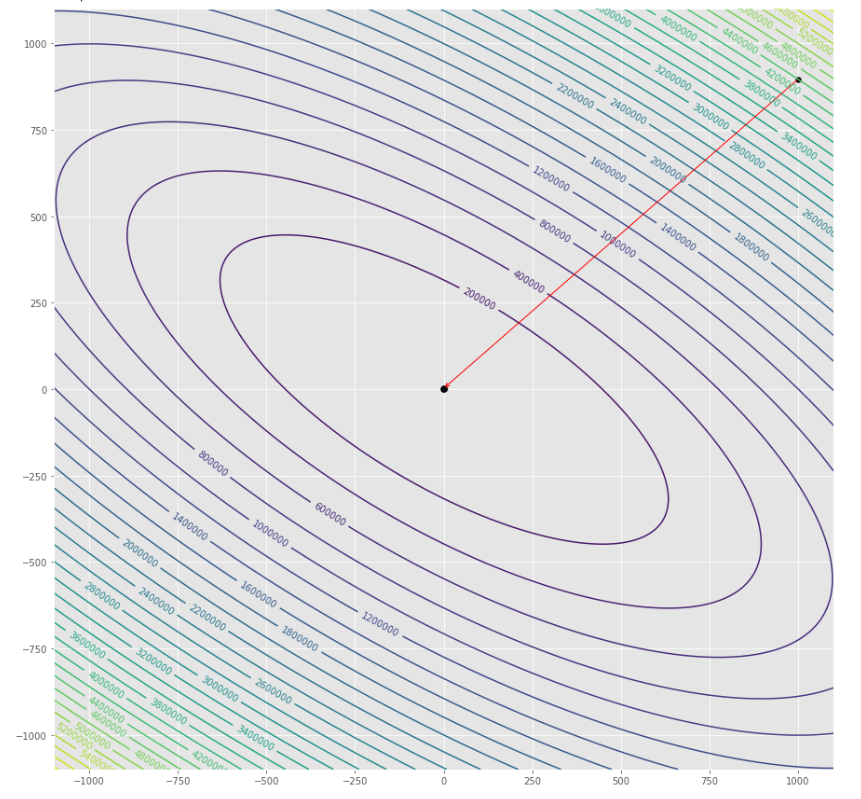
윤곽 줄거리 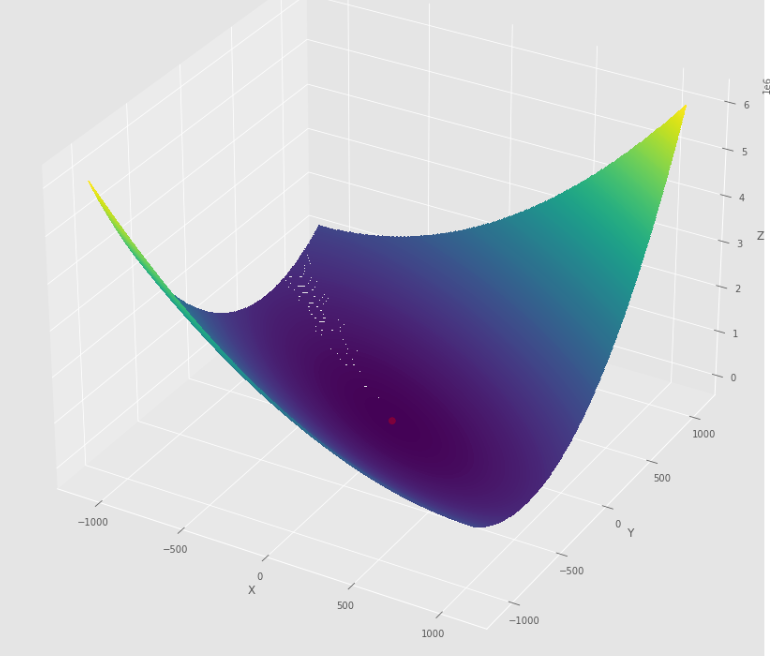
3D 플롯
pmc . quasi_newton_dfp ( h , X , tol )
pm3 . quasi_newton_dfp ( h , X , tol ) Y* = [-1. 1.5] 최소에 도달하기 전에 Quasi Newton DFP 알고리즘에 의해 수행 된 단계의 순서 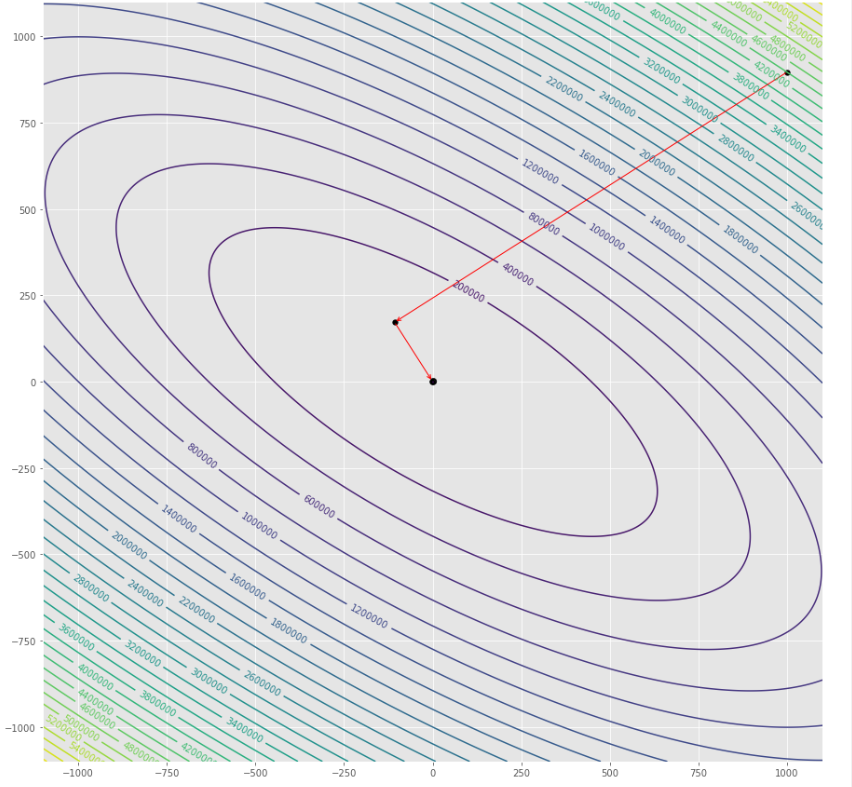
윤곽 줄거리 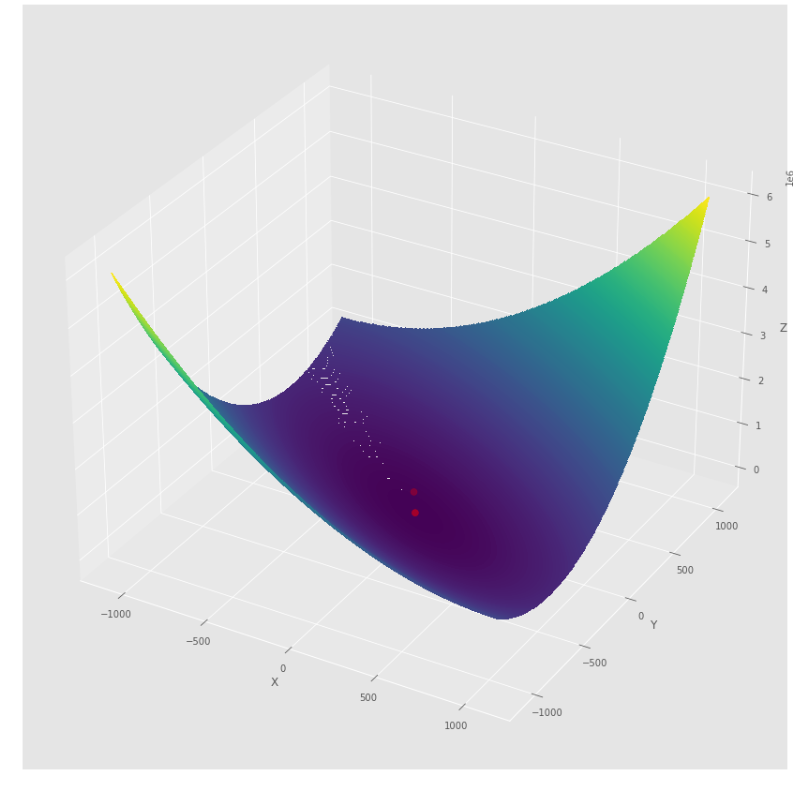
3D 플롯
pmc . sgd ( h , X , tol , alpha )
pm3 . sgd ( h , X , tol , alpha ) Y* = [-1.01 1.52] 최소에 도달하기 전에 확률 적 구배 하강 알고리즘에 의해 수행 된 단계의 순서 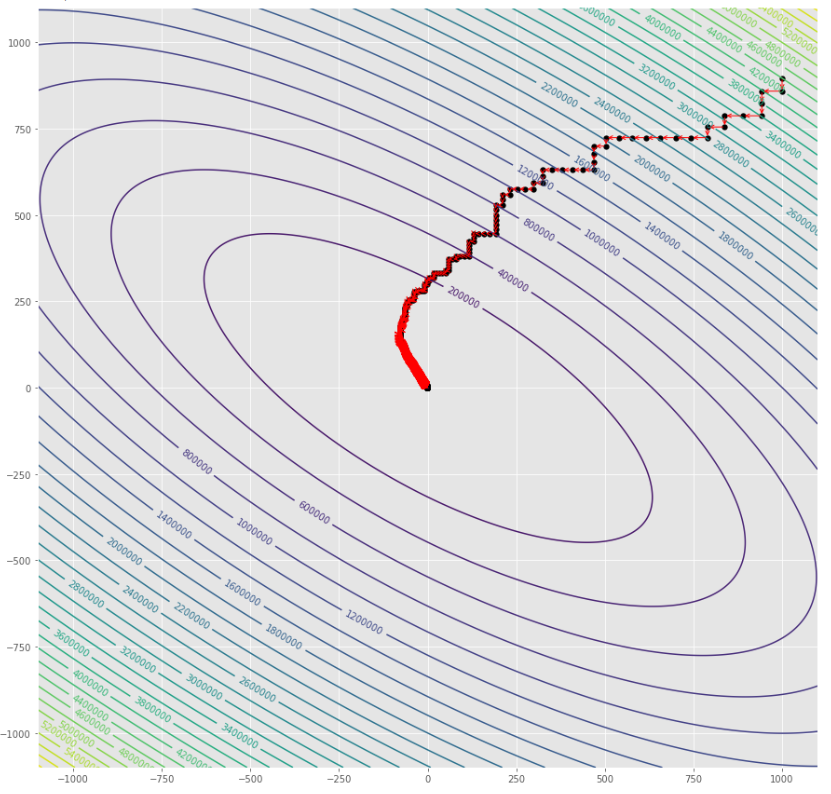
윤곽 줄거리 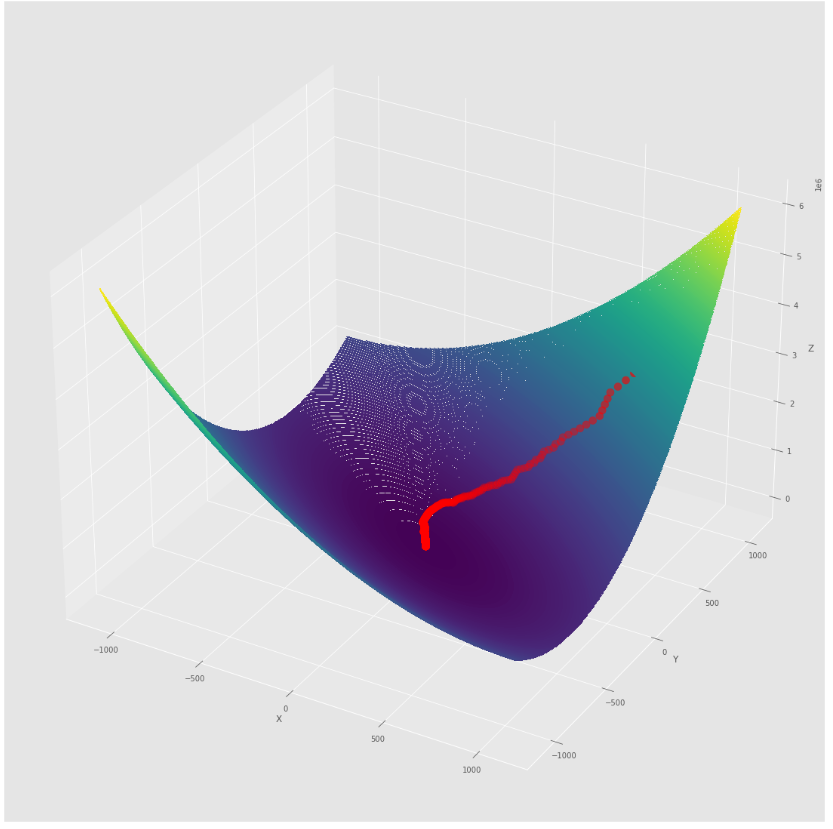
3D 플롯
alpha = 100 #it must be high because of BLS
c = 2
pmc . sgd_with_bls ( h , X , tol , alpha , c )
pm3 . sgd_with_bls ( h , X , tol , alpha , c ) Y* = [-1. 1.5] 최소에 도달하기 전에 BLS 알고리즘으로 확률 론적 구배 출신에 의해 취해진 단계의 순서 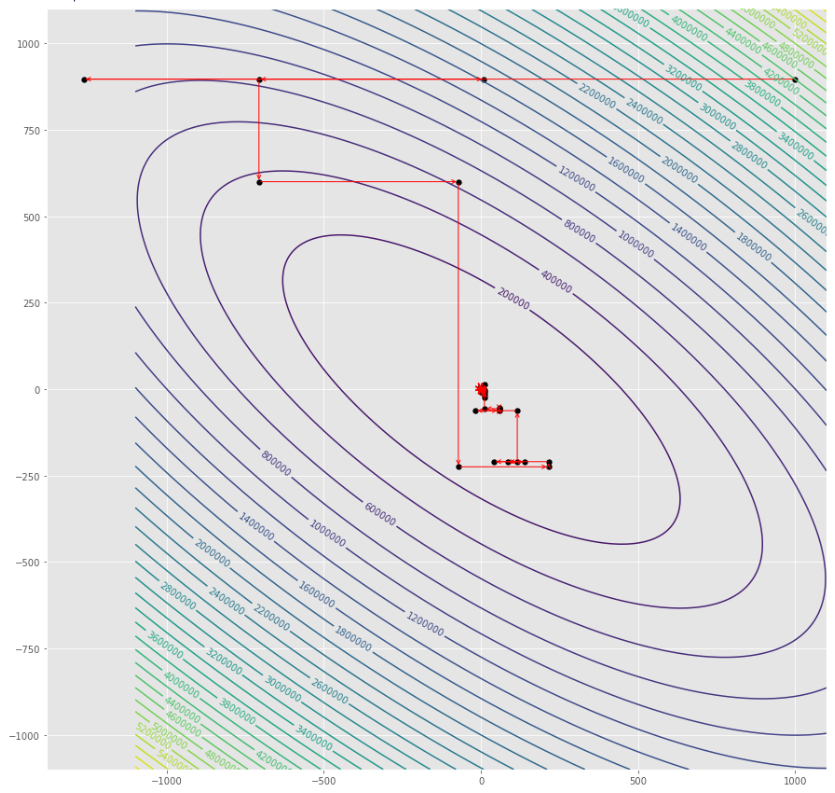
윤곽 줄거리 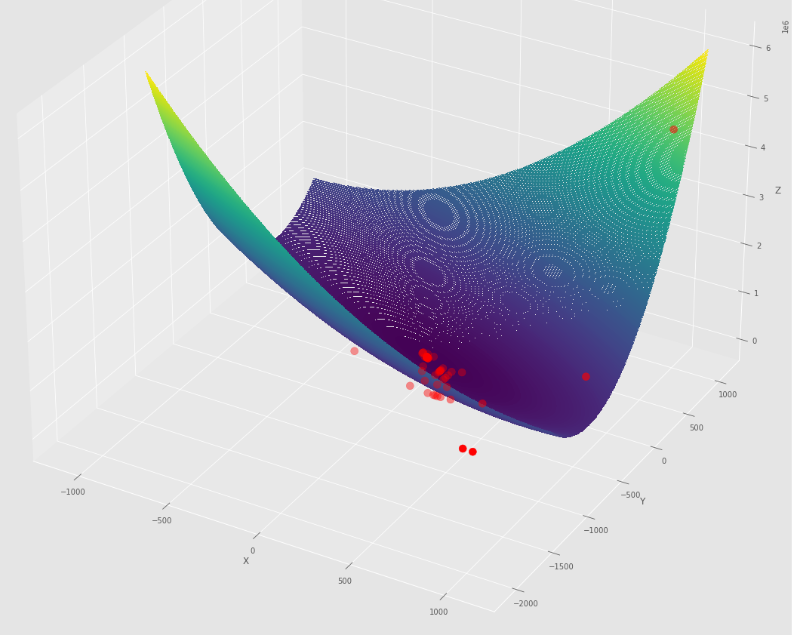
3D 플롯
pm3 . compare_all_time ( h , X , 1.e-2 , 1.e-1 , 100 , 2 ) 실행 시간 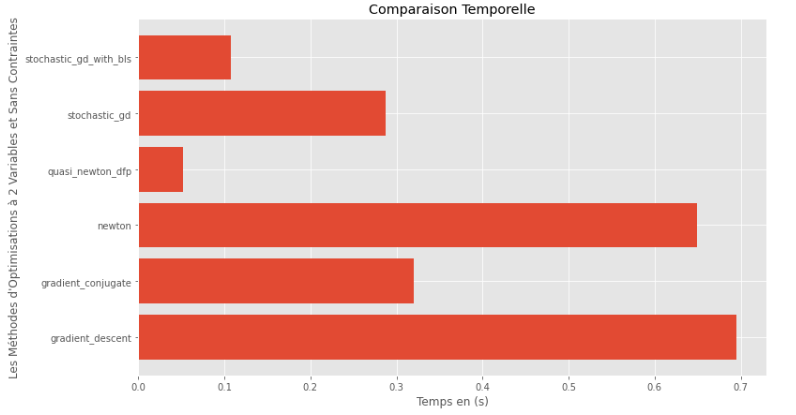
pm3 . compare_all_precision ( h , X , 1.e-2 , 1.e-1 , 100 , 2 ) True와 Computed Minims 사이의 간격 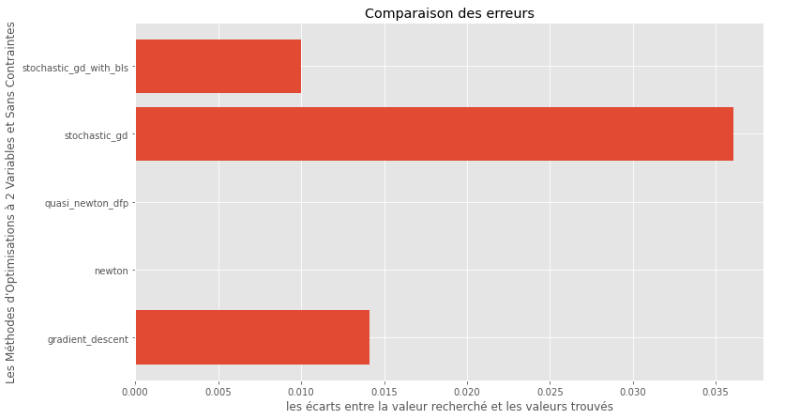
런타임 및 정확도 그래프 에서이 볼록한 다변량 함수에 대해 평가 된 알고리즘 중 Quasi Newton DFP 방법은 최적의 선택으로 나타나서 높은 정확도와 특히 런타임이 혼합되어 있음을 추론 할 수 있습니다.
import pyoptim . my_numpy . inverse as npi 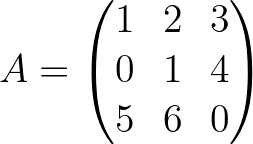
A = np . array ([[ 1. , 2. , 3. ],[ 0. , 1. , 4. ],[ 5. , 6. , 0. ]]) A_1 = npi . gaussjordan ( A . copy ())
I = A @ A_1
I = np . around ( I , 1 )
print ( 'A_1 = n n ' , A_1 )
print ( ' n A_1*A = n n ' , I ) A_1 =
[[-24. 18. 5.]
[ 20. -15. -4.]
[ -5. 4. 1.]]
A_1*A =
[[1. 0. 0.]
[0. 1. 0.]
[0. 0. 1.]] import pyoptim . my_numpy . decompose as npd A = np . array ([[ 1. , 2. , 3. ],[ 0. , 1. , 4. ],[ 5. , 6. , 0. ]]) #A is not positive definite
B = np . array ([[ 2. , - 1. , 0. ],[ - 1. , 2. , - 1. ],[ 0. , - 1. , 2. ]]) #B is positive definite
Y = np . array ([ 45 , - 78 , 95 ]) #randomly chosen column vector L , U , P = npd . LU ( A )
print ( "P = n " , P , " n n L = n " , L , " n n U = n " , U )
print ( " n " , A == P @ L @ U ) P =
[[0. 0. 1.]
[0. 1. 0.]
[1. 0. 0.]]
L =
[[1. 0. 0. ]
[0. 1. 0. ]
[0.2 0.8 1. ]]
U =
[[ 5. 6. 0. ]
[ 0. 1. 4. ]
[ 0. 0. -0.2]]
[[ True True True]
[ True True True]
[ True True True]] L = npd . choleski ( A ) # A is not positive definite
print ( L )
print ( "--------------------------------------------------" )
L = npd . choleski ( B ) # B is positive definite
print ( 'L = n ' , L , ' n ' )
C = np . around ( L @( L . T ), 1 )
print ( 'B = L@(L.T) n n ' , B == C ) A must be positive definite !
None
--------------------------------------------------
L =
[[ 1.41421356 0. 0. ]
[-0.70710678 1.22474487 0. ]
[ 0. -0.81649658 1.15470054]]
B = L@(L.T)
[[ True True True]
[ True True True]
[ True True True]] import pyoptim . my_numpy . solve as nps 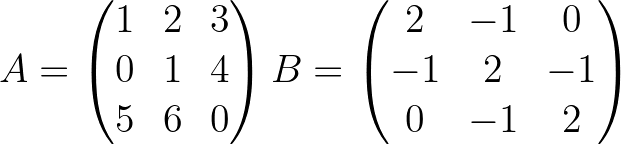
A = np . array ([[ 1. , 2. , 3. ], [ 0. , 1. , 4. ], [ 5. , 6. , 0. ]]) # A is not positive definite
B = np . array ([[ 2. , - 1. , 0. ], [ - 1. , 2. , - 1. ], [ 0. , - 1. , 2. ]]) # B is positive definite
Y = np . array ([ 45 , - 78 , 95 ]) # randomly chosen column vector X = nps . gaussjordan ( A , Y )
print ( "X = n " , X )
print ( " n A@X=Y n " , A @ X == Y , ' n ' )
print ( '---------------------------------------------------------------' )
X = nps . gaussjordan ( B , Y )
print ( "X = n " , X )
Y_ = np . around ( B @ X , 1 )
print ( " n B@X=Y n " , Y_ == Y , ' n ' ) X =
[[-2009.]
[ 1690.]
[ -442.]]
A@X=Y
[[ True]
[ True]
[ True]]
---------------------------------------------------------------
X =
[[18.5]
[-8. ]
[43.5]]
B@X=Y
[[ True]
[ True]
[ True]]
X = nps . LU ( A , Y )
print ( "X* = n " , X )
print ( " n AX*=Y n " , A @ X == Y )
print ( "-------------------------------------------------------------------------------" );
X = nps . LU ( B , Y )
print ( "X* = n " , X )
Y_ = np . around ( B @ X , 1 )
print ( " n BX*=Y n " , Y_ == Y ) X* =
[[-2009.]
[ 1690.]
[ -442.]]
AX*=Y
[[ True]
[ True]
[ True]]
-------------------------------------------------------------------------------
X* =
[[18.5]
[-8. ]
[43.5]]
BX*=Y
[[ True]
[ True]
[ True]] X = nps . choleski ( A , Y )
print ( "-------------------------------------------------------------------------------" )
X = nps . choleski ( B , Y )
print ( "X = n " , X )
Y_ = np . around ( B @ X , 1 )
print ( " n BX*=Y n " , Y_ == Y ) !! A must be positive definite !!
-------------------------------------------------------------------------------
X =
[[18.5]
[-8. ]
[43.5]]
BX*=Y
[[ True]
[ True]
[ True]]