Varbiable V Irus amp Licons (VARVAMP) é uma ferramenta para projetar iniciadores para vírus altamente diversos. A entrada é um alinhamento de suas sequências virais (total-genoma).
Para muitos gêneros de vírus, é difícil projetar iniciadores específicos de pan. O VarVamp resolve isso introduzindo caracteres ambíguos em iniciadores e minimiza as incompatibilidades na extremidade 3 '. Os primers podem não funcionar para algumas seqüências do seu alinhamento de entrada, mas devem reconhecer a grande maioria.
Varvamp vem em três sabores diferentes:

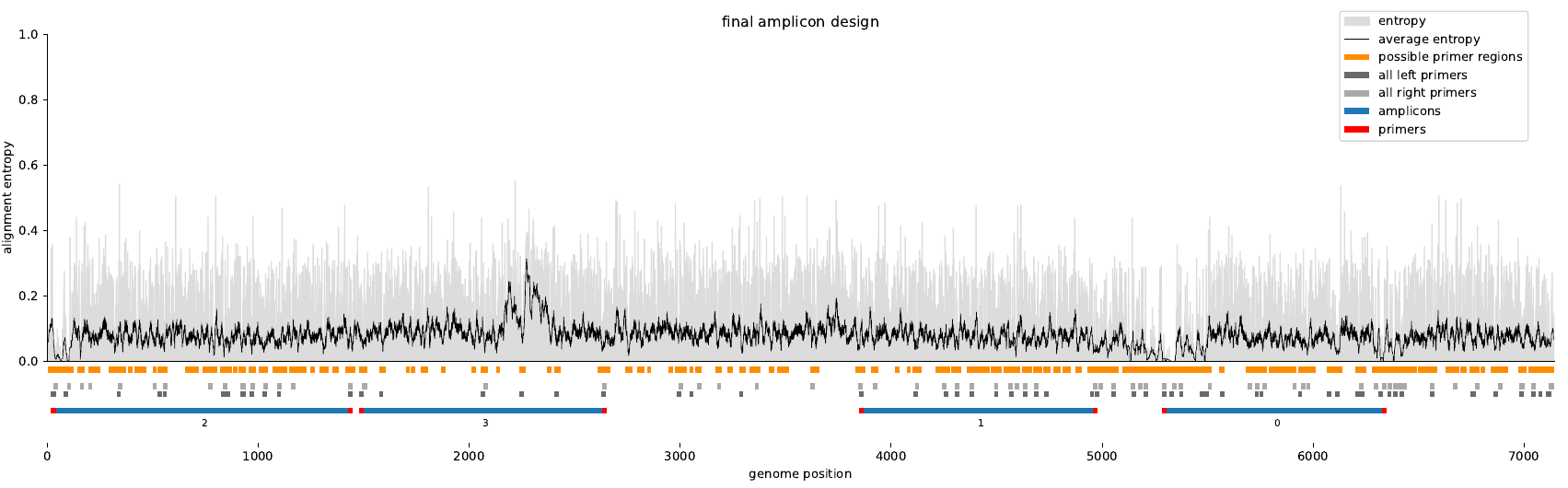
Single : Varvamp pesquisa os melhores primers e relatórios de backup de amplicons que não são sobrepostos que podem ser usados para abordagens de triagem baseadas em PCR.
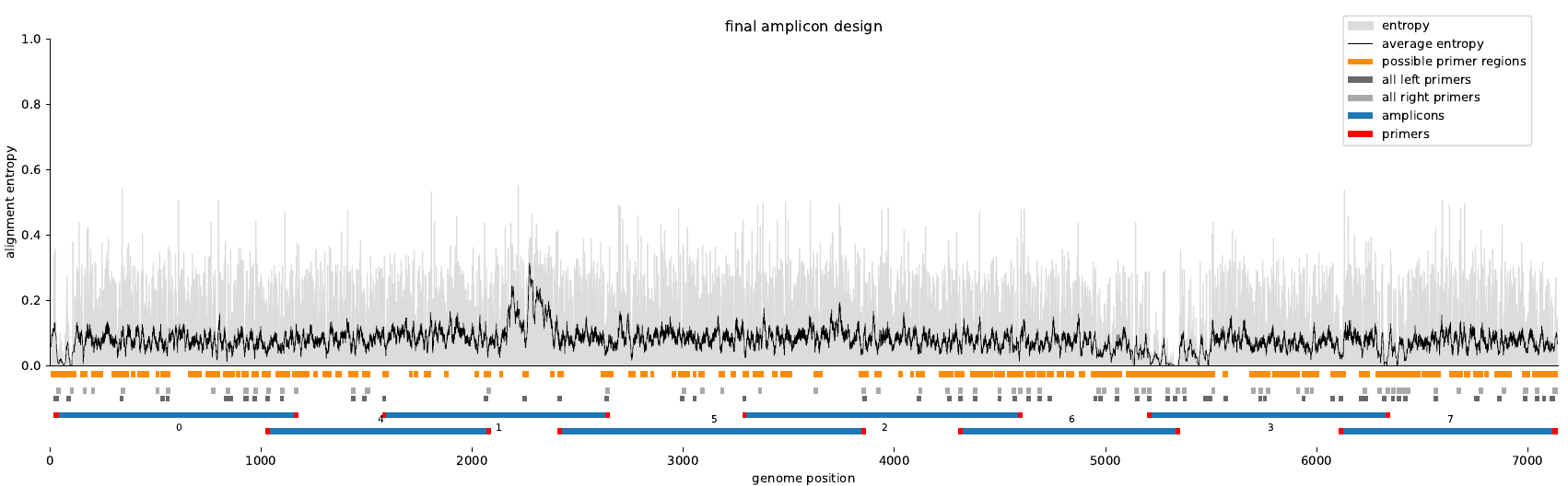
Ladrilho : O VARVAMP usa uma abordagem baseada em gráfico para projetar amplicons sobrepostos que ladrilhos todo o genoma viral. Isso projeta amplicons adequados para o seqüenciamento de nanoporos de nanoporos de Oxford ou Illumina.
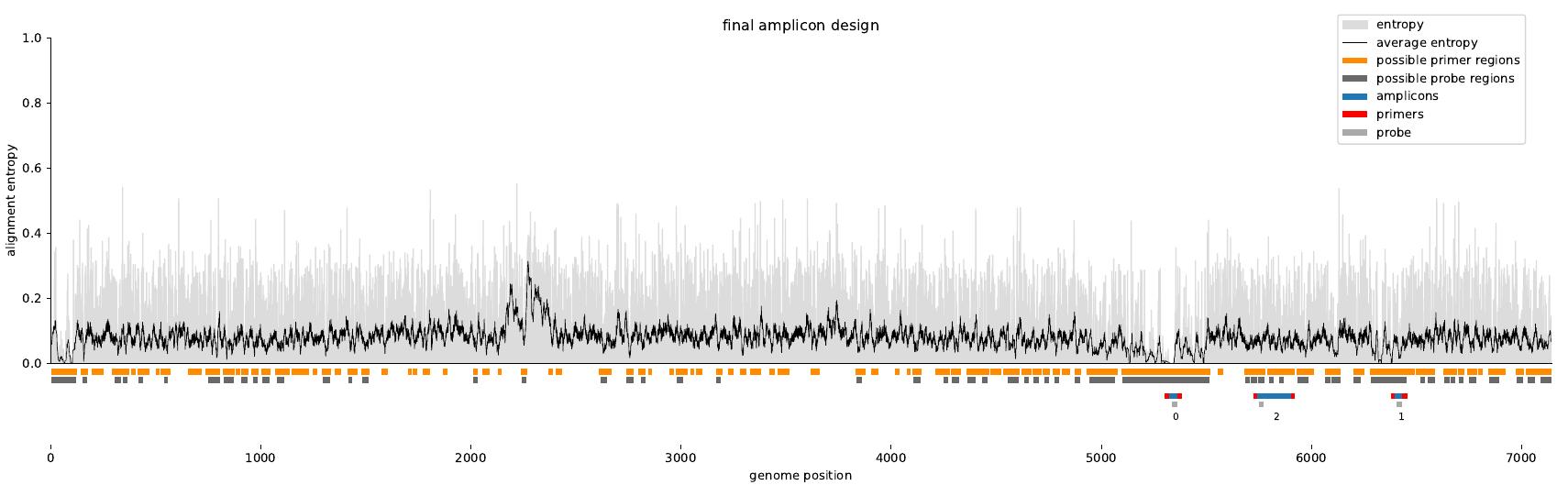
QPCR : Varvamp procura pequenos amplicons com uma sonda interna otimizada (TAQMAN). Minimiza as diferenças de temperatura entre os iniciadores e as verificações das estruturas secundárias do Amplicon.
Nós, em colaboração com especialistas para os respectivos vírus, já projetamos e avaliamos os esquemas de iniciadores de laboratório úmido para vários patógenos virais. Todos os dados de entrada e saídas VARVAMP estão disponíveis gratuitamente aqui.
Além disso, os iniciadores VarVamp estão agora disponíveis no Primerschemes. O VarVamp agora relata arquivos de cama iniciador no formato ARTICV3. Sinta -se à vontade para contribuir com esquemas recém -projetados por meio deste repositório do GitHub do Quicklab. Use a página primal desenvolvida por Chris Kent para gerar dados para solicitações de tração compatíveis.
VARVAMP: design automatizado de iniciadores específicos para pan para sequenciamento completo do genoma completo e qPCR de patógenos virais altamente diversos.
biorxiv pré -impressão
Isenção de responsabilidade importante: para o design do primer, o Varvamp usa o Primer3 para verificar se os KMers digeridos de uma sequência são iniciadores em potencial. Algumas das funções para isso foram adaptadas do PrimalsCheme e eu não reivindico crédito.
O código de remaking está sob a licença GPLV3. O código não tem garantia; sem a garantia implícita de comercialização ou aptidão para uma finalidade específica. Este programa é um software livre: você pode redistribuí -lo e/ou modificá -lo nos termos da licença pública geral da GNU, conforme publicado pela Free Software Foundation, versão 3 da licença ou (por sua opção) qualquer versão posterior.