Variable V Irus Ampons (VARVAMP) es una herramienta para diseñar cebadores para virus altamente diversos. La entrada es una alineación de sus secuencias virales (genoma completo).
Para muchos géneros de virus, es difícil diseñar cebadores específicos de PAN. Varvamp resuelve esto introduciendo caracteres ambiguos en cebadores y minimiza los desajustes en el extremo 3 '. Es posible que los cebadores no funcionen para algunas secuencias de su alineación de insumos, pero deben reconocer la gran mayoría.
Varvamp viene en tres sabores diferentes:

Single : Varvamp busca los mejores cebadores e informa los amplicones no superpuestos que se pueden utilizar para enfoques de detección basados en PCR.
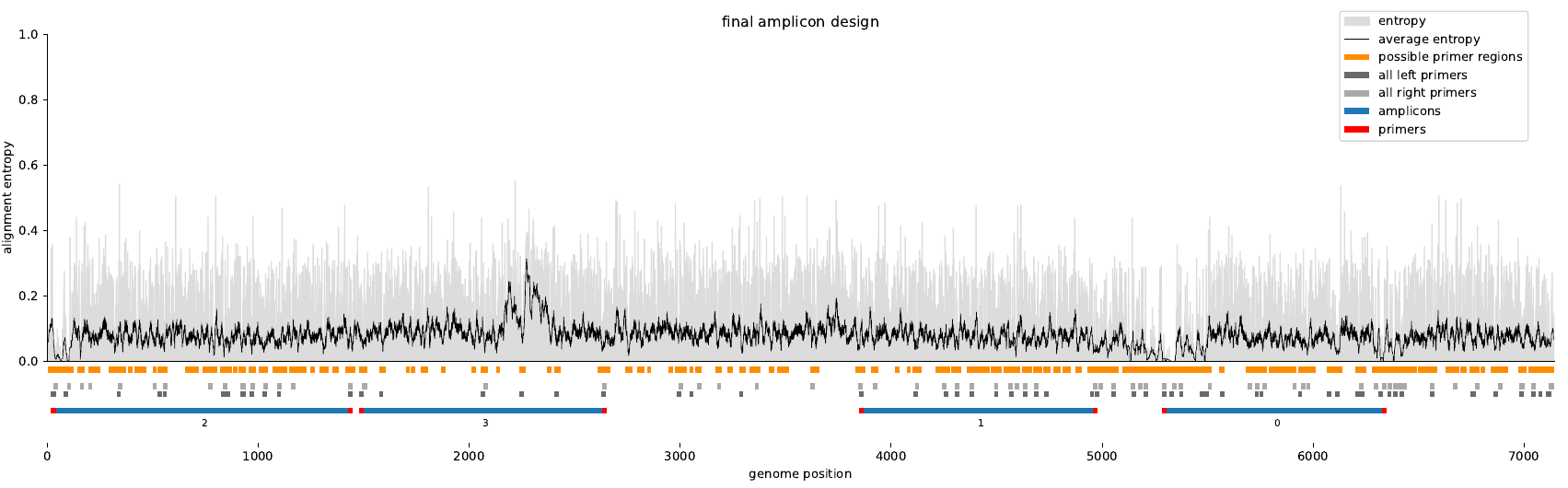
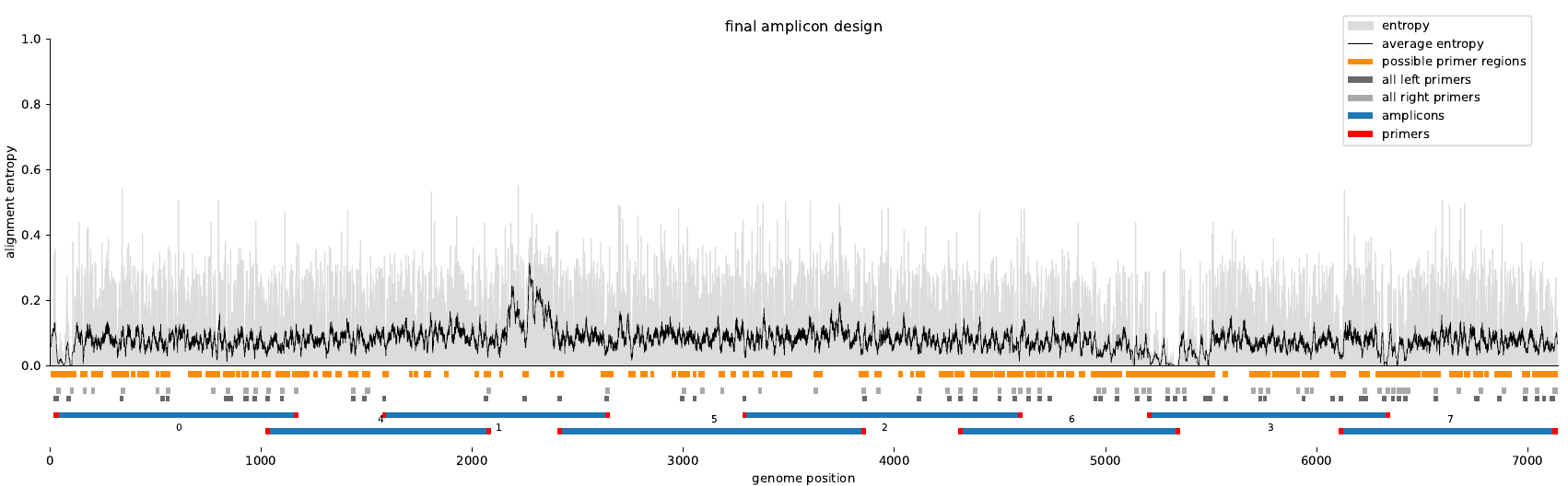
Azuleos : Varvamp utiliza un enfoque basado en gráficos para diseñar amplicones superpuestos que se muevan todo el genoma viral. Esto diseña amplicones que son adecuados para la secuenciación de genoma completo de Oxford Nanopore o Illumina.
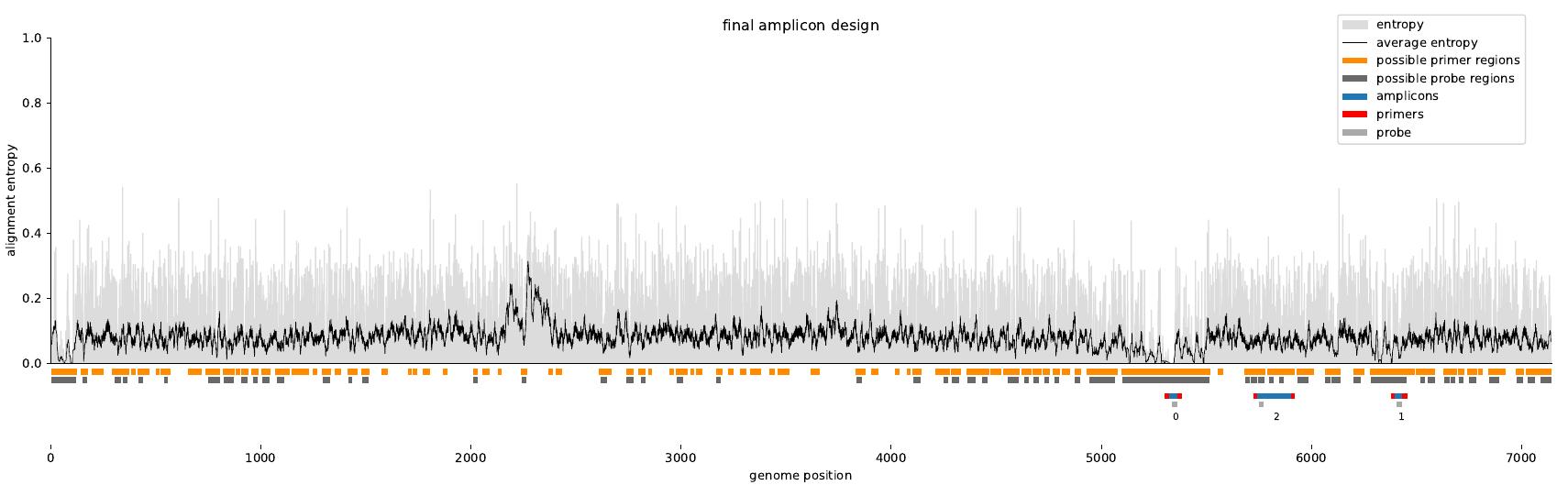
QPCR : Varvamp busca pequeños amplicones con una sonda interna optimizada (Taqman). Minimiza las diferencias de temperatura entre los cebadores y las verificaciones de estructuras secundarias de amplicón.
Nosotros, en colaboración con especialistas para los virus respectivos, ya hemos diseñado y húmedo los esquemas de cebadores evaluados para varios patógenos virales. Todos los datos de entrada y las salidas de Varvamp están disponibles gratuitamente aquí.
Además, los cebadores Varvamp ahora están disponibles en PrimerSchemes. Varvamp Now informa archivos de lecho de imprimación en formato Articv3. Siéntase libre de contribuir con esquemas de nuevo diseño a través de este repositorio de GitHub del QuickLab. Use PRIMAL-PAGE desarrollado por Chris Kent para generar datos para solicitudes de extracción compatibles.
Varvamp: diseño automatizado de cebador específico PAN para secuenciación del genoma completo de mosaico y QPCR de patógenos virales altamente diversos.
preimpresión de biorxiv
Descargo de responsabilidad importante: para el diseño del cebador, Varvamp usa Primer3 para verificar si los kmers digeridos de una secuencia son cebadores potenciales. Algunas de las funciones para esto fueron adaptadas de Primalscheme y no reclamo crédito.
El código Remaing está bajo la licencia GPLV3. El código no tiene ninguna garantía; Sin siquiera la garantía implícita de comerciabilidad o estado físico para un propósito particular. Este programa es un software gratuito: puede redistribuirlo y/o modificarlo bajo los términos de la Licencia Pública General de GNU publicada por Free Software Foundation, ya sea la versión 3 de la licencia o (a su opción) cualquier versión posterior.