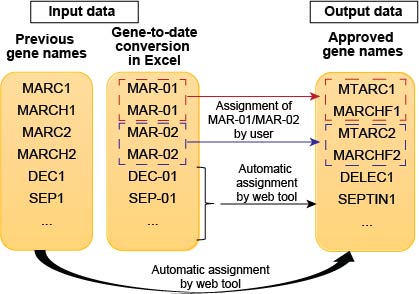
La conversion automatique des gènes en dates dans Excel peut être problématique, car les dates converties ne sont pas reconnues dans les bases de données de voie. Cet outil Web sert ainsi à convertir les anciens noms de gènes ou remonte aux noms de gènes mis à jour recommandés par le Hugo Gene Nomenclature Committee (HGNC). L'instance en cours d'exécution de l'application est déployée à: https://share.streamlit.io/kuanrongchan/date-to-gene-converter/main/date_gene_tool.py
Les utilisateurs peuvent télécharger leur fichier ou fichiers .csv ou .xlsx. Assurez-vous que la première colonne contient les noms de gènes. Avoir une description du gène sur la deuxième colonne est utile pour cartographier les gènes Mar-01 et MAR-02, bien que cette étape ne soit pas obligatoire. CheckBox est fourni aux utilisateurs pour inspecter leurs données. Si aucune donnée n'est téléchargée, un ensemble de données de démonstration composé d'une liste restreinte de gènes est préchargé. Les utilisateurs peuvent utiliser l'ensemble de données de démonstration préchargé pour explorer les fonctionnalités et les fonctionnalités de l'outil Web.
Si la première colonne contient les anciens noms de gènes, ces gènes seront mis à jour vers les nouveaux noms de gènes à l'aide du webtool. Si la première colonne contient des dates, elles seront converties en noms de gènes mis à jour, à l'exception de Mar-01 et MAR-02 car ces termes peuvent être mappés à plus d'un gène.
Lorsqu'il y a des valeurs en double Mar-01, MAR-01 sera annotée comme MAR-01_1ST et MAR-01_2ND. Les utilisateurs devront affecter manuellement les noms de gènes correspondants aux valeurs à l'aide des widgets fournis. Si la description du gène est fournie dans l'ensemble de données, les utilisateurs auront juste besoin de faire correspondre le nom du gène à la description du gène. Sinon, les utilisateurs devront vérifier leur ensemble de données bruts pour déterminer ce que signifient le MAR-01_1ST et MAR-01_2nd. Le même processus vaut également pour les valeurs à mars 02.
Enfin, les utilisateurs peuvent clés dans les gènes d'intérêt (par exemple MARCHF1) pour inspecter si les données d'expression des gènes ont en effet été mises à jour avec les nouveaux noms de gènes.
Veuillez exécuter les étapes suivantes pour exécuter localement l'outil Gene Updater:
Veuillez installer ce qui suit:
Pour installer Streamlit, exécutez le code suivant dans le terminal:
pip install streamlit
Installez les packages suivants localement en tapant les commandes suivantes dans le terminal:
pip install pandas
pip install numpy
pip install regex
pip install inflect
pip install dateparser
pip install openpyxl
pip install xlrd
pip install XlsxWriter
pip install streamlit-tags
Pour créer un nouveau dossier appelé streamlit_apps et en faire notre répertoire actuel, saisissez ce qui suit dans le terminal:
mkdir streamlit_apps
cd streamlit_apps
Exécutez les commandes suivantes dans le terminal:
mkdir date_gene_tool
cd date_gene_tool
touch date_gene_tool
Cela générera un fichier local nommé qui est nommé date_gene_tool.py. Après avoir localisé le fichier date_gene_tool.py, ouvrez le fichier dans l'éditeur de texte (par exemple, le code SUblime ou Visual Studio).
Lorsque vous ouvrez le fichier, copiez et collez les codes situés à: https://github.com/kuanrongchan/date-to-gene-converter/blob/main/date_gene_tool.py.
N'oubliez pas d'enregistrer le fichier après avoir tapé les codes.
Dans l'adresse GitHub (https://github.com/kuanrongchan/date-to-gene-converter), téléchargez les 2 fichiers suivants et placez les 2 fichiers dans le dossier date_gene_tool (avec le fichier date_gene_tool.py):
Vous pouvez exécuter le programme localement en tapant le terminal:
streamlit run date_gene_tool.py
Cela devrait ouvrir un nouvel onglet avec l'application Gene Updater apparaissant dans votre navigateur par défaut
En résumé, pour exécuter le fichier localement, veuillez télécharger ce référentiel, installer les exigences et exécuter le script avec les codes suivants dans la ligne de commande
cd path/to/folder
pip install -r requirements.txt
streamlit run date_gene_tool.py
Notez que les utilisateurs peuvent également télécharger directement tous les fichiers dans GitHub au format de fichier ZIP en appuyant sur le widget Dropdown "Code" pour exécuter le programme localement.
Vous pouvez également accéder aux fichiers directement depuis Zenodo
Cette application est développée par Clara Koh, Justin Ooi et Kuan Rong Chan de la Duke-Nus Medical School, Singapour. Pour plus de détails sur ce que nous faisons, veuillez visiter notre site Web à: kuanrongchan.com
Pour citer votre utilisation de Gene Updater, veuillez référencer cet article (https://www.nature.com/articles/s41598-022-17104-3):
Koh, CWT, Ooi, JSG, Joly, Glc et al. Gene Updater: un outil Web qui corrige automatiquement et met à jour pour les noms de gènes mal identifiés. Sci Rep 12, 12743 (2022). https://doi.org/10.1038/S41598-022-17104-3