Die automatische Umwandlung von Genen in Datteln in Excel kann problematisch sein, da die konvertierten Daten in den Pathway -Datenbanken nicht erkannt werden. Dieses Web -Tool dient somit dazu, die alten Gennamen oder Daten in die aktualisierten Gennamen zu konvertieren, wie vom Hugo -Gen -Nomenklaturkomitee (HGNC) empfohlen. Die laufende Instanz der App wird unter: https://share.streamlit.io/kuanrongchan/date-toge-converter/main/date_gene_tool.py bereitgestellt
Benutzer können ihre .csv- oder .xlsx -Dateien oder -Dateien hochladen. Stellen Sie sicher, dass die erste Spalte die Gennamen enthält. Eine Genbeschreibung in der zweiten Spalte ist nützlich für die Zuordnung der Gene von Mar-01 und Mar-02, obwohl dieser Schritt nicht obligatorisch ist. Das Kontrollkästchen wird für Benutzer bereitgestellt, um ihre Daten zu inspizieren. Wenn keine Daten hochgeladen werden, wird ein Demo-Datensatz, der aus einer eingeschränkten Liste von Genen besteht, vorbelastet. Benutzer können den vorbelasteten Demo-Datensatz verwenden, um die Funktionen und Funktionen des Web-Tools zu untersuchen.
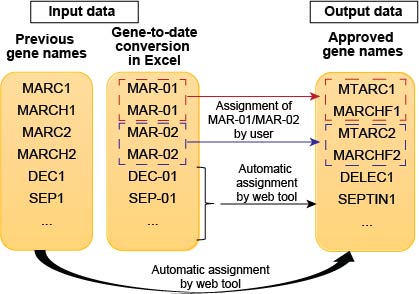
Wenn die erste Spalte die alten Gennamen enthält, werden diese Gene mithilfe der Webtool auf die neuen Gennamen aktualisiert. Wenn die erste Spalte Daten enthält, werden sie mit Ausnahme von Mar-01 und Mar-02 in die aktualisierten Gennamen konvertiert, da diese Begriffe auf mehr als ein Gen abgebildet werden können.
Wenn es doppelte MAR-01-Werte gibt, wird Mar-01 als Mar-01_1st und Mar-01_2nd annotiert. Benutzer müssen die entsprechenden Gennamen den Werten unter Verwendung der bereitgestellten Widgets manuell zuweisen. Wenn im Datensatz eine Genbeschreibung bereitgestellt wird, müssen Benutzer nur den Gennamen mit der Genbeschreibung übereinstimmen. Andernfalls müssen Benutzer ihren RAW-Datensatz überprüfen, um festzustellen, was der Mittelwert von Mar-01_1st und Mar-01_2nd bedeutet. Der gleiche Prozess gilt auch für die Werte von März-02.
Schließlich können Benutzer in den interessierenden Genen (z. B. Märzf1) untersuchen, ob die Genexpressionsdaten tatsächlich mit den neuen Gennamen aktualisiert wurden.
Bitte führen Sie die folgenden Schritte aus, um das Gen -Updater -Tool lokal auszuführen:
Bitte installieren Sie Folgendes:
Führen Sie zum Installieren von Stromlit den folgenden Code in Terminal aus:
pip install streamlit
Installieren Sie die folgenden Pakete lokal, indem Sie die folgenden Befehle in Terminal eingeben:
pip install pandas
pip install numpy
pip install regex
pip install inflect
pip install dateparser
pip install openpyxl
pip install xlrd
pip install XlsxWriter
pip install streamlit-tags
Um einen neuen Ordner namens streamlit_apps zu erstellen und es zu unserem aktuellen Verzeichnis zu machen, geben Sie Folgendes in das Terminal ein:
mkdir streamlit_apps
cd streamlit_apps
Führen Sie die folgenden Befehle im Terminal aus:
mkdir date_gene_tool
cd date_gene_tool
touch date_gene_tool
Dadurch wird eine lokale Datei generiert, die als DATE_GENE_TOOL.PY bezeichnet wird. Öffnen Sie die Datei in Textedato (z. B. Sublime oder Visual Studio Code), nachdem Sie die Datei date_gene_tool.py aufgefunden haben.
Wenn Sie die Datei öffnen, kopieren und fügen Sie die Codes ein, die sich unter https://github.com/kuanrongchan/date-togee-converter/blob/main/date_gene_tool.py befinden.
Denken Sie daran, die Datei nach dem Eingeben der Codes zu speichern.
In der Github-Adresse (https://github.com/kuanrongchan/date-togen-converter) laden Sie die folgenden 2 Dateien herunter und platzieren Sie die 2 Dateien in dem Ordner date_gene_tool (zusammen mit der Datei date_gene_tool.py):
Sie können das Programm lokal ausführen, indem Sie Terminal eingeben:
streamlit run date_gene_tool.py
Dies sollte eine neue Registerkarte mit der Gen -Updater -App öffnen, die in Ihrem Standardbrowser angezeigt wird
Zusammenfassend laden Sie dieses Repository, um die Datei lokal auszuführen, die Anforderungen herunter und führen
cd path/to/folder
pip install -r requirements.txt
streamlit run date_gene_tool.py
Beachten Sie, dass Benutzer auch alle Dateien in GitHub im ZIP -Dateiformat direkt herunterladen können, indem sie das Dropdown -Widget "Code" drücken, um das Programm lokal auszuführen.
Sie können auch auf die Dateien direkt von Zenodo zugreifen
Diese App wurde von Clara Koh, Justin Ooi und Kuan Rong Chan von der Duke-Nus Medical School in Singapur entwickelt. Weitere Informationen zu unserer Tätigkeit finden Sie auf unserer Website unter: kuanrongchan.com
Um Ihre Verwendung von Gene Updater zu zitieren, verweisen Sie bitte auf diesen Artikel (https://www.nature.com/articles/s41598-022-17104-3):
Koh, Cwt, Ooi, JSG, Joly, Glc et al. Gen -Updater: Ein Web -Tool, das automatisch und aktualisiert für Excel -fehl identifizierte Gennamen. SCI Rep 12, 12743 (2022). https://doi.org/10.1038/s41598-022-17104-3