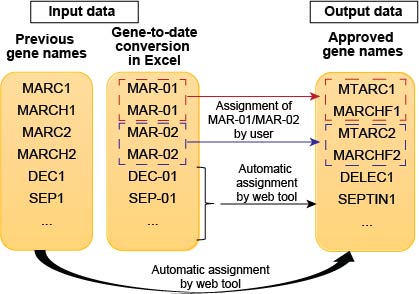
La conversión automática de genes a fechas en Excel puede ser problemática, ya que las fechas convertidas no se reconocen en las bases de datos de la vía. Por lo tanto, esta herramienta web sirve para convertir los viejos nombres de genes o las fechas en los nombres de genes actualizados según lo recomendado por el Comité de Nomenclatura de Gene Hugo (HGNC). La instancia en ejecución de la aplicación se implementa en: https://share.streamlit.io/kuanrongchan/date-to-gene-converter/main/date_gene_tool.py
Los usuarios pueden cargar su archivo o archivos .csv o .xlsx. Asegúrese de que la primera columna contenga los nombres de genes. Tener una descripción del gen en la segunda columna es útil para mapear los genes Mar-01 y Mar-02, aunque este paso no es obligatorio. La casilla de verificación se proporciona para que los usuarios inspeccionen sus datos. Si no se cargan datos, un conjunto de datos de demostración que consiste en una lista restringida de genes se realiza precargados. Los usuarios pueden usar el conjunto de datos de demostración precargado para explorar las características y funcionalidades de la herramienta web.
Si la primera columna contiene los antiguos nombres de genes, estos genes se actualizarán a los nuevos nombres de genes utilizando el WebTool. Si la primera columna contiene fechas, se convertirán en los nombres de genes actualizados, con la excepción de Mar-01 y Mar-02, ya que estos términos se pueden asignar a más de un gen.
Cuando hay valores duplicados de MAR-01, Mar-01 se anotará como Mar-01_1st y Mar-01_2nd. Los usuarios deberán asignar manualmente los nombres de genes correspondientes a los valores utilizando los widgets proporcionados. Si se proporciona una descripción del gen en el conjunto de datos, los usuarios solo deberán hacer coincidir el nombre del gen con la descripción del gen. De lo contrario, los usuarios tendrán que verificar su conjunto de datos sin procesar para determinar qué significan Mar-01_1ST y Mar-01_2nd. El mismo proceso también se aplica a los valores de MAR-02.
Finalmente, los usuarios pueden obtener los genes de interés (por ejemplo, MarchF1) para inspeccionar si los datos de expresión génica se han actualizado con los nuevos nombres de genes.
Ejecute los siguientes pasos para ejecutar la herramienta Gene Updater localmente:
Instale lo siguiente:
Para instalar Streamlit, ejecute el siguiente código en la terminal:
pip install streamlit
Instale los siguientes paquetes localmente escribiendo los siguientes comandos en la terminal:
pip install pandas
pip install numpy
pip install regex
pip install inflect
pip install dateparser
pip install openpyxl
pip install xlrd
pip install XlsxWriter
pip install streamlit-tags
Para crear una nueva carpeta llamada Streamlit_Apps y convertirlo en nuestro directorio actual, escriba lo siguiente en el terminal:
mkdir streamlit_apps
cd streamlit_apps
Ejecute los siguientes comandos en el terminal:
mkdir date_gene_tool
cd date_gene_tool
touch date_gene_tool
Esto generará un archivo local llamado que se denomina date_gene_tool.py. Después de localizar el archivo date_gene_tool.py, abra el archivo en el editor de texto (por ejemplo, sublime o el código de estudio de Visual).
Cuando abra el archivo, copie y pegue los códigos que se encuentran en: https://github.com/kuanrongchan/date-to-gene-converter/blob/main/date_gene_tool.py.
Recuerde guardar el archivo después de escribir los códigos.
En la dirección GitHub (https://github.com/kuanrongchan/date-to-gene-converter), descargue los siguientes 2 archivos y coloque los 2 archivos dentro de la carpeta date_gene_tool (junto con el archivo date_gene_tool.py):
Puede ejecutar el programa localmente escribiendo en terminal:
streamlit run date_gene_tool.py
Esto debería abrir una nueva pestaña con la aplicación Gene Updater que aparece en su navegador predeterminado
En resumen, para ejecutar el archivo localmente, descargue este repositorio, instale los requisitos y ejecute el script con los siguientes códigos en la línea de comando
cd path/to/folder
pip install -r requirements.txt
streamlit run date_gene_tool.py
Tenga en cuenta que los usuarios también pueden descargar directamente todos los archivos dentro de GitHub en el formato de archivo ZIP presionando el widget desplegable "Código" para ejecutar el programa localmente.
También puede acceder a los archivos directamente desde Zenodo
Esta aplicación está desarrollada por Clara Koh, Justin Ooi y Kuan Rong Chan de la Facultad de Medicina de Duke-Nus, Singapur. Para obtener más detalles sobre lo que hacemos, visite nuestro sitio web en: kuanrongchan.com
Para citar su uso de Gene Updater, consulte este artículo (https://www.nature.com/articles/S41598-022-17104-3):
Koh, CWT, OOI, JSG, Joly, Glc et al. Gene Updater: una herramienta web que se dirige y actualizas en autocorrección de Excel para nombres de genes erróneos. Sci Rep 12, 12743 (2022). https://doi.org/10.1038/s41598-022-17104-3