Rafael JosipPenić1 , TinVlašić2 ,Roland G. Huber 3 ,Yue Wan 2 , Milešikić2
1克羅地亞薩格勒布大學電氣工程與計算學院
2新加坡基因組研究所(GIS),科學,技術與研究機構(A*Star),新加坡
3新加坡科學,技術與研究機構(A*Star)生物信息學研究所(BII)
這是“ Rinalmo:通用RNA語言模型可以很好地概括在結構預測任務上”的官方實施。
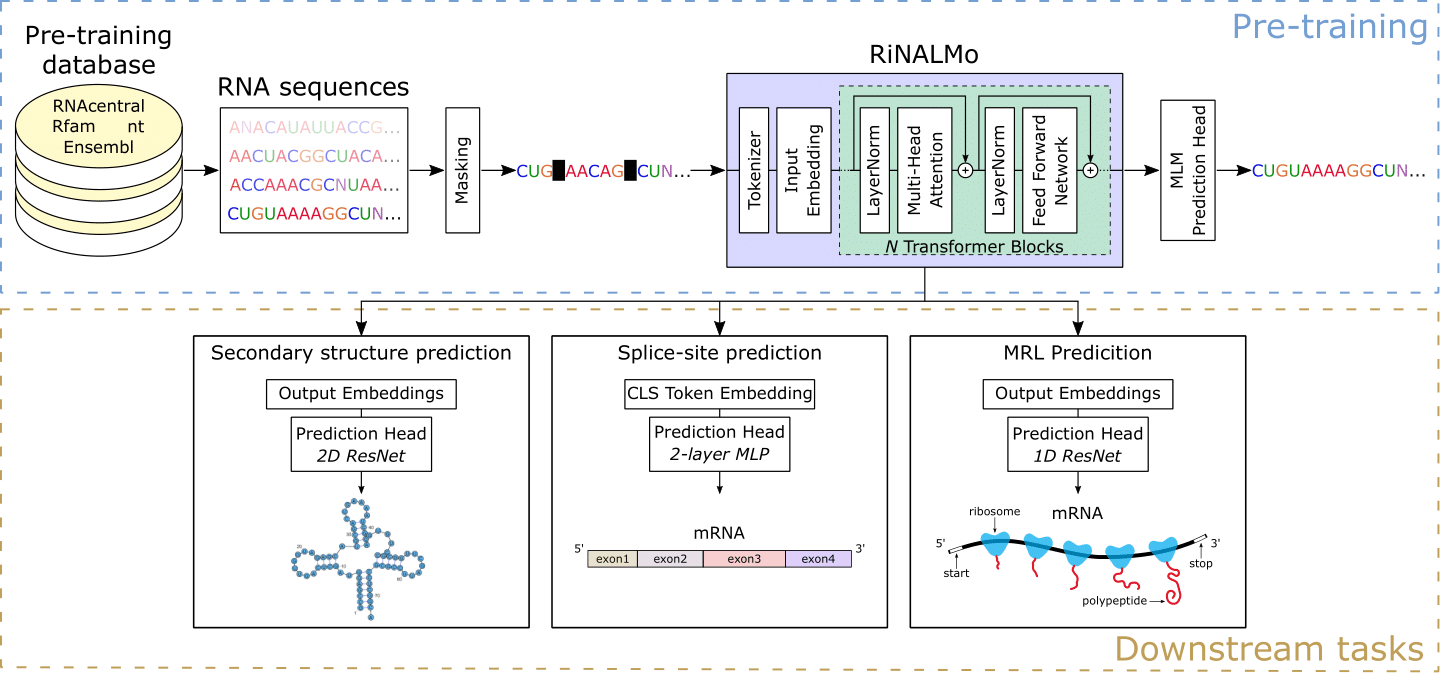
核糖核酸(RNA)在基本生物過程中起多種關鍵作用。最近,RNA已成為一個有趣的藥物靶標,強調需要提高我們對其結構和功能的理解。多年來,測序技術產生了大量未標記的RNA數據,這隱藏了重要的知識和潛力。在蛋白質語言模型的成功中,我們引入了核糖核酸語言模型(Rinalmo),以幫助揭示RNA的隱藏代碼。 Rinalmo是迄今為止最大的RNA語言模型,從幾個可用數據庫中的3600萬個非編碼RNA序列進行了6.5億個參數。 Rinalmo能夠提取隱藏的知識並捕獲隱式嵌入RNA序列中的基礎結構信息。 Rinalmo在幾個下游任務上實現了最新的結果。值得注意的是,我們表明它的概括能力可以克服其他深度學習方法無法概括看不見的RNA家族的其他深度學習方法。
使用以下命令進行安裝(先決條件: Python>=3.8和CUDA>=11.8 ):
git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMo
pip install .
pip install flash-attn==2.3.2安裝後,您可以輕鬆地使用Rinalmo獲得核苷酸表示:
import torch
from rinalmo . pretrained import get_pretrained_model
DEVICE = "cuda:0"
model , alphabet = get_pretrained_model ( model_name = "giga-v1" )
model = model . to ( device = DEVICE )
model . eval ()
seqs = [ "ACUUUGGCCA" , "CCCGGU" ]
tokens = torch . tensor ( alphabet . batch_tokenize ( seqs ), dtype = torch . int64 , device = DEVICE )
with torch . no_grad (), torch . cuda . amp . autocast ():
outputs = model ( tokens )
print ( outputs [ "representation" ])git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMoenvironment.yml中。 # create conda environment for RiNALMo
conda env create -f environment.yml
# activate RiNALMo environment
conda activate rinalmomkdir weights
cd weights
wget https://zenodo.org/records/10725749/files/rinalmo_giga_pretrained.pt # Download fine-tuned weights for secondary structure prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-16s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-23s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-5s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-srp_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-grp1_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-telomerase_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tmRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-RNaseP_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_bprna_ft.pt
# Download fine-tuned weights for splice-site prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_acceptor_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_donor_ft.pt
# Download fine-tuned weights for mean ribosome loading prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_mrl_ft.pt
cd ..您還可以在https://drive.google.com/drive/folders/1vgk3gy8c01o01o01o01o01o01o01o0wqfppmmyx5voy4wy8ebqs5下載小型Rinalmo型號(Rinalmo-148M(Mega)和Rinalmo-33,5M(Micro))的預訓練的重量。
我們為三個下游任務提供了預訓練的Rinalmo權重和微調權重:平均核醣體負載預測,二級結構預測和剪接位點預測。為了進行評估和微調,使用train_<downstream_task>.py腳本。
為了評估所提供的微調Rinalmo模型和預測頭,請使用以下輸入參數運行腳本:
# skip fine-tuning and run the evaluation on the test set
--test_only
# path to the '.pt' file containing fine-tuned model weights
--init_params
# dataset on which you would like to evaluate the fine-tuned model
--dataset
# download and prepare data (if needed)
--prepare_data
# Directory that will contain or already contains training, validation and test data
data_dir
# directory for all the output files
--output_dir要評估ArchiveII 5S RRNA測試數據集上的微調Rinalmo模型和預測頭進行輔助結構預測,請使用rinalmo_giga_ss_archiveII-5s_ft.pt 。在這裡,我們提供一個示例運行命令。
python train_sec_struct_prediction.py ./ss_data --test_only --init_params ./weights/rinalmo_giga_ss_archiveII-5s_ft.pt --dataset archiveII_5s --prepare_data --output_dir ./outputs/archiveII/5s/ --accelerator gpu --devices 1
為了微調rinalmo,請使用--pretrained_rinalmo_weights ./weights/rinalmo_giga_pretrained.pt輸入參數。使用--help來了解其他可用參數。對於剪接站點預測任務,可以在https://git.unistra.fr/nscalzitti/spliceator.git上獲得數據集和數據預處理代碼。
版權2024šikić實驗室-AI基因組學
根據Apache許可證(版本2.0(“許可”)獲得許可;除了符合許可外,您不得使用此文件。您可以在
http://www.apache.org/licenses/license-2.0
除非適用法律要求或以書面形式同意,否則根據許可證分配的軟件是按照“原樣”分發的,沒有任何明示或暗示的任何形式的保證或條件。請參閱許可證,以獲取執行許可條款和限制的特定語言。
Rinalmo參數可根據創意共享歸因4.0國際(CC By 4.0)許可證提供。您可以在以下網址找到詳細信息:https://creativecommons.org/licenses/4.0/legalcode。
如果您發現我們的工作對您的研究有用,請引用:
@article { penic2024_rinalmo ,
title = { RiNALMo: General-Purpose RNA Language Models Can Generalize Well on Structure Prediction Tasks } ,
author = { Penić, Rafael Josip and Vlašić, Tin and Huber, Roland G. and Wan, Yue and Šikić, Mile } ,
journal = { arXiv preprint arXiv:2403.00043 } ,
year = { 2024 }
}如果您有任何疑問,請隨時給作者發送電子郵件或打開問題。
這項工作得到了國家研究基金會(NRF)競爭研究計劃(CRP)的部分支持,該計劃識別登革熱病毒中的功能性RNA第三紀結構(NRF-CRP27-2021RS-0001),部分由A*Star下的Grant Gap2:A*Star下的GAP2:A**Star RNA-RNA模型(A*Star RNA模型(A*Star RNA-FM)(A*Star RNA-FM)(a a*star rna-fm) (i i223d1)。
該論文的計算工作部分是在新加坡國家超級計算中心的資源上進行的,https://www.nscc.sg。