Rafael Josip Penić 1 , Tin Vlašić 2 , Roland G. Huber 3 , Yue Wan 2 , Mile Šikić 2
1 คณะวิศวกรรมไฟฟ้าและคอมพิวเตอร์มหาวิทยาลัยซาเกร็บโครเอเชีย
2 Genome Institute of Singapore (GIS), หน่วยงานด้านวิทยาศาสตร์เทคโนโลยีและการวิจัย (A*Star), สิงคโปร์
3 Bioinformatics Institute (BII), หน่วยงานด้านวิทยาศาสตร์เทคโนโลยีและการวิจัย (A*Star), สิงคโปร์
นี่คือการดำเนินการอย่างเป็นทางการของกระดาษ "Rinalmo: แบบจำลองภาษา RNA ที่มีวัตถุประสงค์ทั่วไปสามารถพูดคุยได้ดีในงานการทำนายโครงสร้าง"
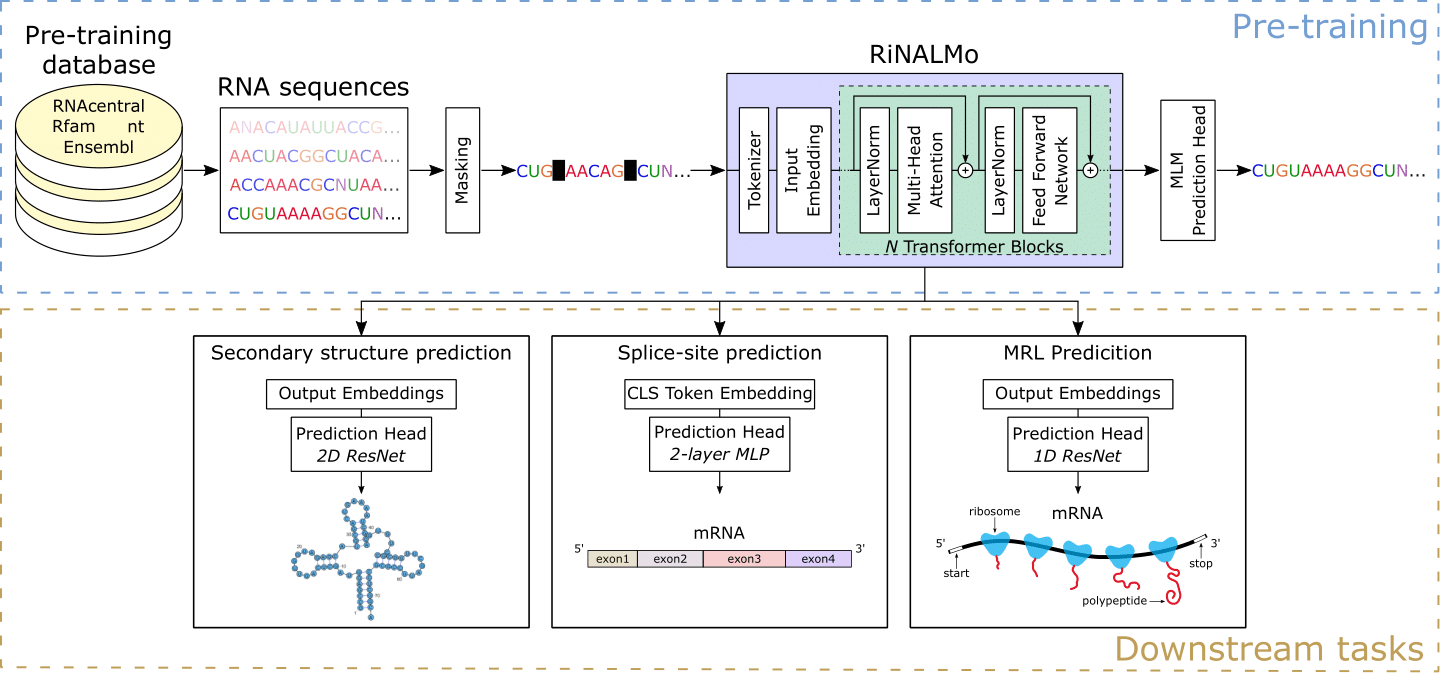
กรด Ribonucleic (RNA) มีบทบาทสำคัญในกระบวนการทางชีวภาพขั้นพื้นฐาน เมื่อเร็ว ๆ นี้ RNA ได้กลายเป็นเป้าหมายยาที่น่าสนใจโดยเน้นถึงความจำเป็นในการปรับปรุงความเข้าใจของเราเกี่ยวกับโครงสร้างและหน้าที่ของมัน ในช่วงหลายปีที่ผ่านมาเทคโนโลยีการเรียงลำดับได้สร้างข้อมูล RNA ที่ไม่มีป้ายกำกับจำนวนมหาศาลซึ่งซ่อนความรู้และศักยภาพที่สำคัญ แรงบันดาลใจจากความสำเร็จของแบบจำลองภาษาโปรตีนเราแนะนำโมเดลภาษากรด ribonucleic (Rinalmo) เพื่อช่วยเปิดเผยรหัสที่ซ่อนอยู่ของ RNA Rinalmo เป็นรูปแบบภาษา RNA ที่ใหญ่ที่สุดจนถึงปัจจุบันด้วยพารามิเตอร์ 650 ล้านพารามิเตอร์ที่ได้รับการฝึกอบรมล่วงหน้าเกี่ยวกับลำดับ RNA ที่ไม่มีการเข้ารหัส 36 ล้านครั้งจากฐานข้อมูลที่มีอยู่หลายแห่ง Rinalmo สามารถแยกความรู้ที่ซ่อนอยู่และจับข้อมูลโครงสร้างพื้นฐานที่ฝังอยู่ภายในลำดับ RNA โดยปริยาย Rinalmo บรรลุผลที่ล้ำสมัยในงานดาวน์สตรีมหลายงาน โดยเฉพาะอย่างยิ่งเราแสดงให้เห็นว่าความสามารถในการวางนัยทั่วไปของมันสามารถเอาชนะการไร้ความสามารถของวิธีการเรียนรู้ลึกอื่น ๆ สำหรับการทำนายโครงสร้างรองเพื่อพูดคุยเกี่ยวกับครอบครัว RNA ที่มองไม่เห็น
ใช้คำสั่งต่อไปนี้สำหรับการติดตั้ง (ข้อกำหนดเบื้องต้น: Python>=3.8 และ CUDA>=11.8 ):
git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMo
pip install .
pip install flash-attn==2.3.2หลังจากการติดตั้งคุณสามารถใช้ Rinalmo เพื่อรับการเป็นตัวแทนนิวคลีโอไทด์ได้อย่างง่ายดาย:
import torch
from rinalmo . pretrained import get_pretrained_model
DEVICE = "cuda:0"
model , alphabet = get_pretrained_model ( model_name = "giga-v1" )
model = model . to ( device = DEVICE )
model . eval ()
seqs = [ "ACUUUGGCCA" , "CCCGGU" ]
tokens = torch . tensor ( alphabet . batch_tokenize ( seqs ), dtype = torch . int64 , device = DEVICE )
with torch . no_grad (), torch . cuda . amp . autocast ():
outputs = model ( tokens )
print ( outputs [ "representation" ])git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMoenvironment.yml # create conda environment for RiNALMo
conda env create -f environment.yml
# activate RiNALMo environment
conda activate rinalmomkdir weights
cd weights
wget https://zenodo.org/records/10725749/files/rinalmo_giga_pretrained.pt # Download fine-tuned weights for secondary structure prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-16s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-23s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-5s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-srp_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-grp1_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-telomerase_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tmRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-RNaseP_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_bprna_ft.pt
# Download fine-tuned weights for splice-site prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_acceptor_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_donor_ft.pt
# Download fine-tuned weights for mean ribosome loading prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_mrl_ft.pt
cd ..นอกจากนี้คุณยังสามารถดาวน์โหลดน้ำหนักที่ได้รับการฝึกอบรมล่วงหน้าสำหรับรุ่น Rinalmo ขนาดเล็ก (Rinalmo-148m (mega) และ Rinalmo-33,5m (micro)) ที่ https://drive.google.com/drive/folders/1vgk3gy8c01o0wqfpmyx5
เราให้น้ำหนัก Rinalmo ที่ผ่านการฝึกอบรมมาก่อนและน้ำหนักที่ปรับแต่งสำหรับงานดาวน์สตรีมสามงาน: ค่าการทำนายการโหลดไรโบโซม, การทำนายโครงสร้างรองและการทำนายไซต์ประกบ สำหรับทั้งการประเมินผลและการปรับแต่งให้ใช้ train_<downstream_task>.py สคริปต์.
ในการประเมินโมเดล Rinalmo ที่ได้รับการปรับแต่งและหัวทำนายโปรดเรียกใช้สคริปต์โดยใช้อาร์กิวเมนต์อินพุตต่อไปนี้:
# skip fine-tuning and run the evaluation on the test set
--test_only
# path to the '.pt' file containing fine-tuned model weights
--init_params
# dataset on which you would like to evaluate the fine-tuned model
--dataset
# download and prepare data (if needed)
--prepare_data
# Directory that will contain or already contains training, validation and test data
data_dir
# directory for all the output files
--output_dir ในการประเมินโมเดล Rinalmo ที่ปรับแต่งอย่างละเอียดและหัวการทำนายบนชุดข้อมูลการทดสอบ ArchiveII 5S RRNA สำหรับการทำนายโครงสร้างรองให้ใช้ rinalmo_giga_ss_archiveII-5s_ft.pt น้ำหนัก ที่นี่เราให้คำสั่ง run ตัวอย่าง
python train_sec_struct_prediction.py ./ss_data --test_only --init_params ./weights/rinalmo_giga_ss_archiveII-5s_ft.pt --dataset archiveII_5s --prepare_data --output_dir ./outputs/archiveII/5s/ --accelerator gpu --devices 1
เพื่อปรับแต่ง Rinalmo ให้ใช้ --pretrained_rinalmo_weights ./weights/rinalmo_giga_pretrained.pt อาร์กิวเมนต์อินพุต ใช้ --help เพื่อเรียนรู้เกี่ยวกับอาร์กิวเมนต์อื่น ๆ ที่มีอยู่ สำหรับงานทำนาย Splice-Site ชุดข้อมูลและรหัสการประมวลผลข้อมูลล่วงหน้ามีอยู่ที่ https://git.unistra.fr/nscalzitti/spliceator.git
ลิขสิทธิ์ 2024 Šikić Lab - AI ใน Genomics
ได้รับใบอนุญาตภายใต้ใบอนุญาต Apache เวอร์ชัน 2.0 ("ใบอนุญาต"); คุณไม่สามารถใช้ไฟล์นี้ยกเว้นตามใบอนุญาต คุณอาจได้รับสำเนาใบอนุญาตที่
http://www.apache.org/licenses/license-2.0
เว้นแต่ว่ากฎหมายที่บังคับใช้หรือตกลงเป็นลายลักษณ์อักษรซอฟต์แวร์ที่แจกจ่ายภายใต้ใบอนุญาตจะถูกแจกจ่ายตาม "ตามพื้นฐาน" โดยไม่มีการรับประกันหรือเงื่อนไขใด ๆ ไม่ว่าจะโดยชัดแจ้งหรือโดยนัย ดูใบอนุญาตสำหรับภาษาเฉพาะที่ควบคุมการอนุญาตและข้อ จำกัด ภายใต้ใบอนุญาต
พารามิเตอร์ Rinalmo มีให้ภายใต้ข้อกำหนดของใบอนุญาต Creative Commons Attribution 4.0 International (CC By 4.0) คุณสามารถค้นหารายละเอียดได้ที่: https://creativeCommons.org/licenses/by/4.0/legalcode
หากคุณพบว่างานของเรามีประโยชน์ในการวิจัยของคุณโปรดอ้างอิง:
@article { penic2024_rinalmo ,
title = { RiNALMo: General-Purpose RNA Language Models Can Generalize Well on Structure Prediction Tasks } ,
author = { Penić, Rafael Josip and Vlašić, Tin and Huber, Roland G. and Wan, Yue and Šikić, Mile } ,
journal = { arXiv preprint arXiv:2403.00043 } ,
year = { 2024 }
}หากคุณมีคำถามใด ๆ โปรดส่งอีเมลถึงผู้เขียนหรือเปิดปัญหา
งานนี้ได้รับการสนับสนุนบางส่วนจากโครงการวิจัยการแข่งขันแห่งชาติ (NRF) (CRP) ภายใต้โครงการ ระบุโครงสร้าง RNA ตติยภูมิในการทำงานในไวรัสไข้เลือดออก (NRF-CRP27-2021RS-0001) และส่วนหนึ่งโดย A*Star ภายใต้ GAP2: A
งานคำนวณสำหรับกระดาษได้ดำเนินการบางส่วนบนทรัพยากรของศูนย์การคำนวณแห่งชาติ, สิงคโปร์ https://www.nscc.sg