Rafael JosipPenić1 、 TinVlašić2 、Roland G. Huber 3 、Yue Wan 2 、 Milešikić2
1クロアチア大学ザグレブ大学電気工学とコンピューティングの学部
2シンガポールのゲノム研究所(GIS)、科学技術研究機関(A*星)、シンガポール
3 Bioinformatics Institute(BII)、科学技術研究機関(A*星)、シンガポール
これは、論文「RinalMo:汎用RNA言語モデルが構造予測タスクでよく一般化できる」の公式実装です。
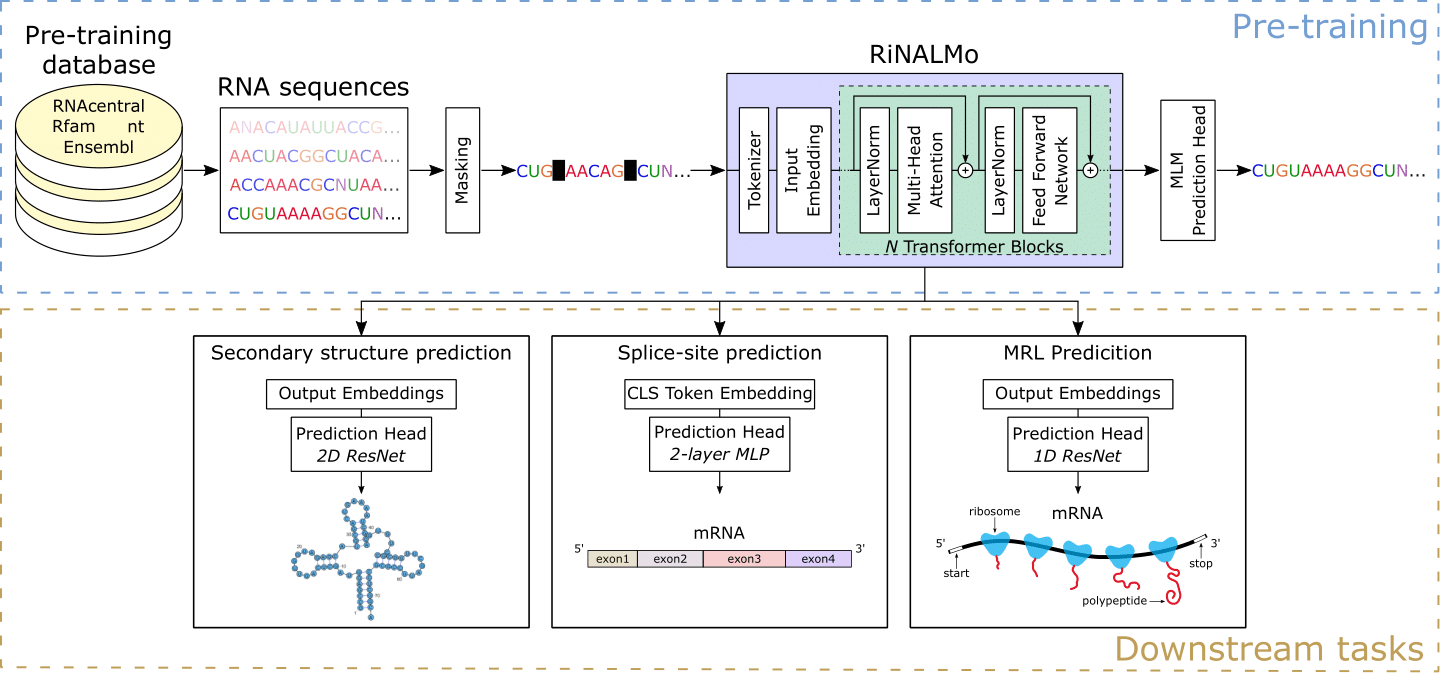
リボ核酸(RNA)は、基本的な生物学的プロセスにおいてさまざまな重要な役割を果たします。最近、RNAは興味深い薬物標的となっており、その構造と機能の理解を改善する必要性を強調しています。長年にわたり、シーケンシングテクノロジーは、重要な知識と潜在能力を隠す膨大な量の非標識RNAデータを生成してきました。タンパク質言語モデルの成功に動機付けられたリボ核酸言語モデル(RinalMO)を導入して、RNAの隠されたコードを発表するのに役立ちます。 RinalMOは、これまでで最大のRNA言語モデルであり、いくつかの利用可能なデータベースから3,600万個の非コーディングRNA配列で事前に訓練された6億5,000万個のパラメーターがあります。 RinalMoは、隠された知識を抽出し、RNA配列内に暗黙的に埋め込まれた基礎となる構造情報をキャプチャすることができます。 RinalMoは、いくつかのダウンストリームタスクで最先端の結果を達成しています。特に、その一般化能力が、目に見えないRNAファミリーに一般化するための二次構造予測のための他の深い学習方法の不能を克服できることを示しています。
インストールに次のコマンドを使用します(前提条件: Python>=3.8およびCUDA>=11.8 ):
git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMo
pip install .
pip install flash-attn==2.3.2設置後、RinalMOを使用してヌクレオチド表現を取得できます。
import torch
from rinalmo . pretrained import get_pretrained_model
DEVICE = "cuda:0"
model , alphabet = get_pretrained_model ( model_name = "giga-v1" )
model = model . to ( device = DEVICE )
model . eval ()
seqs = [ "ACUUUGGCCA" , "CCCGGU" ]
tokens = torch . tensor ( alphabet . batch_tokenize ( seqs ), dtype = torch . int64 , device = DEVICE )
with torch . no_grad (), torch . cuda . amp . autocast ():
outputs = model ( tokens )
print ( outputs [ "representation" ])git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMoenvironment.ymlに含める必要があります。 # create conda environment for RiNALMo
conda env create -f environment.yml
# activate RiNALMo environment
conda activate rinalmomkdir weights
cd weights
wget https://zenodo.org/records/10725749/files/rinalmo_giga_pretrained.pt # Download fine-tuned weights for secondary structure prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-16s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-23s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-5s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-srp_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-grp1_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-telomerase_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tmRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-RNaseP_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_bprna_ft.pt
# Download fine-tuned weights for splice-site prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_acceptor_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_donor_ft.pt
# Download fine-tuned weights for mean ribosome loading prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_mrl_ft.pt
cd ..また、https://drive.google.com/drive/folders/1vgk3gy8c01o0wqfppmyx5voy4wy8ebqs5に、小規模なリナルモモデル(Rinalmo-148m(Mega)およびRinalmo-33,5M(Micro))の事前に訓練された重量をダウンロードすることもできます。
3つのダウンストリームタスクの事前に訓練されたRinalMOウェイトと微調整された重みを提供します:平均リボソーム負荷予測、二次構造予測、およびスプライスサイト予測。評価と微調整の両方に使用してtrain_<downstream_task>.pyスクリプトを使用します。
提供された微調整されたRinalMOモデルと予測ヘッドを評価するために、次の入力引数を使用してスクリプトを実行してください。
# skip fine-tuning and run the evaluation on the test set
--test_only
# path to the '.pt' file containing fine-tuned model weights
--init_params
# dataset on which you would like to evaluate the fine-tuned model
--dataset
# download and prepare data (if needed)
--prepare_data
# Directory that will contain or already contains training, validation and test data
data_dir
# directory for all the output files
--output_dir微調整されたRinalMOモデルと予測ヘッドのArchiveiii 5s RRNAテストデータセットのセカンダリ構造予測の評価を評価するには、 rinalmo_giga_ss_archiveII-5s_ft.ptウェイトを使用します。ここでは、runコマンドの例を提供します。
python train_sec_struct_prediction.py ./ss_data --test_only --init_params ./weights/rinalmo_giga_ss_archiveII-5s_ft.pt --dataset archiveII_5s --prepare_data --output_dir ./outputs/archiveII/5s/ --accelerator gpu --devices 1
rinalmoを微調整するには、 --pretrained_rinalmo_weights ./weights/rinalmo_giga_pretrained.pt入力引数を使用してください。 --helpを使用して、他の利用可能な議論について学びます。スプライスサイト予測タスクの場合、データセットとデータ前処理コードはhttps://git.unistra.fr/nscalzitti/spliceator.gitで入手できます。
Copyright 2024šikićLab -AI in Genomics
Apacheライセンス、バージョン2.0(「ライセンス」)に基づいてライセンスされています。ライセンスに準拠している場合を除き、このファイルを使用することはできません。ライセンスのコピーを取得できます
http://www.apache.org/licenses/license-2.0
適用法で要求されていないか、書面で合意されていない限り、ライセンスに基づいて配布されたソフトウェアは、明示または黙示のいずれかの保証または条件なしに、「現状のまま」に基づいて配布されます。ライセンスに基づく権限と制限を管理する特定の言語のライセンスを参照してください。
RinalMOパラメーターは、Creative Commons Attribution 4.0 International(CC by 4.0)ライセンスの条件の下で利用可能になります。詳細は、https://creativecommons.org/licenses/by/4.0/legalcodeで見つけることができます。
私たちの研究があなたの研究で役立つと思うなら、引用してください:
@article { penic2024_rinalmo ,
title = { RiNALMo: General-Purpose RNA Language Models Can Generalize Well on Structure Prediction Tasks } ,
author = { Penić, Rafael Josip and Vlašić, Tin and Huber, Roland G. and Wan, Yue and Šikić, Mile } ,
journal = { arXiv preprint arXiv:2403.00043 } ,
year = { 2024 }
}ご質問がある場合は、著者に電子メールを送信するか、問題を開いてください。
この研究は、デング熱ウイルス(NRF-CRP27-2021RS-0001)の機能的RNA三次構造を特定するプロジェクトの下で、国立研究財団(NRF)競争研究プログラム( CRP )によって部分的にサポートされていました。
この論文の計算作業は、シンガポールhttps://www.nscc.sgの国立スーパーコンピューティングセンターのリソースで部分的に実行されました。