Rafael Josip Penić 1 ، Tin Vlašić 2 ، Roland G. Huber 3 ، Yue Wan 2 ، Mile šikić 2
1 كلية الهندسة الكهربائية والحوسبة ، جامعة زغرب ، كرواتيا
2 معهد الجينوم في سنغافورة (GIS) ، وكالة العلوم والتكنولوجيا والبحث (A*Star) ، سنغافورة
3 معهد المعلوماتية الحيوية (BII) ، وكالة العلوم والتكنولوجيا والبحث (A*Star) ، سنغافورة
هذا هو التنفيذ الرسمي للورقة "RinalMo: يمكن أن تعمم نماذج لغة الحمض النووي الريبي للأغراض العامة بشكل جيد على مهام التنبؤ بالهيكل".
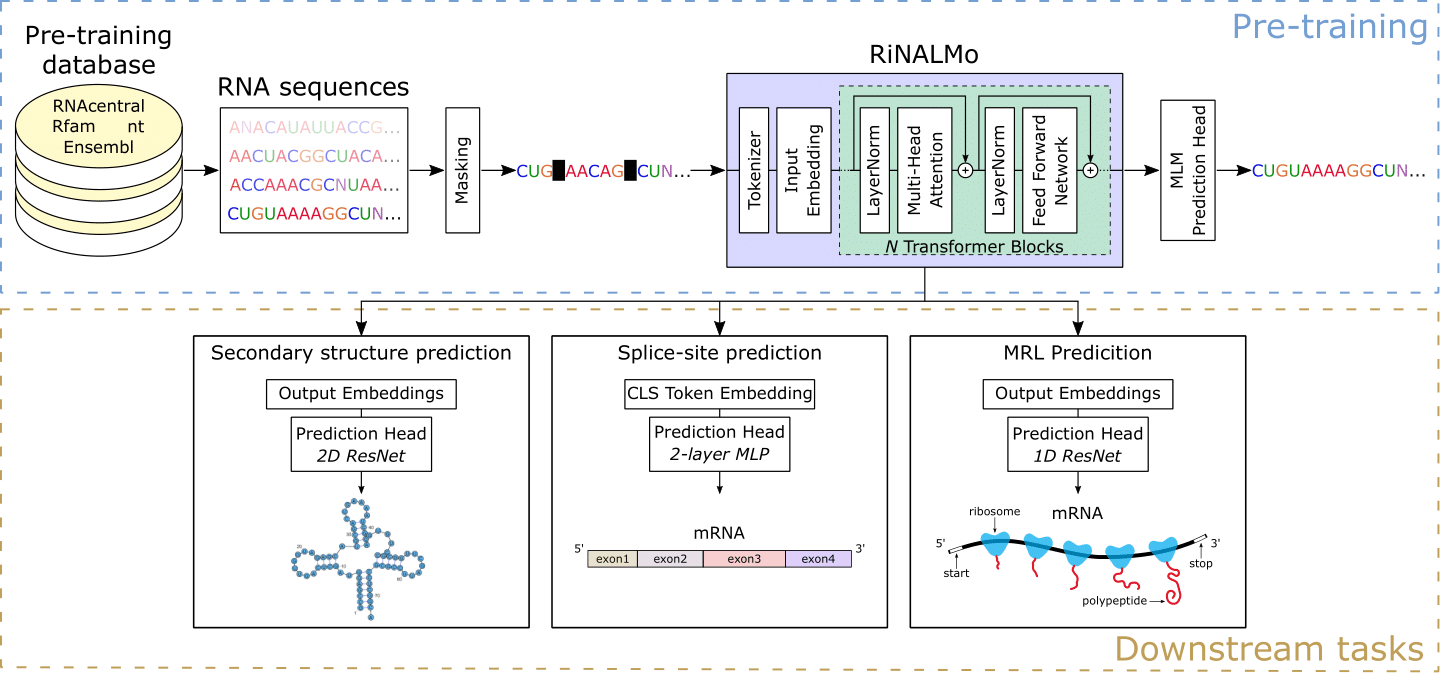
يلعب حمض الريبونوكليك (RNA) مجموعة متنوعة من الأدوار الحاسمة في العمليات البيولوجية الأساسية. في الآونة الأخيرة ، أصبح الحمض النووي الريبي هدفًا للاهتمام ، مما يؤكد على الحاجة إلى تحسين فهمنا لهياكلها ووظائفها. على مر السنين ، أنتجت تقنيات التسلسل قدرًا هائلاً من بيانات الحمض النووي الريبي غير المسبق ، والتي تخفي المعرفة والإمكانات المهمة. بدافع من نجاحات نماذج لغة البروتين ، نقدم نموذج لغة حمض الريبون النووي (RinalMO) للمساعدة في كشف النقاب عن رمز RNA الخفي. Rinalmo هو أكبر نموذج لغة الحمض النووي الريبي حتى الآن مع 650 مليون معلمة تم تدريبها مسبقًا على 36 مليون تسلسل الحمض النووي الريبي غير المشفر من عدة قواعد بيانات متاحة. Rinalmo قادر على استخراج المعرفة المخفية والتقاط معلومات الهيكل الأساسي المضمّن ضمنيًا في تسلسل الحمض النووي الريبي. يحقق Rinalmo نتائج أحدث على أحدث مهام في اتجاه مجرى النهر. والجدير بالذكر أن قدرات التعميم الخاصة بها يمكن أن تتغلب على عجز أساليب التعلم العميق الأخرى للتنبؤ بالهيكل الثانوي للتعميم على عائلات الحمض النووي الريبي غير المرئي.
استخدم الأوامر التالية للتثبيت (المتطلبات الأساسية: Python>=3.8 و CUDA>=11.8 ):
git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMo
pip install .
pip install flash-attn==2.3.2بعد التثبيت ، يمكنك بسهولة استخدام RinalMo للحصول على تمثيلات النوكليوتيدات:
import torch
from rinalmo . pretrained import get_pretrained_model
DEVICE = "cuda:0"
model , alphabet = get_pretrained_model ( model_name = "giga-v1" )
model = model . to ( device = DEVICE )
model . eval ()
seqs = [ "ACUUUGGCCA" , "CCCGGU" ]
tokens = torch . tensor ( alphabet . batch_tokenize ( seqs ), dtype = torch . int64 , device = DEVICE )
with torch . no_grad (), torch . cuda . amp . autocast ():
outputs = model ( tokens )
print ( outputs [ "representation" ])git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMoenvironment.yml . # create conda environment for RiNALMo
conda env create -f environment.yml
# activate RiNALMo environment
conda activate rinalmomkdir weights
cd weights
wget https://zenodo.org/records/10725749/files/rinalmo_giga_pretrained.pt # Download fine-tuned weights for secondary structure prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-16s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-23s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-5s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-srp_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-grp1_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-telomerase_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tmRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-RNaseP_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_bprna_ft.pt
# Download fine-tuned weights for splice-site prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_acceptor_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_donor_ft.pt
# Download fine-tuned weights for mean ribosome loading prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_mrl_ft.pt
cd ..يمكنك أيضًا تنزيل الأوزان التي تم تدريبها مسبقًا لنماذج Rinalmo الأصغر (Rinalmo-148m (MEGA) و Rinalmo-33،5m (micro)) على https://drive.google.com/drive/folders/1vgk3gy8c01o0wqfmyx5voy4wy8ebqs5.
نحن نقدم أوزان Rinalmo التي تم تدريبها مسبقًا والأوزان التي يتم ضبطها لثلاث مهام في اتجاه مجرى النهر: متوسط التنبؤ بتحميل الريبوسوم ، والتنبؤ بالهيكل الثانوي والتنبؤ بموقع الوصية. لكل من التقييم والضوء الاستخدام train_<downstream_task>.py .
من أجل تقييم نماذج Rinalmo التي تم ضبطها المقدمة ورؤوس التنبؤ ، يرجى تشغيل البرامج النصية باستخدام وسيطات الإدخال التالية:
# skip fine-tuning and run the evaluation on the test set
--test_only
# path to the '.pt' file containing fine-tuned model weights
--init_params
# dataset on which you would like to evaluate the fine-tuned model
--dataset
# download and prepare data (if needed)
--prepare_data
# Directory that will contain or already contains training, validation and test data
data_dir
# directory for all the output files
--output_dir لتقييم نموذج Rinalmo الذي تم ضبطه بشكل جيد ورأس التنبؤ على مجموعة بيانات اختبار Archiveii 5S RRNA للتنبؤ بالهيكل الثانوي ، استخدم أوزان rinalmo_giga_ss_archiveII-5s_ft.pt . هنا ، نحن نقدم أمر تشغيل مثال.
python train_sec_struct_prediction.py ./ss_data --test_only --init_params ./weights/rinalmo_giga_ss_archiveII-5s_ft.pt --dataset archiveII_5s --prepare_data --output_dir ./outputs/archiveII/5s/ --accelerator gpu --devices 1
من أجل ضبط rinalmo ، استخدم- --pretrained_rinalmo_weights ./weights/rinalmo_giga_pretrained.pt . استخدم --help للتعرف على الحجج الأخرى المتاحة. بالنسبة لمهمة التنبؤ بموقع لصق ، تتوفر رمز المعالجة المسبقة لمجموعة البيانات ورمز البيانات على https://git.unistra.fr/nscalzitti/Spliceator.git.
حقوق الطبع والنشر 2024 šikić Lab - AI في الجينوم
مرخصة بموجب ترخيص Apache ، الإصدار 2.0 ("الترخيص") ؛ لا يجوز لك استخدام هذا الملف إلا في الامتثال للترخيص. يمكنك الحصول على نسخة من الترخيص على
http://www.apache.org/licenses/license-2.0
ما لم يكن مطلوبًا بموجب القانون المعمول به أو الموافقة على الكتابة ، يتم توزيع البرامج الموزعة بموجب الترخيص على أساس "كما هي" ، دون ضمانات أو شروط من أي نوع ، إما صريحة أو ضمنية. راجع ترخيص الأذونات والقيود التي تحكم اللغة المحددة بموجب الترخيص.
يتم توفير معلمات RinalMO بموجب شروط ترخيص Creative Commons Attribution 4.0 International (CC BY 4.0). يمكنك العثور على التفاصيل على: https://creativecommons.org/licenses/by/4.0/legalcode.
إذا وجدت عملنا مفيدًا في بحثك ، فيرجى الاستشهاد:
@article { penic2024_rinalmo ,
title = { RiNALMo: General-Purpose RNA Language Models Can Generalize Well on Structure Prediction Tasks } ,
author = { Penić, Rafael Josip and Vlašić, Tin and Huber, Roland G. and Wan, Yue and Šikić, Mile } ,
journal = { arXiv preprint arXiv:2403.00043 } ,
year = { 2024 }
}إذا كان لديك أي أسئلة ، فلا تتردد في إرسال بريد إلكتروني إلى المؤلفين أو فتح مشكلة.
تم دعم هذا العمل جزئيًا من قبل برنامج البحوث التنافسية للبحوث الوطنية (NRF) (CRP) تحت تحديد هياكل RNA الوظيفية في فيروس حمى الضنك (NRF-CRP27-2021RS-0001) وفي جزء من النجمة A*under Grant GAP2: A*Star RNA-Foundation (A*Star RNA-FM) (I23D1AG079).
تم تنفيذ العمل الحسابي للورقة جزئيًا على موارد المركز الوطني للحوسبة الفائقة ، سنغافورة https://www.nscc.sg.